MODULO 3 – D
LA SINTESI PROTEICA
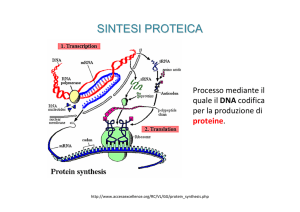
La TRADUZIONE o SINTESI PROTEICA o PROTEOSINTESI
costituisce la seconda fase del processo di espressione genica, ovvero il
processo in cui l’informazione contenuta nel DNA dei geni viene convertita
in proteine che svolgono nella cellula numerose funzioni.
La sintesi proteica prevede 3 fasi:
· INIZIO: processo che vede i ribosomi legarsi al codone di avvio AUG
dell’ mRNA, ovvero il punto in cui l’ mRNA comincia a codificare la
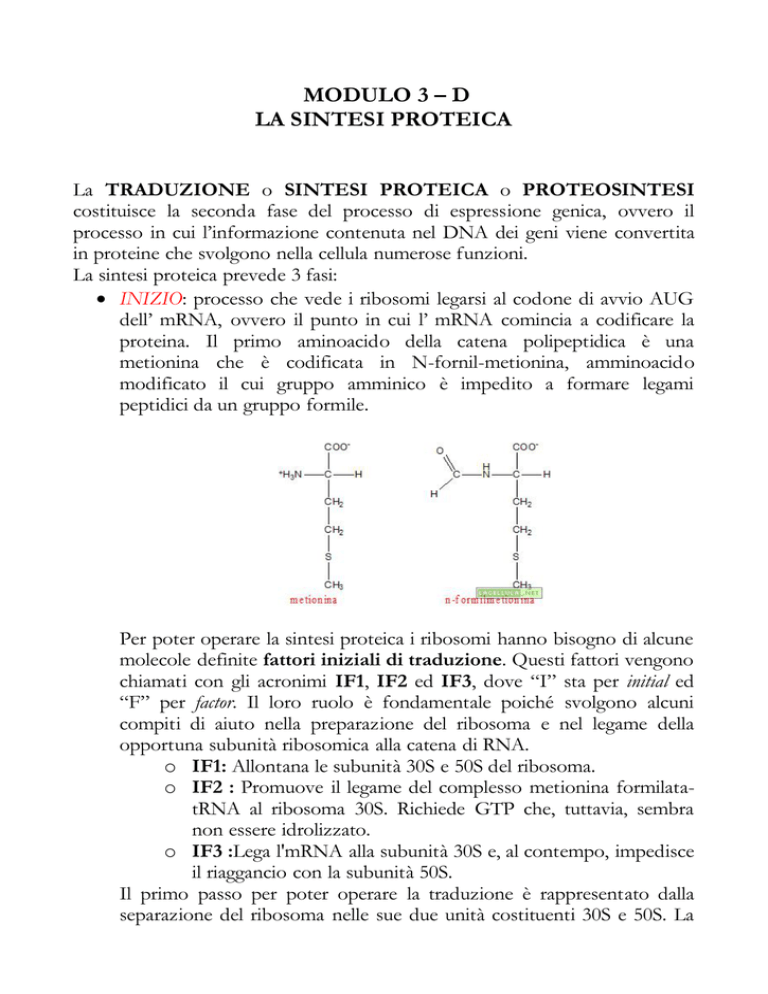
proteina. Il primo aminoacido della catena polipeptidica è una
metionina che è codificata in N-fornil-metionina, amminoacido
modificato il cui gruppo amminico è impedito a formare legami
peptidici da un gruppo formile.
Per poter operare la sintesi proteica i ribosomi hanno bisogno di alcune
molecole definite fattori iniziali di traduzione. Questi fattori vengono
chiamati con gli acronimi IF1, IF2 ed IF3, dove “I” sta per initial ed
“F” per factor. Il loro ruolo è fondamentale poiché svolgono alcuni
compiti di aiuto nella preparazione del ribosoma e nel legame della
opportuna subunità ribosomica alla catena di RNA.
o IF1: Allontana le subunità 30S e 50S del ribosoma.
o IF2 : Promuove il legame del complesso metionina formilatatRNA al ribosoma 30S. Richiede GTP che, tuttavia, sembra
non essere idrolizzato.
o IF3 :Lega l'mRNA alla subunità 30S e, al contempo, impedisce
il riaggancio con la subunità 50S.
Il primo passo per poter operare la traduzione è rappresentato dalla
separazione del ribosoma nelle sue due unità costituenti 30S e 50S. La
separazione del ribosoma è coadiuvata da IF1. In questo modo la
subunità libera 30S è capace di legare a sé, grazie all'ausilio di IF3, la
catena di mRNA, formando un ponte dove i IF si liberano.
· ALLUNGAMENTO: Il ribosoma è il luogo dove viene operato
l'allungamento. Possiamo idealmente suddividere il ribosoma in tre siti
che svolgono differenti ruoli. Il sito P (peptidico) è quello dove si trova,
temporaneamente, la catena in fase di allungamento; il sito A
(amminoacidico) è dove viene condotto il nuovo aminoacido legato al
tRNA da inserire nella catena; il sito E (exit) è il sito da dove viene
espulso il tRNA.
Dopo la formazione del complesso d’inizio, il ribosoma presenta due
siti disponibili per il legame dell’ amminoacil-tRNA: il sito di aggancio,
P, che contiene un fattore principale per la sintesi l’ fMetRNA, e il sito
A che espone la 2° tripletta del tRNA. L’ amminoacido-tRNA entra nel
sito A del ribosoma ed accoppia le sue basi con quelle dell’ mRNA.
L’affinità nel sito è bassa, cosi si necessita del fattore di allungamento
Ef-tu. L’amminoacil-tRNA si lega al sito A in un complesso ternario Eftu-amminoacil-tRNA-GTP. L’ fMet-tRNA non si posiziona nel sito A
ma nel sito P.
A causa dell’idrolisi in GTP per ogni molecola di amminoacil-tRNA, la
reazione produce il rilascio del complesso Ef-tu-GDP dal ribosoma. Il
complesso binario non è più in grado di legare amminoacil-tRNA. Cosi
grazie al fattore Ef-Ts vi è un processo di rigenerazione da Ef-tu-GDP a
Ef-tu-GTP.
I processi successivi sono noti come TRASLOCAZIONE (ovvero
passaggio da un sito all’altro):
o il tRNA, privo di amminoacido è rilasciato dal sito P;
o il peptidil-tRNA si muove dal sito A al P, dove si lega
all’mRNA attraverso un legame codon-anticodon;
o lo scorrimento dell’mRNA su di esso permette che il codon si
posizioni sul sito A.
La traslocazione sembra essere mediata dal fattore Ef-G che si lega al
ribosoma.
· TERMINAZIONE: è determinata dall’arrivo del codon di termine
(UAG, UGA o UAA) nel sito A, mediato dai fattori di rilascio:
o RF1: riconosce i codoni di terminazione;
o RF2: riconosce i codoni di terminazione;
o RF3: idrolizza il donatore di energia;
o RRF: è il fattore di rilascio totale: stacca l’mRNA che verrà
degradato e l’ultimo tRNA.