Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
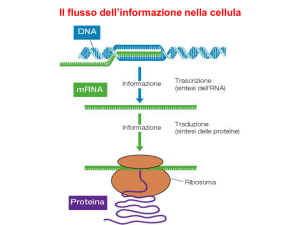
Nel Capitolo 3 abbiamo esaminato alcuni aspetti della traduzione. Abbiamo appreso come i ribosomi agiscano da fabbrica cellulare delle proteine e come l’RNA
transfer giochi un importante ruolo di adattatore, legando un amminoacido a un
estremo e un codone dell’RNA messaggero all’altro. Nei Capitoli 15 e 16 abbiamo
approfondito le nostre conoscenze sui meccanismi di inizio, allungamento e terminazione della traduzione, senza descrivere in dettaglio i ribosomi e i tRNA. In
questo capitolo discuteremo ancora del processo di traduzione, cercando di focalizzare la nostra attenzione su questi due componenti essenziali della cellula.
17.1 Ribosomi
Nel Capitolo 3 abbiamo descritto il ribosoma di E. coli come una particella
composta da due subunità con un caratteristico coefficiente di sedimentazione di 70S. Le due subunità ribosomali vengono definite in base al loro
coefficiente di sedimentazione 30S e 50S. Abbiamo inoltre appreso che la
subunità piccola decodifica l’mRNA mentre la subunità grande assembla gli
amminoacidi attraverso legami peptidici. In questo paragrafo concentreremo
la nostra attenzione sul ribosoma batterico, e in particolare su struttura, composizione, assemblaggio e funzione di questo ribosoma.
Struttura fine del ribosoma 70S
La cristallografia a raggi X fornisce un’ottima informazione strutturale, ma è
difficile da ottenere quando si analizza una particella grande e asimmetrica
come il ribosoma. Nonostante le difficoltà Harry Noller e colleghi a partire dal
1999 sono riusciti a ottenere strutture cristallografiche dei ribosomi del batterio
Thermus thermophilus. Questi studi hanno fornito strutture cristallografiche
estremamente dettagliate di ribosomi intatti, a una risoluzione di 7,8 Å.
Così nel 2001 Noller e colleghi hanno cristallizzato in Thermus thermophilus un complesso costituito dal ribosoma 70S, insieme a un analogo
dell’mRNA e del tRNA legati ai siti P ed E nel ribosoma.
Questi cristalli hanno fornito la struttura cristallografica a una risoluzione
di 5,5 Å, che rappresenta un notevole perfezionamento rispetto a quelle ottenute precedentemente. Gli stessi studiosi hanno inoltre cristallizzato questi
complessi sia in presenza che in assenza di un tRNA legato al sito A e ciò ha
permesso di ottenere la struttura del tRNA legato al sito A, per differenza, a
una risoluzione di 7 Å.
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W2 • La traduzione
Parte VI
La Figura 17.1 mostra la struttura cristallografica del ribosoma 70S. In (a-d)
è mostrato il ribosoma nei quattro orientamenti. L’rRNA 16S che fa parte
(a)
(d)
(b)
(e)
(c)
(f)
(g)
Figura 17.1 Struttura cristallografica del ribosoma 70S di
Thermus thermophilus. (a-d) Differenti visioni della struttura, ciascuna ruotata di 90° rispetto all’asse verticale. (a) La
subunità 30S è di fronte alla subunità 50S. Colori: l’rRNA 16S,
in celeste; le proteine ribosomali 30S, in blu; l’rRNA 23S, in
grigio, l’rRNA 5S, in blu scuro; le proteine ribosomali 50S, in
porpora; i tRNA in A, P ed E, rispettivamente in oro, in arancio,
in rosso; le proteine ribosomali sono indicate con i numeri. (e)
Subunità 50S in alto e subunità 30S in basso, i tre tRNA nel
centro. (f e g) Interfaccia tra la subunità 50S e 30S, rispettivamente, con la subunità 30S ruotata di 180° in modo tale da
rivelare i tRNA nell’interfaccia. (Da: Yusupov et al., Science
292: p. 885. ©2001 by the AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
della subunità 30S, è in celeste, mentre le proteine in blu. L’rRNA 23S della
subunità 50S è in grigio, l’rRNA 5S è in blu scuro, mentre le proteine sono
in porpora. I tRNA nei siti A, P ed E sono rispettivamente in oro, arancione
e rosso, sebbene questi siano difficili da vedere in (a-d) poiché si trovano
nel solco tra le due subunità ribosomali. Sono state identificate, inoltre, la
maggior parte delle proteine ribosomali. È da notare come la proteina L9 si
trovi lontano dal corpo centrale del ribosoma (in [a] di sinistra). La Figura
17.1e mostra un ribosoma visto dall’alto in cui sono chiaramente visibili i tre
tRNA. È da notare come il braccio dell’anticodone dei tre tRNA sia posizionato sulla parte inferiore della subunità 30S.
Le Figure 17.1 (f) e (g) mostrano le due subunità separate rivelando le posizioni dei tRNA. La particella 30S è stata ruotata di 180 gradi intorno al suo
asse verticale rendendo così visibili i tre tRNA. Si osserva che il solco dove i
tRNA sono legati è allineato con l’rRNA in entrambe le subunità; le proteine
sono molto più periferiche da questo punto di osservazione. Questo suggerisce che siano gli RNA ribosomali, e non le proteine, a stabilire interazioni
fondamentali con i tRNA, nel riconoscimento che avviene nella subunità 30S
e nella sintesi del legame peptidico nella subunità 50S. Inoltre, il ribosoma interagisce con porzioni conservate di tutti e tre i tRNA, permettendo il legame
esattamente allo stesso modo con tutti i differenti tRNA che incontra.
Sottolineiamo ancora, in (g), come il braccio dell’anticodone del tRNA
sia posizionato nella parte inferiore della subunità 30S. Gli anticodoni dei
tRNA nei siti A e P si trovano a una distanza di circa 10 Å, probabilmente
abbastanza distanti da non permettere il legame ai codoni adiacenti. Il ribosoma risolve questo problema mediante la torsione di 45 gradi dell’mRNA tra
i codoni nei siti A e P (Figura 17.2). Questo permette ai due codoni di essere
portati in stretta vicinanza, per essere così decodificati dai tRNA. La Figura
17.1f mostra come anche nella subunità 50S i tRNA nei siti A e P siano posti
tra loro in stretta vicinanza. Sebbene in questa figura sia difficile da notare,
i bracci accettori di questi due tRNA si inseriscono nella tasca peptidil-transferasica della subunità 50S con una distanza reciproca di circa 5 Å. Questa
stretta vicinanza è necessaria affinché sia facilitato il legame peptidico tra
l’amminoacido portato dal tRNA nel sito A e il peptide nascente legato al
tRNA nel sito P.
E
P
A
mRNA
Figura 17.2 Visione stereo dell’appaiamento codone-anticodone nei siti A e P. Sono
rappresentati tutti e tre i tRNA, il codice dei colori è come in Figura 17.1 (A, oro; P, arancio,
E in rosso). Le basi dei codoni e degli anticodoni sono rappresentate con il modello a sfere
e bastoncini in basso. Da notare il ripiegamento tra i codoni dell’mRNA di circa 45 gradi.
L’anticodone del tRNA nel sito E non è mostrato in quanto non si appaia con l’mRNA. (Da:
Yusupov et al., Science 292: p. 893. ©2001 by the AAAS.)
•
W3
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W4 • La traduzione
Parte VI
La struttura cristallografica del ribosoma 70S rivela 12 contatti tra le due
subunità (ponti inter-subunità), come mostrato in Figura 17.3. Molti di questi
contatti avvengono tra RNA piuttosto che tra proteine e tutte le interazioni
vicine ai siti di legame per i tRNA coinvolgono esclusivamente l’RNA. È da
notare come i ponti B2a, B3, B5 e B6 coinvolgano tutti un dominio a singola
elica (elica 44) dell’rRNA 16S nella subunità 30S (Figura 17.2). Quest’ elica
dà il maggior contributo ai contatti tra le due subunità e, come meglio vedremo nel corso di questo capitolo, ha anche un ruolo importante nel riconoscimento codone-anticodone. Poiché la traslocazione dei tRNA dal sito A al sito
P e dal sito P al sito E richiede un movimento di 20-50 Å, è molto probabile
che alcuni dei suddetti ponti siano dinamici: la formazione e la rottura di
questi legami potrebbe permettere queste traslocazioni.
La Figura 17.4 è una rappresentazione schematica dei ribosomi, dove sono
messi in rilievo tre punti importanti. Primo, esiste una larga cavità tra le due
subunità ribosomali in cui si possono posizionare i tre tRNA. Secondo, i
tRNA interagiscono con la subunità 30S mediante i loro anticodoni, i quali
legano l’mRNA che è a sua volta legato alla subunità 30S. Terzo, i tRNA interagiscono con la subunità 50S attraverso i loro bracci accettori. Infatti i bracci
accettori devono essere vicini durante la formazione del legame peptidico che
avviene nella subunità 50S. Durante questa reazione il peptide legato al braccio accettore del peptidil-tRNA nel sito P incontra l’amminoacido legato al
braccio accettore dell’amminoacil-tRNA nel sito A.
Nel 2005, Jamie Doudna Cate e colleghi raggiunsero un ulteriore risultato. Essi ottennero la struttura cristallografica del ribosoma 70S di E. coli a
una risoluzione di 3.5 Å. Non solo era la migliore risoluzione mai ottenuta di
un ribosoma 70S, ma era la struttura a lungo cercata del ribosoma di E. coli
che complementava decenni di dati biochimici e genetici. Prima che questa
struttura fosse disponibile, gli scienziati avevano cercato di adattare i dati
biochimici e genetici sul ribosoma di E. coli alla struttura del ribosoma di un
altro batterio (T. thermophilus). Questo probabilmente è un valido approccio
in molti casi, ma restano sempre dei dubbi, specialmente se si considerano gli
(a)
(b)
(e)
Figura 17.3 Visione dell’interfaccia che mostra i ponti tra le subunità. (a e b) Le subunità 50S e 30S rispettivamente. In entrambe le subunità, l’rRNA maggiore è in grigio, l’rRNA
5S in blu scuro, sopra la particella 50S, e le proteine in blu chiaro. I tRNA sono colorati
come in Figura 17.1, in oro, arancio e rosso. I ponti RNA-RNA tra le subunità sono in rosa,
mentre i ponti proteina-proteina sono in giallo. Tutti i ponti sono numerati (B1a, B2a, ecc.).
(Da: Yusupov et al., Science 292: p. 890. © 2001 by the AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
•
W5
70S
(a)
30S
tRNA
3
50S
Catena
crescente
Canale
di uscita
50S
30S
Sito di
legame
per EF-G
e EF-Tu
Sito di
(c)
riconoscimento
mRNA
(b)
A P
E
E
P
A
Sito
della peptidiltransferasi
Figura 17.4 Rappresentazione schematica del ribosoma (a) Il ribosoma 70S, dove si
mostra l’ampia cavità tra le subunità, capace di accogliere tre tRNA alla volta. Si osserva
il peptidil-tRNA nel sito P con la catena polipeptidica nascente che sporge attraverso il
canale di uscita della subunità 50S. È da notare come l’interazione tra la subunità 30S e il
tRNA coinvolge l’estremità finale dell’anticodone del tRNA, mentre l’interazione tra il tRNA
e la subunità 50S coinvolge principalmente il braccio accettore del tRNA. (b) La subunità
30S con un mRNA e i tre tRNA legati. (c) La subunità 50S con un mRNA e i tre tRNA legati.
(Da: Liljas, A., Function is structure. Science 285:2078, 1999. Copyright ©1999 American
Association for the Advancement of Science. Riproduzione autorizzata.)
ambienti estremamente differenti in cui questi due batteri crescono: l’intestino dei mammiferi e le sorgenti di acqua calda, rispettivamente.
La struttura ottenuta più recentemente contiene una grande quantità di
dati che non sono stati ancora completamente analizzati. Tuttavia sono emerse alcune interassanti scoperte. Inaspettatamente, ogni unità cellulare del cristallo conteneva due differenti strutture ribosomali, chiamate “ribosoma I”
e “ribosoma II”. Le principali differenze tra le due strutture sono dovute a
movimenti del corpo rigido dei domini ribosomali. Il più evidente di questi
movimenti era una rotazione della testa della particella 30S, di 6 gradi verso il sito E, dal ribosoma I al ribosoma II. Questa rotazione era ancora più
pronunciata (12 gradi verso sito E) quando si confrontava la struttura di T.
thermophilus con il ribosoma II di E. coli.
Questa rotazione della testa è quasi certamente collegata alla traslocazione dell’mRNA e dei tRNA attraverso il ribosoma. Infatti, nel 2000 Joachin
Frank e Rajendra Kumar Agrawal hanno effettuato uno studio di microscopia crio-elettronica dei ribosomi durante la traslocazione, e hanno notato che
le due subunità si muovono l’una rispetto all’altra. Inoltre, il canale per l’mRNA si allarga durante il processo per permettere il movimento, e si chiude
nuovamente dopo la traslocazione. Dunque, il ribosoma sembra agire come
un ingranaggio durante la traslocazione, e la rotazione della testa della particella 30S è probabilmente parte di questa azione ruotante.
Negli eucarioti i ribosomi citoplasmatici sono molto più complessi rispetto a quelli batterici. Nei mammiferi l’intero ribosoma ha un coefficiente di
sedimentazione di 80S ed è composto dalle subunità 40S e 60S. Gli organelli
r
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W6 • La traduzione
Parte VI
eucariotici hanno anch’essi ribosomi propri, anche se sono meno complessi,
confrontati con quelli batterici.
SOMMARIO La struttura cristallografica del ribosoma 70S di Thermus
thermophilus in un complesso con un analogo dell’mRNA e di tre tRNA,
ha chiarito che le posizioni e la struttura terziaria dei tre rRNA, e delle proteine ribosomali, può essere determinata. Inoltre sono state evidenziate la
forma e la posizione dei tRNA nei siti A, P ed E. I siti di legame per i tRNA
nel ribosoma sono composti principalmente dall’rRNA piuttosto che dalle
proteine. Gli anticodoni dei tRNA nei siti A e P sono posti in stretta vicinanza grazie a un appaiamento con i codoni adiacenti nella subunità 30S,
generando una torsione di 45 gradi dell’mRNA tra i due codoni. Nella
tasca peptidil-transferasica della subunità 50S i bracci accettori del tRNA
nei siti A e P sono a loro volta posti in stretta vicinanza, appena 5 Å. Questo è in accordo con la necessità dei due bracci di interagire durante la
formazione del legame peptidico. Sono stati inoltre osservati dodici punti
di contatto tra le due subunità, la maggior parte dei quali è mediata da
interazioni RNA-RNA.
La struttura cristallografica del ribosoma di E. coli contiene due strutture che differiscono per i movimenti del corpo rigido dei domini del ribososma, relativamente l’uno rispetto all’altro. In particolare, la testa della
particella 30S ruota di 6 gradi, e addirittura di 12 gradi se confrontata con
il ribosoma di T. thermophilus. Questa rotazione è probabilmente parte
dell’azione ruotante del ribosoma che avviene durante la traslocazione.
I ribosomi citoplasmatici degli eucarioti sono molto più grandi e complessi di quelli procariotici, ma i ribosomi degli organelli sono addirittura
più piccoli di quelli procariotici.
Composizione del ribosoma
Nel Capitolo 3 abbiamo appreso che la subunità ribosomale 30S di E. coli è
composta da una molecola di rRNA 16S e da 21 proteine ribosomali, mentre la subunità 50S contiene due molecole di rRNA (5S e 23S) e 34 proteine ribosomali. Gli rRNA sono relativamente facili da purificare eseguendo
un’estrazione fenolica che elimina le proteine lasciando l’rRNA in soluzione.
Le dimensioni dell’rRNA possono essere quindi determinate grazie a ultracentrifugazione.
A differenza degli rRNA le proteine ribosomali, essendo eterogenee, devono essere risolte con tecniche più fini. Le proteine ribosomali che compongono la subunità 30S possono essere visualizzate effettuando un’elettroforesi
mono-dimensionale in gel SDS PAGE; si possono così identificare delle bande che corrispondono a proteine con massa che va dai 60 agli 8 kD; tuttavia
alcune proteine non possono essere risolte con questa tecnica. Nel 1970 E.
Kaldschmidt e H.G. Wittmann hanno adottato una tecnica elettroforetica
bi-dimensionale per risolvere tutte le proteine che compongono entrambe le
subunità ribosomali. In questa versione della tecnica le due corse elettroforetiche successive sono state effettuate utilizzando un semplice gel nativo PAGE
(senza SDS) a due differenti condizioni di pH e a concentrazioni differenti di
acrilammide. In Figura 17.5 sono mostrati i risultati delle elettroforesi bidimensionali delle proteine ribosomali delle subunità 30S e 50S di E. coli. Ogni
macchia corrisponde a una proteina ribosomale; le proteine che compongono
la subunità 30S vengono chiamate S1-S21, mentre quelle che costituiscono la
subunità 50S sono chiamate L1-L33 (L34 non è visibile in questo esperimento). Il suffisso S viene attribuito alle proteine della subunità piccola (Small
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
(a)
Ribosomi e tRNA
•
W7
(b)
Figura 17.5 Elettroforesi bidimensionale delle proteine che compongono le subunità ribosomale 30S (a) e 50S (b) di
E. coli. Le proteine sono identificate dai numeri, S designa le proteine della subunità piccola, L quelle della subunità grande.
L’elettroforesi in prima dimensione (orizzontale) è stata effettuata a pH 8,6 con una concentrazione di acrilammide dell’ 8%;
l’elettroforesi in seconda dimensione (verticale) è stata effettuata a pH 4,6 con acrilammide al 18%. Le proteine S11 e L31 non
sono visibili sul gel, ma la loro posizione, determinata da altri esperimenti, è marcata con cerchi tratteggiati. (Da: Kaltschmidt,
E. e H.G. Wittmann, Ribosomal proteins XII: Number of proteins in small and large ribosomal subunits of Escherichia coli as
determined by two-dimensional gel electrophoresis. Proceedings of the National Academy of Sciences USA 67 (1970) f. 1-2,
pp. 1277-78.)
subunit), L alle proteine della subunità grande (Large subunit). I numeri che
identificano le proteine vengono assegnati partendo dalla proteina più grande
e terminano con la più piccola. Infatti S1 ha una massa di circa 60 kD, S21
pesa circa 8 kD. Come si può notare tutte le proteine sono abbastanza distanti
tra loro, ciò sottolinea una buona risoluzione.
I ribosomi eucariotici sono molto più complessi. La subunità 40S dei
mammiferi contiene l’rRNA 18S e circa 30 proteine. La subunità 60S è costituita da tre rRNA (5S, 5,8S e 28S) e circa 40 proteine. Come abbiamo appreso nei capitoli 10 e 16, gli rRNA 5,8S, 18S e 28S provengono tutti dallo stesso
trascritto, generato dall’RNA polimerasi I, mentre l’rRNA 5S viene trascritto
separatamente dall’RNA polimerasi III. Gli rRNA degli organelli eucariotici
sono spesso più piccoli della loro controparte procariotica: per esempio la
subunità piccola dei mitocondri di mammifero possiede un rRNA con un
coefficiente di sedimentazione di 12S.
SOMMARIO La subunità 30S di E. coli contiene l’rRNA 16S e 21 proteine
(S1-S21). La subunità 50S è costituita dagli rRNA 5S e 23S e da 34 proteine (L1-L34). I ribosomi citoplasmatici degli eucarioti hanno dimensioni
maggiori e contengono più molecole di rRNA e proteine rispetto a quelli
procariotici.
Assemblaggio del ribosoma
Una volta che le proteine ribosomali sono state purificate, possono essere aggiunte agli rRNA per ricostituire un ribosoma completamente attivo, almeno
in linea di principio. Masayasu Nomura e colleghi hanno raggiunto la prima
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W8 • La traduzione
Parte VI
ricostituzione con la subunità 30S del ribosoma di E. coli. I ricercatori hanno
infatti aggiunto le proteine ribosomali purificate della subunità 30S all’rRNA
16S ottenendo una particella attiva. La funzionalità della particella 30S ricostituita è stata valutata aggiungendo la subunità 50S nativa per formare una
particella 70S che può effettuare una traduzione in vitro.
Questa ricostituzione in vitro ha dimostrato come la particella 30S abbia
capacità autoassemblante. Infatti, per ottenere una subunità 30S attiva non è
stato necessario aggiungere nessun materiale estraneo. Questo ha permesso a
Nomura di dare risposta a una domanda molto interessante: In quale ordine i
componenti si assemblano? Egli ha risposto a questa domanda in molti modi.
In una prima serie di esperimenti, Nomura e colleghi hanno aggiunto ogni
proteina all’rRNA 16S dopo averle marcate radioattivamente; successivamente hanno misurato il loro legame, tramite conte allo scintillatore (senza l’rRNA 16S non si ha nessuna formazione di particella). Due proteine, S4 e S8, si
legano molto bene all’rRNA 16S (80-90%, tanto quanto la particella nativa
30S); altre quattro proteine vengono legate, anche se con minor efficienza,
mentre le restanti non vengono legate. Ciò suggerisce che S4 e S8 siano le
prime proteine a essere legate. Una volta che queste due proteine sono legate,
un’altra serie di proteine è capace di aggregarsi e dopo queste un’ulteriore serie
acquista la capacità di legarsi.
In Figura 17.6 è mostrato uno degli esperimenti in cui Nomura e colleghi
hanno cercato di individuare quali proteine fossero necessarie per il legame di
S12. In (a) si dimostra che S4, S7, S8, S13, S16 e S20 non sono sufficienti per il
legame di S12. In (b) si mostra, invece, che le proteine S4, S8, S16 e S17 sono
sufficienti. L’unica proteina non presente in (a) ma presente in (b) è S17. Questo
indica che la proteina S17 è importante per il legame di S12, anche se l’esperimento in sé non svela se la proteina S12 sia capace di legare direttamente S17.
(a)
(b)
(c)
0,8
A260 (
10
0,4
5
0
5
10
15 0
5
10 15 0
Numero frazione
5
10
Radioattività (cpm in centinaia)
)
15
15
Figura 17.6 Effetti delle proteine ribosomali sul legame di S12 al complesso proteine-16S rRNA. Nomura e colleghi hanno aggiunto [ 3H]S12 all’rRNA 16S e le seguenti
proteine: (a) S4, S7, S8, S13, S16, e S20; (b) S4, S8, S16, e S17; (c) tutte le proteine meno
S12. Dopo l’incubazione, i ricercatori hanno sottoposto il complesso a ultracentrifugazione
in gradiente di saccarosio, raccolto i campioni e determinato la radioattività tramite conta
delle emissioni radioattive (in rosso), e la loro assorbanza tramite spettrofotometria a 260
nm (in blu). Hanno osservato la maggior efficienza di legame nell’esperimento di controllo
(c), ma anche un significativo legame in (b), con solo quattro proteine e nessun legame in
(a) dove manca S17. (Da: Held, W.A Ballou, S. Mizushima e M. Nomura, Assembly mapping of 30S ribosomal proteins from Esherichira coli: Further studies. Journal of Biological
Chemistry 249:3109, 1974. Copyright ©1974 The American Society for Biochemistry & Molecular Biology, Bethesda, MD. Riproduzione autorizzata.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
Seguendo questa strategia Nomura ha costruito la mappa di assemblaggio mostrata in Figura 17.7. Le frecce nella mappa indicano che la proteina
facilita il legame di un’altra. Per esempio, S17 facilita il legame di S12. Più le
frecce sono spesse più il fenomeno è marcato, per esempio S17 ha un effetto
più marcato nel facilitare il legame di S12 rispetto a S5.
Questa mappa di assemblaggio riflette anche la situazione in vivo? Alcune
prove suggeriscono che sia possibile. Per esempio, le proteine che si assemblano per ultime nella mappa sono anche le proteine più esterne della particella ribosomale. L’indicazione è rafforzata anche dal fatto che queste proteine
sono più sensibili a enzimi proteolitici e ad agenti che interagiscono con le
proteine. Inoltre, mutanti di E. coli sensibili alle basse temperature producono
una subunità 30S difettiva, con solo nove proteine legate a temperature non
permissive. Queste sono presumibilmente le prime proteine che si assemblano
con l’rRNA 16S in vivo, e tutte meno una sono le prime proteine nella mappa
di assemblaggio.
Riassemblare la subunità 50S è molto più complicato. Gli rRNA di questa
subunità e le proteine ribosomali non sono, infatti, capaci di autoassemblarsi
in vitro. Questo è dovuto principalmente al carattere temperatura-dipendente del meccanismo di assemblaggio: infatti alle temperature necessarie per
l’assemblaggio in vitro le proteine si denaturano. Questo problema è stato
aggirato indirizzando gli esperimenti su un altro organismo: Bacillus stearothermophilus, un batterio termofilo. I ricercatori hanno pensato che un batterio capace di sopravvivere ad alte temperature debba possedere proteine più
resistenti al calore; queste proteine sarebbero, quindi, in grado di resistere
senza denaturarsi al processo di ricostituzione della subunità 50S in vitro. E
a conferma di questo è stato possibile ricostituire, in vitro, a una temperatura
di 60 °C, la particella 50S di B. stearothermophilus. Dal 1974 inoltre è stato
16S
RNA
S4
S8
S20
S7
S15
S17 S16
S13
S19
S9
S12
S18
S5
S14
S6
S10
S11
S3
S2
S21
S1
?
Figura 17.7 Mappa di assemblaggio della subunità ribosomale 30S. Le frecce scure
indicano una forte effetto di facilitazione al legame, le frecce sottili indicano un effetto
di minor entità. La freccia che parte dall’area ombreggiata più grande, e conduce a S11,
indica che alcune proteine di quest’area facilitano il legame di S11, ma non si sa quale. Le
proteine sopra la linea tratteggiata sono state trovate nella ricostituzione intermedia della
particella (RI). Quelle al di sotto della linea tratteggiata sono mancanti in questo intermedio. (Da: Held, W.A., B. Ballou, S. Mizushima e M. Nomura, Assembly mapping of 30S ribosomal protein from Esherichira coli: Further studies. Journal of Biological Chemistry 249:
3109, 1974. Copyright ©1974 The American Society for Biochemistry & Molecular Biology,
Bethesda, MD. Riproduzione autorizzata.)
•
W9
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W10 • La traduzione
Parte VI
possibile ricostituire in vitro anche la subunità 50S di E. coli utilizzando un
approccio a due fasi, che elimina la necessità di alte temperature. Nonostante i
successi, ancora non è disponibile una mappa di ricostituzione della subunità
50S di nessun organismo.
SOMMARIO L’assemblaggio in vitro della subunità ribosomale 30S inizia
dall’rRNA 16S. In seguito, le proteine si assemblano in modo sequenziale
e cooperativo; in tale processo le proteine aggiunte nei primi stadi aiutano
quelle successive a legare la particella durante l’assemblaggio.
Struttura fine della subunità 30S
Non appena identificata la sequenza degli rRNA di E. coli, i biologi molecolari hanno iniziato a proporre modelli delle loro strutture secondarie. L’idea
era quella di identificare la forma più stabile della molecola di rRNA cercando
tra le forme con più appaiamenti intramolecolari. La Figura 17.8 mostra un
modello della struttura secondaria dell’rRNA 16S, che è stata verificata da
esperimenti di cristallografia a raggi X della subunità ribosomale 30S. Come
si può notare, per questo modello esistono un gran numero di appaiamenti
di basi. Inoltre è possibile notare, evidenziati con colori differenti, almeno
tre domini che si ripiegano indipendentemente (uno dei quali possiede due
sottodomini).
Com’è correlata la disposizione tridimensionale dell’rRNA 16S al posizionamento delle proteine ribosomali nella subunità intatta? Il miglior modo
per rispondere a questa domanda è di effettuare degli esperimenti di cristallografia a raggi X. V. Ramakrishnan e colleghi nel 2000 hanno ottenuto la
struttura cristallografica della subunità 30S di Thermus thermophilus a una risoluzione di 3,0 Å. Circa nello stesso periodo un gruppo di ricercatori capeggiato da Francois Franceschi ha ottenuto la stessa struttura a una risoluzione
Figura 17.8 Struttura secondaria dell’rRNA 16S. Questa struttura è stata costruita in base a criteri di appaiamento ottimale delle
basi e in base a dati di cristallografia a raggi X della subunità 30S
di Thermus thermophilus. Due
eliche (H27 e H44) e l’ansa 530,
discussi più avanti nel capitolo,
sono evidenziati. In rosso, il dominio al 5’; in verde, il dominio centrale; in giallo, il dominio maggiore
al 3’; in azzurro, il dominio minore al 3’. (Da: Wimberly, B.T., D.E.
Brodersen, W.M. Clemons Jr.,
R.J. Morgan-Warren, A.P. Carter,
C. Vonrhein, T. Hartsch e V. Ramakrishnan, Structure of the 30S
ribosomal subunit. Nature 407 (21
Sep 2000) f. 2a, p. 329. Copyright
©MacMillan Magazines Ltd.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
di 3,3 Å. La struttura ottenuta da Ramakrishnan e colleghi conteneva tutte e
tre le regioni strutturate della molecola (più del 99% della molecola stessa) e
20 proteine ribosomali (il 95% delle proteine che costituiscono la subunità).
Le porzioni delle proteine mancanti nella struttura cristallografica erano solo
alle estremità non strutturate.
La Figura 17.9a mostra un diagramma stereo dell’rRNA 16S, e sottolinea
come tutte le caratteristiche importanti nella struttura del ribosoma siano mimate dall’rRNA, inclusa la testa, la piattaforma e la base (qui chiamata corpo).
Inoltre è possibile notare un collo che congiunge la testa al corpo e un becco (a
volte chiamato naso), che protrude dalla testa verso sinistra. Il codice dei colori è lo stesso adottato in Figura 17.8 e sottolinea come gli elementi di struttura
secondaria corrispondano a elementi tridimensionali indipendenti. In Figura
17.9b è mostrata un’immagine fronte/retro della subunità 30S con le proteine
legate all’RNA. Le proteine non apportano variazioni rilevanti nella forma
della subunità. In altre parole, nella struttura della particella le proteine non
contribuiscono a nessuna delle parti importanti. Con questo non si intende
che l’rRNA 16S possa raggiungere il ripiegamento visto in figura in assenza
delle proteine ribosomali, ma esattamente che l’rRNA costituisce la maggior
parte della subunità e che il ripiegamento dello scheletro di rRNA mima la
(a)
(b)
Fronte
Retro
Figura 17.9 Struttura cristallografica della subunità 30S. (a) Diagramma stereo dell’rRNA 16S della subunità 30S di Thermus thermophilus. Le caratteristiche più importanti sono
indicate di seguito: H, testa; Be, becco; Sh, spalla; N, collo; P, piattaforma; Bo, corpo; Sp,
sperone. I colori sono gli stessi della Figura 17.8. (b) Visione fronte/retro della subunità
30S con proteine (in porpora) aggiunte all’RNA (in grigio). La porzione frontale è convenzionalmente riconosciuta come la parte della subunità 30S che interagisce con la 50S.
Notare che queste sono due differenti visioni del ribosoma e non dei diagrammi stereo.
(Da: Wimberly, B.T., D.E. Brodersen, W.M. Clemons Jr., R.J. Morgan-Warren, A.P. Carter,
C. Vonrhein, T. Hartsch e V. Ramakrishnan, Structure of the 30S ribosomal subunit. Nature
407 (21 Sep 2000) f. 2b, p. 329. Copyright ©Macmillan Magazines Ltd.)
•
W11
Biologia molecolare 2/ed
Robert F. Weaver
W12 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
forma della particella intatta. La posizione delle proteine è coerente con quella
identificata con i metodi precedenti.
SOMMARIO Studi sulla sequenza dell’rRNA 16S hanno portato alla formazione di un modello della sua struttura secondaria (per appaiamento di
basi intramolecolare) della molecola. Studi di cristallografia a raggi X hanno
confermato le conclusioni di questa analisi. Questi studi hanno messo in
evidenza un esteso appaiamento di basi dell’rRNA 16S il cui ripiegamento
essenzialmente delinea i contorni della particella intera. Gli studi di cristallografia a raggi X hanno inoltre confermato la posizione di molte delle proteine della particella ribosomale 30S.
Interazioni degli antibiotici con la subunità 30S Ramakrishnan e colleghi hanno anche ottenuto la struttura cristallografica della subunità 30S
in associazione con tre differenti antibiotici: spectinomicina, che inibisce la
traslocazione; streptomicina, che causa errori di traduzione; paromomicina,
che aumenta il tasso di errore attraverso un altro meccanismo. Questi dati,
valutati insieme con la struttura cristallina della sola subunità 30S, hanno
apportato ulteriori informazioni sul meccanismo di traduzione.
I ricercatori hanno sovrapposto nella struttura della subunità 30S la posizione di tre amminoacil-tRNA dalla struttura del ribosoma 70S intero (vedi
Figura 17.1). La Figura 17.10 (a e b) mostra due differenti visioni del posizio-
(c)
(a)
(b)
Figura 17.10 Posizione dei siti A, P ed E nella subunità ribosomale 30S. (a) e (b) Due differenti visioni stereo della posizione
dedotta dei bracci dell’anticodone e dei codoni dell’mRNA sulla subunità ribosomale 30S. I bracci degli anticodoni sono colorati
in fucsia (sito A), rosso (sito P) e giallo (sito E). I codoni dell’mRNA sono colorati in verde (sito A), blu (sito P), fucsia tratteggiato
(sito E). (c) La struttura secondaria dell’rRNA 16S mostra le regioni coinvolte in ciascuno dei tre siti, il codice dei colori è lo stesso
descritto per i bracci dell’anticodone in (a) e in (b): fucsia sito A, rosso sito P, giallo sito E. (Da: Carter, A.P., W.M. Clemons Jr., D.E.
Brodersen, R.J. Morgan-Warren, B.T. Wimberly e V. Ramakrishnan, Functional insights from the structure of the 30S ribosomal
subunit and its interactions with antibiotics. Nature 407 (21 Sep 2000) f. 1, p. 341. Copyright ©MacMillan Magazines Ltd.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
namento degli anticodoni degli amminoacil-tRNA legati al sito A, P ed E, e
dei codoni di un ipotetico mRNA nella subunità 30S. È interessante notare
come i codoni e gli anticodoni nei siti A e P risiedano in una regione vicina
al collo della subunità 30S, regione che è quasi priva di proteine. Quindi il riconoscimento codone-anticodone avviene in un ambiente povero di proteine
ma ricco di segmenti dell’rRNA 16S. La Figura 17.10c fa risaltare quale sia la
regione dell’rRNA 16S ssociata alla posizione dei tre siti del ribosoma.
Le posizioni che assumono i tre antibiotici nella subunità 30S danno indicazioni sulle due attività della subunità stessa: traslocazione e decodificazione (riconoscimento codone-anticodone). La geometria della subunità 30S
suggerisce che il meccanismo di traslocazione potrebbe coinvolgere i movimenti della testa rispetto al corpo. L’antibiotico spectinomicina, che inibisce
la traslocazione, è una molecola costituita da tre anelli rigidi. Questa molecola
interagisce con la subunità 30S in una regione della testa che probabilmente
svolge la funzione di perno nella traslocazione, bloccandone i movimenti.
La streptomicina incrementa il tasso di errore nella traduzione interferendo con il riconoscimento codone-anticodone e con il meccanismo di correzione delle bozze. La posizione che occupa l’antibiotico nella subunità 30S
(Figura 17.11) fornisce alcune indicazioni sul suo meccanismo di azione. La
streptomicina si associa molto vicino al sito A, cioè dove avviene la decodificazione. In particolare ha una forte interazione con A913 dell’elica H27
dell’rRNA 16S.
Questa interazione della streptomicina è importante in quanto si pensa
che l’elica H27 abbia due appaiamenti alternativi durante la traduzione, che
ne influenzano il grado di accuratezza. Il primo profilo è chiamato stato ram
(da Ribosome AMbiguity, ambiguità del ribosoma). Come dice il nome, questo schema di appaiamento dell’elica H27 stabilizza sia le interazioni codoneanticodone, sia quelle con anticodoni diversi, quindi il tasso di errore nella
traduzione è maggiore (le strutture cristalline ottenute da Ramakrishnan e
colleghi contengono l’elica H27 nello stato ram). Il profilo di appaiamento
alternativo è restrittivo e richiede un accurato appaiamento tra codone e
anticodone. Se il ribosoma è bloccato nello stato ram, accetta facilmente gli
amminoacil-tRNA non complementari e non può passare allo stato restrittivo necessario per l’attività di correzione delle bozze. Questo comporta una
traduzione non accurata. Se il ribosoma è bloccato nello stato restrittivo la
traduzione è iperaccurata, raramente avvengono errori di appaiamento, ma
gli amminoacil-tRNA hanno difficoltà a legarsi nel sito A, quindi anche in
questo caso la traduzione è inefficiente.
L’interazione tra streptomicina e subunità 30S indica che l’antibiotico
stabilizza lo stato ram. Ciò potrebbe ridurre l’accuratezza con due meccanismi distinti. Primo, potrebbe favorire lo stato ram durante la decodificazione
permettendo appaiamenti codoni-anticodoni non complementari. Secondo,
potrebbe inibire la transizione allo stato restrittivo necessario per l’attività di
correzione delle bozze.
Alcune mutazioni della proteina ribosomale 12S conferiscono resistenza o
dipendenza alla streptomicina. Quasi tutte queste mutazioni si trovano in una
regione di S12 che stabilizza i nucleotidi 908-915 dell’elica H27 e 524-527 di
H18. Queste regioni dell’rRNA 16S sono anche coinvolte nella stabilizzazione dello stato ram. Queste considerazioni sperimentali hanno portato Ramakrishnan e colleghi a proporre due ipotesi. Primo, le mutazioni di S12 che
causano resistenza alla streptomicina destabilizzano lo stato ram sufficiente
Ribosomi e tRNA
•
W13
Biologia molecolare 2/ed
Robert F. Weaver
W14 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
(a)
(b)
(c)
(d)
Figura 17.11 Interazione della streptomicina con la subunità ribosomale 30S (a) Diagramma stereo della streptomicina e
delle regioni vicine alla subunità ribosomale 30S. La streptomicina è rappresentata con il modello a sfere e bastoncini con una
nuvola di densità elettronica (ottenuta per differenza della densità elettronica della subunità 30S con e senza l’antibiotico). Sono
inoltre mostrate le eliche prossimali dell’rRNA 16S. Va notata in particolare l’elica H27 (giallo) che è fondamentale per l’attività
di questo antibiotico. Notare, inoltre, la posizione dell’unica proteina presente nei pressi del sito A (S12) (rispettivamente sito A,
rosa chiaro; S12, rosso) importante per l’azione della streptomicina. Gli amminoacidi importanti nel fenomeno di resistenza alla
streptomicina sono mostrati in rosso. (b) Interazioni di una specifica regione della molecola di streptomicina (gli anelli numerati I,
II e III), con gli atomi prossimali della subunità 30S. Notare le interazioni tra A913 dell’elica H27 con la lys45 della proteina S12. (c)
Un’altra visione stereo della streptomicina con le regioni vicine. Il codice dei colori è lo stesso di(a). Notare ancora l’elica H27 ( in
giallo) e S12 (in rosa chiaro). (d) Localizzazione del sito di legame della streptomicina all’interno della subunità 30S. La streptomicina è rappresentata in rosso nel punto in cui le eliche dell’rRNA 16S colorate convergono. (Da: Carter, A.P., W.M. Clemons Jr.,
D.E. Brodersen, R.J. Morgan-Warren, B.T. Wimberly e V. Ramakrishnan, Functional insights from the structure of the 30S ribosomal subunit and its interactions with antibiotics. Nature 407 (21 Sep 2000) f. 5, p. 345. Copyright ©Macmillan Magazines Ltd.)
a controbilanciare lo stato ram prodotto dall’antibiotico. Questo permette al
ribosoma di lavorare bene anche in presenza di streptomicina. Secondo, le
mutazioni in S12 che causano dipendenza da streptomicina destabilizzano lo
stato ram; in questo modo i ribosomi mutanti necessitano dell’antibiotico per
stabilizzare lo stato ram stesso. Di conseguenza il ribosoma non può portare
avanti una normale traduzione in assenza della streptomicina.
In altre parole, una traduzione accurata ed efficiente dipende da un equilibrio tra lo stato ram e quello restrittivo del ribosoma. La streptomicina può
spostare l’equilibrio verso inaccuratezza ed efficienza favorendo lo stato ram,
mentre le mutazioni in S12 possono spostare questo equilibrio verso accuratezza e inefficienza favorendo lo stato restrittivo. Per verificare questa ipotesi
gli studiosi necessiteranno della struttura della subunità 30S nello stato restrittivo per poterla poi confrontare con la struttura dello stato ram fornita da
Ramakrishnan e colleghi.
Anche la paromomicina diminuisce l’accuratezza della traduzione, legandosi al sito A. Nel 2000 Ramakrishnan e collaboratori hanno dimostrato che
questo antibiotico si lega al solco maggiore dell’elica H44 ed espone le basi
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
A1492 e A1493. In questo modo, essa forza le suddette basi fuori dal solco
maggiore esponendole a una interazione con il solco minore tra il codone e
l’anticodone nel sito A. Le basi A1492 e A1493 sono universalmente conservate e assolutamente necessarie per la traduzione; mutazioni in ciascuna delle
due basi sono letali.
Queste considerazioni hanno condotto alle seguenti ipotesi: durante la
normale decodificazione le basi A1492 e A1493 vengono esposte, formando
legami idrogeno con i gruppi ossidrile in posizione 2’−OH degli zuccheri,
nel solco minore formato dall’appaiamento codone-anticodone nel sito A.
Ciò stabilizza l’interazione codone e anticodone ed è rilevante, in quanto un
appaiamento di sole tre paia di basi ha una bassa stabilità. L’estrusione delle
due basi richiede energia, ma la paromomicina elimina la necessità di questa
energia, forzando le basi a protrudere. In tal modo la paromomicina stabilizza
il legame dell’amminoacil-tRNA includendo nel sito A gli amminoacil-tRNA
non corretti, incrementando così il tasso di errore.
Nella struttura cristallografica della subunità 30S, in presenza di paromomicina non erano presenti codoni e anticodoni, in questo modo non si aveva
una prova diretta delle interazioni tra le basi A1492, A1493 e il solco minore
del duplex formato dall’appaiamento codone-anticodone.
Nel 2001 Ramakrishnan e collaboratori hanno fornito una prova diretta
della loro ipotesi. Essi hanno immerso i cristalli delle subunità ribosomali 30S
di Thermus thermophilus in una soluzione contenente un oligonucleotide di
17 nt corrispondente al braccio anticodone del tRNA della fenilalanina e un
oligonucleotide di sei uridine che codifica la difenilalanina. Queste molecole
sono entrambe abbastanza piccole da poter essere inserite nella loro posizione
caratteristica della subunità 30S, mimando rispettivamente l’anticodone di
un amminoacil-tRNA e il codone di un mRNA.
La Figura 17.12 mostra un’immagine tridimensionale delle regioni selezionate nella struttura cristallografica di questo complesso. In (a) si mostra
chiaramente che la base A1493 dell’elica 44 contatta i gruppi 2’−OH degli
zuccheri di entrambi i nucleotidi nel solco minore del primo appaiamento
codone-anticodone (U1-A36). In (b) si mostrano le interazioni meno favorevoli con la base A1493 se A36 dell’anticodone è sostituita da G. In (c) la base
A1492 dell’elica 44 e G530 dell’ansa 530 dell’rRNA 16S contattano i gruppi 2’−OH degli zuccheri di entrambi i nucleotidi nel secondo appaiamento
codone-anticodone (U2-A35). Questi sono i due appaiamenti più stringenti
nel riconoscimento codone-anticodone, e vengono stabilizzati dall’estrusione
delle basi A1492-A1493, in aggiunta ad altri elementi ribosomali.
Il terzo appaiamento codone-anticodone (d, appaiamento vacillante U3G34) è anche stabilizzato da elementi ribosomali, inclusi G530 e P48 della
proteina ribosomale S12, ma non da A1492-A1493.
In Figura 17.13 è riassunto quello che la struttura cristallografica suggerisce
sul ruolo di A1492, A1493 e della paromomicina nel riconoscimento codoneanticodone. Confrontando (a) e (b) possiamo notare come la paromomicina
si leghi all’elica 44 forzando A1942 e A1943 fuori dell’elica esponendoli nel
sito A. Invece (c) illustra il riconoscimento codone-anticodone in assenza di
paromomicina e mostra come A1942 e A1943 occupino la stessa posizione
anche in presenza di paromomicina. In questo caso le due basi dell’rRNA
sono in posizione perfetta per inserirsi tra la prima e la seconda coppia di
basi del solco minore dell’elica formata dall’appaiamento codone-anticodone,
occupando la posizione dei ribosi. In (d) si illustra la stessa struttura in pre-
Ribosomi e tRNA
•
W15
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W16 • La traduzione
Parte VI
s12
16S RNA Ser50 16S RNA
A1492
G530 C518
16S RNA A1493
(a)
codone U1
anticodone A36
(c)
anticodone A35
codone U2
A1493
16S RNA
C518
G530
16S RNA
C1054
s12
P48
(b)
anticodone
errato G36
codone U1
anticodone G34
(d)
codone U3
Figura 17.12 Immagini stereo delle interazioni nell’appaiamento codone-anticodone ed elementi della subunità ribosomale 30S. (a) Legame della base A1493 dell’elica 44 nel solco minore formato dall’appaiamento di U1-A36 (b) come in (a),
inoltre è mostrato il risultato della sostituzione di A36 nell’anticodone con G, si forma così un appaiamento instabile G-U tra
G36 e U1. Ora le posizioni di G36 ( rosso) e U1 (violetto) possono essere contrastate con le normali posizioni di A36 (oro) e U1
(porpora). È da notare che U1 è stato scalzato in modo tale da aver perso la sua normale interazione con A1493 (rappresentato
dalla linea nera tratteggiata). Ciò destabilizza l’interazione e aiuta il ribosoma a discriminare tra un appaiamento A-U codoneanticodone corretto e un appaiamento G-U sbagliato che coinvolge la prima base nel codone. (c) Legame di A1492 e G530 nel
solco minore generato dall’accoppiamento delle basi U2-A35. (d) L’appaiamento instabile U3-G34 porta U3 a interagire con
G530 e, tramite Mg2+ (sfera fucsia), con C518 e la prolina 48 della proteina S12. La base C1054 dell’rRNA 16S è in prossimità di
G34. (Da: da Ogle et al., Science 292: p. 900 ©2001 by the AAAS.)
Figura 17.13 Struttura di parte del sito A in presenza o assenza di tRNA, mRNA e paromomicina. (a) Il sito A da solo.
Notare la posizione di A1492 e A1493 dell’elica 44. Le posizioni di queste basi sono molto flessibili. (b) Il sito A in presenza di
paromomicina. Il legame dell’antibiotico all’interno dell’elica 44 forza le basi A1492 e A1493 a posizionarsi all’esterno dell’elica
e all’interno del sito A. (c) Il sito A in presenza dell’mRNA e del braccio dell’anticodone (ASL) del t-RNA posizionato nel sito A.
Le basi A1492 e A1493 assumono la stessa posizione nel sito A che potrebbero avere in presenza di sola paromomicina. (d)
Come in (c) ma in presenza di paromomicina. L’antibiotico determina una piccola differenza poiché le basi A1492 e A1493 hanno
comunque interazioni con il sito A. (Da: Ogle et al., Science 292: p. 900. ©2001 by the AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
senza di paromomicina, e ancora, non suggerisce grandi variazioni rispetto
alla struttura senza antibiotico.
Queste evidenze sono coerenti con l’ipotesi che la paromomicina, estroflettendo dall’elica 44 le basi A1492 e A1943, paghi parte del costo energetico
necessario per l’appaiamento codone-anticodone a carico del ribosoma. Ciò
rende più facile il legame tra codoni e anticodoni non complementari, aumentando la frequenza di errore della traduzione.
SOMMARIO La subunità 30S del ribosoma svolge due ruoli. Facilita la caratteristica decodificazione tra anticodone dell’amminoacil-tRNA e codoni
dell’mRNA, attuando il meccanismo di correzione delle bozze. Inoltre partecipa al meccanismo di traslocazione. Le strutture cristallografiche della
subunità 30S, in presenza di tre antibiotici che influenzano questi due meccanismi, hanno chiarito alcuni aspetti della decodificazione e della traslocazione. La spectinomicina lega la subunità 30S, nei pressi del collo, dove
interferisce con i movimenti della testa necessari per la traslocazione. La
streptomicina interagisce con il sito A della subunità 30S e stabilizza lo stato
ram del ribosoma. Questo riduce la precisione della traduzione perché facilita il legame degli amminoacil-tRNA non complementari al sito A; inoltre
impedisce lo spostamento del processo di traduzione verso lo stato restrittivo necessario per l’attività di correzione delle bozze. La paromomicina lega
il solco minore dell’elica H44 dell’rRNA 16S vicino al sito A. Ciò estrude le
basi A1492 e A1493, stabilizzando l’appaiamento codone-anticodone. Il
processo di estrusione normalmente richiede energia; ma l’antibiotico forza
questo processo stabilizzando le basi in questa posizione. Questo stato del
sito A stabilizza le interazioni codone-anticodone, incluse le interazioni non
complementari; il risultato è una diminuzione nella fedeltà di traduzione.
Interazione della subunità 30S con i fattori di inizio della traduzione Abbiamo visto nel Capitolo 15 come IF1 aiuti gli altri fattori di inizio
nello svolgere i loro ruoli. Un altro ruolo svolto da IF1 è quello di prevenire il
legame dell’amminoacil-tRNA al sito A del ribosoma prima del termine della
di fase di inizio. Questo blocco del sito A probabilmente svolge due ruoli. Primo, finché la subunità 50S non è associata al complesso di inizio, l’azione di
correzione delle bozze diretta da EF-Tu non può avvenire. Il blocco del sito A
impedisce un legame non corretto degli amminoacil-tRNA conferendo precisione alla traduzione. Secondo, assicura che il legame dell’amminoacil-tRNA
iniziatore si leghi al sito P anziché al sito A.
Ramakrishnan e collaboratori hanno determinato la struttura cristallografica della subunità ribosomale 30S legata a IF1 di Thermus thermophilus.
L’immagine in Figura 17.14 (b e c) mostra chiaramente che IF1 si lega al sito
A della particella 30S occludendone l’accesso. IF1 occupa gran parte dello
spazio necessario per il legame del tRNA al sito A.
Gli esperimenti di cristallografia mostrati non includono IF2, ma dal Capitolo 15 sappiamo che IF1 aiuta IF2 a legare l’fMet-tRNA al sito P, inoltre è noto
che IF1 e IF2 interagiscono tra loro. Quindi, il legame di IF1 al sito A potrebbe
permettere a IF1 di indirizzare IF2 al legame della subunità ribosomale 30S,
facilitando in qualche modo l’associazione dell’fMet-tRNA al sito P.
Infine, abbiamo appreso nel Capitolo 15 che IF1 facilita la dissociazione
e la riassociazione delle due subunità ribosomali, lasciando quindi invariato
l’equilibrio tra i due stati. Infatti, unicamente in presenza di IF3, che inibisce la
Ribosomi e tRNA
•
W17
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W18 • La traduzione
(a)
Parte VI
(c)
(b)
N
H
P
Sh
Bo
(d)
Figura 17.14 Struttura cristallografica del complesso IF-1/
subunità 30S. (a) Ingrandimento che mostra IF1 in magenta,
l’elica 44 dell’rRNA 16S in turchese, l’ansa 530 dell’rRNA 16S in
verde e la proteina S12 in arancio. (b) Una visione di insieme
del complesso con gli stessi colori riportati in (a). Il resto della
subunità 30S è in grigio. (c) Visione d’insieme meno IF1; sono
mostrate le posizioni dei tRNA nel sito A (porpora), nel sito P
(arancione scuro) e nel sito E (giallo-verde). Gli altri colori come
in (a). Da notare la sovrapposizione tra il tRNA nel sito A e la posizione di IF1 in (a). (d) Immagine stereo della stretta associazione
delle basi A1492 e A1493 (strutture a bastoncini rosse) dell’elica
44 e le tasche formate da IF1 e S12. R41 e R46 (in blu) sono le arginine di IF1 che interagiscono con A1492 e A1493. Gli altri colori
sono come in (a) (Da: Carter et al., Science 291: p. 500. ©2001
by the AAAS.)
riassociazione, IF1 si comporta da fattore di dissociazione. Tutte le immagini in
Figura 17.14 mostrano uno stretto contatto tra IF1 e l’elica 44 dell’rRNA 16S
nella subunità ribosomale 30S. È noto, inoltre, che l’elica 44 contatta estensivamente la subunità ribosomale 50S. Ramakrishnan e colleghi hanno quindi
ipotizzato che il contatto tra IF1 e l’elica 44 modifica la struttura dell’elica; ciò
potrebbe mimare lo stato di transizione tra l’associazione e la dissociazione delle
subunità ribosomali. Questo potrebbe spiegare come IF1 possa accelerare sia
l’associazione che la dissociazione delle subunità ribosomali.
SOMMARIO La struttura cristallografica di IF1 legato alla subunità ribosomale 30S indica un’interazione di IF1 con il sito A. Il legame in questa posizione blocca chiaramente l’accesso dell’fMet-tRNA al sito A e promuove
il legame del tRNA iniziatore al sito P grazie a interazioni tra IF1 e IF2. IF1
inoltre interagisce con l’elica 44 della subunità 30S; ciò potrebbe spiegare
come IF1 possa aumentare la velocità di associazione e di dissociazione
delle due subunità.
Struttura fine della subunità 50S
Nel 2000 Peter Moore, Thomas Steiz e colleghi hanno posto una pietra miliare nello studio della struttura del ribosoma e nel campo della cristallografia
a raggi X, determinando la struttura cristallografica della subunità ribosomale 50S a una risoluzione di 2,4 Å. Hanno perfezionato gli studi sulla subunità 50S nell’archea Haloarcula marismortui, poiché da questi organismi
possono essere preparate le subunità 50S disponibili per la diffrazione a raggi
X. L’immagine mostrata in Figura 17.15, include 2833 nucleotidi dei 3045
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
•
W19
negli rRNA della subunità (tutti i 122 nucleotidi dell’rRNA 5S) e 27 delle 31
proteine. Le altre proteine non sono ben ordinate nel cristallo e non possono
essere analizzate accuratamente.
È presente una chiara differenza tra le due subunità nella struttura terziaria dei loro rRNA. Mentre l’rRNA 16S della subunità 30S assume una struttura a tre domini, l’rRNA 23S della subunità 50S ha una struttura monolitica senza nessun chiaro confine tra i domini. Moore, Steiz e colleghi hanno
pensato che il motivo di questa differenza fosse dovuto alla necessità di una
cooperazione tra gli rRNA della subunità piccola, mentre ciò non è necessario
per gli rRNA della subunità 50S.
Le immagini più piccole in Figura 17.15 mostrano la localizzazione delle
proteine nella subunità 50S. Come abbiamo visto precedentemente in questo
capitolo, le proteine della subunità 50S generalmente non si trovano all’inter-
Figura 17.15 Struttura cristallografica della subunità ribosomale 50S di Haloarcula marismortui. Le tre strutture grandi
mostrano la subunità in tre differenti orientamenti: (a) vista davanti o a “corona” (così chiamata perché la forma assomiglia a
una corona a tre punte); (b)vista da dietro (corona ruotata di 180 gradi); (c) vista dal basso, che mostra, in posizione centrale,
la fine del tunnel per l’uscita del polipeptide nascente. L’RNA è in grigio mentre le proteine in oro. Le tre strutture più piccole in
basso a sinistra hanno gli stessi orientamenti con segnate le posizioni delle proteine ribosomali. La lettera “e” dopo alcuni numeri indica le proteine in archea che hanno omologie unicamente con gli eucarioti (e non con i batteri). (Da: Ban, N., P. Nissen,
J. Hansen, P.B. Moore e T.A. Steitz, The complete atomic structure of the large ribosomal subunit at 2.4 Å resolution. Science
289 (11 Aug 2000) f. 7, p. 917. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W20 • La traduzione
Parte VI
faccia delle due subunità, e in particolare al centro, dove è presente il sito attivo peptidil-transferasico. Questa ipotesi fu provocatoria in quanto (Capitolo
18) non era nota quale delle due componenti, proteine o RNA, avesse attività
peptidil-transferasica.
Per determinare quali proteine siano presenti nel sito attivo peptidil-transferasico è necessario identificare il sito attivo in una struttura cristallografica. Per fare questo, Moore, Steiz e colleghi hanno immerso i cristalli delle
subunità 50S con due differenti analoghi dei substrati peptidil-transferasici;
hanno quindi eseguito analisi cristallografiche e calcolato differenze nelle
mappe elettroniche. In questo modo, la densità di localizzazione elettronica
corrispondeva ai substrati analoghi e di conseguenza ai siti attivi. Un analogo
(CcdAp-puromicina) è stato progettato da Michael Yarus per mimare lo stato
di transizione o un intermedio durante la reazione peptidil-tranferasica. Il
composto è stato quindi chiamato “analogo di Yarus”.
La Figura 17.16 mostra che l’analogo di Yarus risiede nel solco sulla superficie della subunità 50S, proprio dove si pensa sia presente il sito attivo. In
questa regione è presente solo l’RNA ma non le proteine. Per l’altro analogo è
stata fatta la stessa osservazione. La Figura 17.17 è un modello del sito attivo
in cui è stato rimosso tutto l’RNA; in questo caso si può osservare come le
proteine siano lontane dal fosfato dell’analogo di Yarus, il quale corrisponde
all’atomo di carbonio tetraedico, nello stato di transizione al centro del sito
attivo. La proteina più vicina L13 è a una distanza dal centro del sito attivo di
più di 18 Å, una distanza troppo grande affinché questa proteina possa avere
un ruolo diretto nella catalisi.
Se la proteina non è presente nel sito attivo, l’RNA deve avere attività
enzimatica. La struttura cristallografica rileva che l’adenina 2486 (A2486),
che corrisponde all’adenina 2451 in E. coli, è in stretta vicinanza al carbonio
Figura 17.16 Posizione del sito attivo peptidil-transferasico. Immagine a corona della subunità 50S come in Figura
17.14, con l’analogo di Yarus, posizionato nel sito attivo peptidil-trasferasico, in verde. È da notare l’assenza di proteine
(oro) vicine al sito attivo. (Da: Ban, N., P. Nissen, J. Hansen,
P.B. Moore e T.A. Steitz, The complete atomic structure of
the large ribosomal subunit at 2.4 Å resolution. Science 289
(11 Aug 2000) f. 2, p. 907. Copyright ©AAAS.)
Figura 17.17 Immagine del sito attivo peptidil-transferasico ottenuto rimuovendo gli rRNA. Il fosfato dell’analogo
di Yarus, al centro del sito attivo, è mostrato in fucsia; la lunga
coda color fucsia rappresenta il polipeptide nascente. Sono
inoltre mostrate le quattro proteine ribosomali più vicine al
sito attivo, con le distanze relative riportate in Å. (Da: Nissen,
P., J. Hansen, N. Ban, P.B. Moore e T.A. Steitz, The structural
basis of ribosome activity in peptide bond synthesis. Science 289 (11 Aug 2000) f. 6b, p. 924. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Capitolo 17
Copyright © 2009 - The McGraw-Hill Companies srl
Ribosomi e tRNA
•
W21
tetraedico del sito attivo. Questa base è conservata nei ribosomi in ogni specie
esaminata in tutti e tre i regni viventi, e ciò evidenzia l’importanza del suo
ruolo. Inoltre, il cloramfenicolo e la carbomicina inibiscono l’attività peptidiltransferasica, legandosi vicino ad A2451 in E. coli. E le cellule di E. coli con
mutazioni in A2451 sono resistenti al cloramfenicolo, indicando che questa
base è coinvolta nella reazione.
Se questo modello è corretto, le mutazioni in A2486 dovrebbero ridurre di
diversi ordini di grandezza l’attività peptidil-transferasica. Alexander Mankin
e colleghi hanno verificato questa ipotesi nel 2001, mediante il riassemblaggio della subunità 50S di Thermus Aquaticus, dalle proteine isolate e dagli
rRNA 23S con tutte le tre possibili mutazioni in A2451, la base equivalente
di A2486 in H. marismortui. I ricercatori hanno analizzato l’attività peptidiltransferasica delle subunità 50S ricostituite mediante quattro saggi differenti,
compresa la reazione di frammentazione descritta nel Capitolo 16. Nessuna
delle mutazioni causa un evidente decremento nell’attività; ciascun rRNA
23S mutato presenta almeno il 44% dell’attività del selvatico in almeno uno
dei saggi.
Se l’A2486 non gioca il principale ruolo catalitico, chi lo svolge? Nel 2004
Scott Strobel e colleghi evidenziarono la possibile implicazione del gruppo
2’-OH dell’adenosina terminale del peptidil-tRNA nel sito P. La Figura 17.18
mostra la posizione di questo gruppo 2’-OH rispetto all’amminoacido nel
sito A, che produce un attacco nucleofilo sul carbonio carbonilico che lega il
peptide al tRNA nel sito P. Questo attacco risulterebbe nell’unione del peptide nel sito P all’amminoacil-tRNA nel sito A, una transpeptidazione, la
reazione catalizzata dalla peptidil-trasnsferasi. È chiaro che il gruppo 2’-OH
è molto ben posizionato per giocare un ruolo in questa reazione formando
un legame idrogeno con uno dei protoni sul gruppo amminico, che agisce da
nucleofilo migliore.
Se questa ipotesi fosse corretta, rimuovendo l’ossigeno dalla posizione 2’
dell’adenosina terminale (A76) del peptidil-tRNA si impedirebbe l’attività peptidil-transferasica. Strobel e colleghi saggiarono questa ipotesi in due
modi: sostituendo il gruppo 2’-OH con un atomo di idrogeno (2’-deossiadenosina, dA) oppure con un atomo di fluoro (2’-deossi, 2’-fluoroadenosina, fA). Quando essi effettuarono entrambe queste sostituzioni
sull’adenosina terminale, l’attività peptidil-transferasica veniva
tRNA
severamente inibita. Per effettuare i loro saggi, Strobel e colleghi
CH2 O
A
utilizzarono [35S]fMet-tRNA caricato nel sito P, e poi Lys-tRNA
tRNA
nel sito A. Questo Lys-tRNA era addizionato in esperimenti separati in tre forme rispetto all’adenosina terminale: normale, dA, ed
CH2 O
A
H OH
fA. Quindi, permisero l’attività peptidil-transferasica e un giro di
O
O
traslocazione, posizionando [35S]fMet-Lys-tRNA nel sito P. Poi ag- O C
N
giunsero puromicina e osservarono il tasso di peptidil-puromicina
O
OH
R
H
R
marcata rilasciata dal ribosoma. Poiché la puromicina si lega molto
Sito P
Sito A
rapidamente al sito A, la peptidil-transferasi è tasso-limitante nel
Figura
17.18
Posizione
dei
tRNA
nel sito A
rilascio di peptidil-puromicina, così il tasso di rilascio può essere
e nel sito P durante la reazione peptidilpreso come una misura del tasso della peptidil-transferasi. Strobel e
transferasica. Il gruppo 2’-OH del tRNA nel
colleghi separarono la peptidil-puromicina marcata, rilasciata dalle sito P è in rosso, l’azoto amminico dell’amaltre sostanze marcate, utilizzando l’elettroforesi in strato sottile, e minoacil-tRNA nel sito A è in verde, mentre
il carbonio carbonilico del peptidil-tRNA nel
determinarono la radioattività mediante phosphorimaging.
sito P è in blu. Si noti la vicinanza del grupLa Figura 17.19 mostra i risultati. Con un normale tRNA come po 2’-OH del tRNA nel sito P per attaccare
substrato, la reazione peptidil-transferasica si completava nel pri- l’azoto amminico nel sito A.
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W22 • La traduzione
Parte VI
mo intervallo di tempo (10s). Invece, con entrambi i substrati modificati
essenzialmente non avveniva alcuna reazione, anche dopo le 24 h. Dunque,
sostituendo con un atomo di idrogeno o di fluoro il gruppo 2’-OH del tRNA
nel sito P si bloccava completamente la reazione peptidil-transferasica, e ciò
suggeriva fortemente che il gruppo 2’-OH sia richiesto per la reazione. Lo
stesso comportamento si osservava con i tre substrati e l’ordinario Phe-tRNA,
piuttosto che puromicina, nel sito A, supportando ulteriormente l’importanza del gruppo 2’-OH.
Questo studio lascia però ancora irrisolto il problema del ruolo dell’ A2541
(usando la numerazione di E. coli) altamente conservata dell’rRNA 23S. Per
sondare questo problema Norbert Polacek e colleghi escogitarono un metodo
per modificare la natura, non solo della base, ma anche dello zucchero di A2451.
Quando essi rimuovevano la base adenina dall’A2451, creando un sito abasico,
avvenivano piccoli cambiamenti nell’attività peptidil-transferasica, come misurato attraverso il saggio di rilascio della fMet-puromicina. Invece, quando
rimuovevano il gruppo 2’-OH dall’A2451, riducevano l’attività di circa 10 volte. Inoltre, quando essi rimuovevano sia la base che il gruppo 2’-OH abolivano
quasi completamente l’attività. Al contrario, effettuando le stesse modificazioni
sul nucleoside adiacente, A2450, si aveva solo un modesto effetto sull’attività,
sottolineando così di nuovo la particolare importanza di A2451.
La perdita dell’attività nel ribosoma mancante del gruppo 2’-OH in posizione A2451 dell’rRNA 23S potrebbe essere dovuta alla ridotta affinità per il
tRNA nel sito P. Se così fosse, aumentando le concentrazioni di fMet-tRNA
si incrementerebbe l’attività, ma non è così. Qual è dunque il ruolo di questo
gruppo idrossile? L’evidenza appena esaminata della partecipazione del 2’-OH
⫺
fA76
⫹
time
⫺
⫹
time
19
20
21
22
23
24
⫺
dA76
⫹
time
13
14
15
16
17
18
⫺
1
2
3
4
5
6
Pmn
A76
⫹
time
7
8
9
10
11
12
tRNALys
fMet
origin
fMet-puro
fMet-Lys
fMet-Lys-puro
Figura 17.19 Attività peptidil-transferasica con tRNA modificati. Strobel e colleghi effettuarono la reazione peptidil-transferasica usando un dipeptidil-tRNA marcato nel sito
P e puromicina aggiunta al sito A. Il tRNA nel sito P conteneva una normale A76, dA76,
oppure fA76, o semplicemente fMet-tRNA senza modificazioni (-), come indicato in alto.
Essi effettuarono le reazioni per diversi intervalli di tempo (10 s, 1 min, 6 min, 1 h, e 24 h)
in presenza o in assenza (-) di puromicina, come indicato in alto. Separarono il dipeptidilpuromicina marcato (fMet-Lys-puro) mediante elettroforesi in strato sottile e sottoposero
l’elettroferogramma a phosphorimaging. Solo la normale A76 del tRNA nel sito P era capace di sostenere un’attività peptidil-transferasica rilevabile. (Source: Reprinted from Nature
Structural & Molecular Biology, vol. 11, Joshua S. Weinger, K. Mark Parnell, Silke Dorner,
Rachel Green & Scott A. Strobel, “Substrate-assisted catalysis of peptide bond formation
by the ribosome, “ Fig. 3a, p.1103, copyright 2004, reprinted by permission from Macmillan
Publishers Ltd.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
del tRNA nel sito P nella chimica della transpeptidazione è forte, ma è possibile
che anche il gruppo2’-OH dell’A2451 partecipi in questo modo. In alternativa,
quest’ultimo gruppo idrossilico potrebbe contribuire alla reazione catalitica aiutando i reagenti a posizionarsi appropriatamente nel sito attivo.
Mentre il polipeptide nascente viene assemblato, si pensa che questo esca
attraverso un tunnel della subunità ribosomale 50S. Moore, Steitz e colleghi
hanno chiarito anche alcuni aspetti su questo fenomeno. La Figura 17.20
mostra un’immagine della subunità 50S tagliata nel mezzo, attraverso la protuberanza centrale, per mettere in evidenza il tunnel di uscita del polipeptide.
Nella figura è stato inoltre marcato il sito peptidil-transferasico ed è stato
inserito un polipeptide nel tunnel. Il tunnel ha un diametro medio di 15 Å
con due restringimenti di 10 Å, larghi abbastanza da accogliere un peptide ad
α-elica, ma non abbastanza da permettere altri tipi di ripiegamenti. Un’estesa
parte del tunnel è costituita da RNA idrofilo, quindi i residui idrofobici esposti nella catena polipeptidica nascente non possono interagire con le pareti del
tunnel evitando il rallentamento del processo di uscita.
SOMMARIO La struttura cristallografica della subunità 50S è stata determinata a una risoluzione di 2,4 Å. Tale struttura ha mostrato poche proteine presenti all’interfaccia tra le subunità ribosomali, e addirittura assenza totale di proteine fino a 18 Å dal sito attivo peptidil-transferasico,
marcato con un analogo dello stato di transizione. Il gruppo 2’OH del tRNA
nel sito P è molto ben posizionato per formare un legame idrogeno con
il gruppo amminico dell’amminoacil-tRNA nel sito A, e quindi per aiutare
la reazione peptidil-transferasica. In accordo con questa ipotesi, la rimozione del gruppo idrossile elimina quasi completamente l’attività peptidiltransferasica. Allo stesso modo la rimozione del gruppo 2’-OH dell’ A2451
dell’rRNA 23S inibisce fortemente l’attività peptidil-transferasica. Anche
questo gruppo può partecipare alla catalisi attraverso un legame idrogeno,
oppure aiutando i reagenti a posizionarsi correttamente per la catalisi. Il
tunnel di uscita attraverso la subunità 50S ha un diametro tale da permettere il passaggio di un polipeptide ripiegatato ad α-elica. Le pareti del tunnel sono costituite da RNA, che grazie alla sua idrofilicità permette ai residui idrofobici esposti del polipeptide di scivolare facilmente dal tunnel.
Figura 17.20 Il tunnel di uscita del polipeptide. La subunità 50S è mostrata in modo tale
da sembrare un frutto tagliato a metà e aperto. Questa immagine evidenzia il canale che
parte dal sito attivo peptidil-transferasico (PT). Una α-elica in bianco è posta nel canale
per rappresentare un polipeptide in uscita. (Da: Ban, N., P. Nissen, J. Hansen, P.B. Moore
e T.A. Steitz, The structural basis of ribosome activity in peptide bond synthesis. Science
289 (11 Aug 2000) f. 11a, p. 927. Copyright ©AAAS.)
Ribosomi e tRNA
•
W23
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W24 • La traduzione
Parte VI
I polisomi
Nei precedenti capitoli abbiamo visto come più di una RNA polimerasi possa
trascrivere un gene, nello stesso tempo. Ciò è vero anche per i ribosomi e
l’mRNA. Infatti, più ribosomi possono tradurre lo stesso mRNA in un dato
tempo. Si ha così un poliribosoma o polisoma, come mostrato nella Figura
17.21. In questa immagine possiamo osservare 74 ribosomi che traducono
simultaneamente lo stesso mRNA. Possiamo addirittura osservare qual è la
fine del polisoma, guardando la lunghezza della catena polipeptidica nascente. Questa cresce man mano che il ribosoma si sposta dal 5’ dell’mRNA (dove
inizia la traduzione) al 3’ (dove termina la traduzione). Quindi nella figura il
5’ dell’mRNA è in basso a sinistra e il 3’ è in basso a destra.
Consideriamo il processo di formazione di un polisoma eucariotico. Il
primo ribosoma caricato sopra l’mRNA ricopre il compito più difficile nel
suo “giro pionieristico” di traduzione. L’mRNA proviene dal nucleo carico
di proteine: alcune di queste vengono perse durante i processi di splicing
e poliadenilazione, altre, legate all’mRNA, lo guidano fuori dal nucleo e
lo proteggono dalla degradazione. Ma poiché c’è appena spazio sufficiente
per l’mRNA tra le due subunità ribosomali, queste proteine debbono essere
staccate dall’mRNA quando questo si infila nel primo ribosoma. Queste
proteine sono subito dopo sostitute da altre proteine richieste per il processo
di traduzione.
Il polisoma nella Figura 17.21 è eucariotico (di un moscerino). Poiché
negli eucarioti la trascrizione e la traduzione avvengono in compartimenti
differenti i polisomi si trovano sempre nel citoplasma della cellula, indipendenti dai geni. Anche i procarioti hanno i polisomi, ma l’immagine in questi
Figura 17.21 Fotografia al microscopio elettronico di un polisoma del moscerino
Chironomus. L’estremità 5’ dell’mRNA si trova in basso a sinistra, l’mRNA è piegato in alto,
mentre in basso a destra si trova l’estremità 3’. Le macchie scure sono i ribosomi ancorati
all’mRNA. Per il fatto che molti ribosomi (74) sono legati, la struttura prende il nome di polisoma. Il polipeptide nascente si allontana dai ribosomi man mano che viene sintetizzato
e diventa più grande verso la fine dell’mRNA. Le macchie meno marcate che escono dai
ribosomi rappresentano il polipeptide nascente e non singoli amminoacidi. (Da: Francke et
al., Electron microscopic visualization of a discreet class of giant translation units in salivary glands of Chironomus tetans. EMBO Journal 1 1982, pp. 59-62. European Molecular
Biology Organization.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
organismi è più complicata, in quanto la trascrizione e la traduzione di un
dato gene e del suo mRNA avvengono contemporaneamente e nello stesso
compartimento cellulare. Quindi è possibile vedere l’mRNA nascente mentre viene sintetizzato e, nello stesso tempo, tradotto dai ribosomi. La Figura
17.22 mostra proprio questa situazione in E. coli. Possiamo vedere due segmenti del cromosoma batterico, che corrono paralleli da sinistra a destra; solo
il segmento in alto è trascritto. Possiamo vedere che la trascrizione sta avvenendo da sinistra verso destra poiché i polisomi aumentano muovendosi in
questa direzione; all’aumentare della lunghezza dell’mRNA, questo può accogliere più ribosomi. Non bisogna essere ingannati dalla differenza di scala
tra la Figura 17.21 e la 17.22: i ribosomi appaiono più piccoli e il polipeptide
nascente non è visibile in quest’ultima immagine. È da notare inoltre che le
catene orizzontali in Figura 17.22 sono costituite da DNA mentre quella in
Figura 17.21 è un mRNA. Gli mRNA si trovano in posizione grosso modo
verticale in Figura 17.22.
SOMMARIO Molti mRNA sono tradotti da più di un ribosoma contemporaneamente; si forma così una struttura, definita polisoma, in cui molti
ribosomi traducono un mRNA in tandem. Negli eucarioti i polisomi si trovano nel citoplasma, mentre nei procarioti la trascrizione di un gene e la
traduzione del suo mRNA avvengono contemporaneamente, quindi molti
polisomi si trovano associati a geni trascrizionalmente attivi.
17.2 RNA transfer
Nel 1958 Francis Crick postulò l’esistenza di una molecola adattatrice, presumibilmente di RNA, che poteva servire come un mediatore funzionale tra
una fila di nucleotidi nel DNA (in realtà di mRNA) e una fila di amminoacidi nella proteina corrispondente. Crick postulò la presenza di due o tre nu-
0,5 μm
Figura 17.22 Trascrizione e traduzione simultanea in E. coli. Due frammenti di DNA
attraversano orizzontalmente la figura. Il frammento superiore è trascritto da sinistra verso
destra. Non appena gli mRNA vengono trascritti i ribosomi iniziano la traduzione. Questo
genera i polisomi, che nella figura sono disposti perpendicolarmente al DNA. Il polipeptide
nascente non è visibile nell’immagine. La macchia più tenue, indicata dalla freccia a sinistra dell’immagine, è l’RNA polimerasi che sta iniziando a trascrivere il gene. Si possono
inoltre individuare altre macchie corrispondenti all’RNA polimerasi alla base di alcuni polisomi, dove gli mRNA si connettono al DNA. (Da: O.L. Miller, B.A. Hamkalo e C.A. Thomas
Jr., Visualization of bacterial genes in action. Science 169 (July 1970) p. 394. Copyright
©AAAS.)
•
W25
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W26 • La traduzione
Parte VI
cleotidi nella molecola adattatrice che potessero appaiarsi con i nucleotidi nei
codoni, sebbene a quel tempo nessuno conoscesse la natura di tali codoni, o
comunque, l’esistenza dell’mRNA. L’RNA transfer era stato scoperto da Paul
Zamecnik e collaboratori l’anno precendente, sebbene questi non avessero
compreso il ruolo di adattatore svolto da questa molecola.
La scoperta del tRNA
Dal 1957 Zamecnik e collaboratori hanno lavorato con un sistema di sintesi
delle proteine utilizzando estratti cellulari di ratto. Uno dei componenti del
sistema era chiamato frazione enzimatica a pH 5; questa frazione conteneva
fattori solubili che interagivano con i ribosomi nel dirigere la traduzione di
mRNA aggiunti. La maggior parte dei componenti della frazione enzimatica
a pH 5 erano proteine, ma il gruppo di Zamecnik scoprì che questa frazione
conteneva anche un piccolo RNA. L’importanza di questa scoperta risiede nel
fatto che questo RNA poteva essere accoppiato ad amminoacidi. Per dimostrare ciò, i ricercatori hanno mescolato l’RNA con gli enzimi a pH 5, ATP e
[14C]leucina. La Figura 17.23a mostra come a una maggiore somministrazione di leucina marcata si otteneva più leucina legata all’RNA, che essi separarono da proteine mediante estrazione fenolica. Inoltre, in assenza di ATP non
avveniva nessuna reazione. Oggi sappiamo che questa reazione consiste nel
caricamento di un tRNA con un amminoacido.
Zamecnik e collaboratori non solo dimostrarono che un piccolo RNA
può essere caricato con un amminoacido, ma anche che questo RNA può
donare l’amminoacido legato alla catena peptidica nascente. Eseguirono
l’esperimento aggiungendo alla reazione [14C]leucina caricata sull’RNA pH
5 con i microsomi, cioè piccole sezioni di reticolo endoplasmatico contenenti
i ribosomi. La Figura 17.23b mostra una corrispondenza quasi perfetta tra la
perdita della leucina radioattiva dall’RNA pH 5 e l’inserimento della leucina
(b)
4
3
Proteine
microsomali
3
CMP totali
Addizione della leucina
all'RNA (μmol/mg)
(a)
2
2
1
1
RNA
1
2
3
[Leucina] mM
4
5
5
10
15
20
Minuti
Figura 17.23 Scoperta del tRNA. (a) Il tRNA può essere caricato con una leucina. Zamecnik e colleghi hanno aggiunto leucina marcata alla frazione contenente tRNA e hanno
misurato il legame dell’amminoacido all’RNA in funzione della leucina aggiunta. (b) Il tRNA
carico può donare l’amminoacido alla catena polipeptidica nascente. Zamecnik e colleghi
hanno seguito la perdita di radioattività (cpm) dell’RNA (in blu) e l’acquisto di radioattività
da parte della catena polipeptidica nascente (in rosso) nei microsomi, che contengono i
ribosomi. La relazione reciproca tra queste due curve suggerisce che l’RNA doni il suo amminoacido alla proteina nascente. (Da: Held, W.A., B. Ballou, S. Mizushima e M. Nomura,
et al., Journal of Biological Chemistry 231:244 & 252, 1958. Copyright ©1958 The American
Society for Biochemistry & Molecular Biology, Bethesda, MD. Riproduzione autorizzata.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
nel peptide nascente nei microsomi. Questa rappresenta l’incorporazione della leucina dal leucil-tRNA nel polipeptide nascente sui ribosomi.
SOMMARIO L’RNA transfer è stato identificato come un piccolo RNA indipendente dai ribosomi che poteva essere caricato con un amminoacido,
e che aveva la capacità di trasferire l’amminoacido al peptide nascente.
La struttura del tRNA
Per capire come il tRNA svolge la sua funzione è necessario conoscere la sua
struttura molecolare, e considerando le piccole dimensioni, la sua struttura
molecolare è sorprendentemente complessa. Come le proteine, anche i tRNA
possiedono una struttura primaria, secondaria e terziaria. La struttura primaria è costituita dalla sequenza lineare delle basi che lo compongono; la struttura secondaria è descritta dagli appaiamenti di coppie di basi preferenziali
che le regioni della molecola assumono, a dare origine ad anse e steli (stemloop); la struttura terziaria è costituita dal ripiegamento tridimensionale della
molecola. In questo paragrafo analizzeremo la struttura dei tRNA e la relazione di questa con le loro funzioni.
Nel 1965 Robert Holley e colleghi determinarono la prima sequenza nucleotidica di un acido nucleico naturale, il tRNA dell’alanina in lievito. La
sequenza primaria del tRNA suggeriva almeno tre interessanti strutture secondarie, inclusa una a forma di trifoglio. A partire dal 1969 sono state identificate le sequenze di 14 tRNA ed è quindi diventato chiaro che, a dispetto
delle diverse sequenze nucleotidiche, tutti potevano assumere essenzialmente
la stessa struttura secondaria a “trifoglio” come illustrato in Figura 17.24a.
Mentre studiamo la struttura dei tRNA dobbiamo comunque ricordare che
la loro struttura terziaria non assume del tutto un ripiegamento a trifoglio;
la struttura a trifoglio, infatti, descrive il semplice profilo di appaiamento di
basi della molecola.
La struttura a trifoglio possiede quattro principali regioni di appaiamento
di basi, chiamati bracci (o steli) che definiscono le quattro principali regioni
delle molecola (Figura 17.24b). Il primo visibile in alto nel diagramma è il
braccio accettore, che include appaiate tra loro le due estremità della molecola. L’estremità 3’ porta la sequenza di basi conservata CCA, che protrude
rispetto all’estremità 5’. A sinistra possiamo notare l’ansa D, così chiamata
per la costante presenza della base modificata diidrouracile. In basso è presente l’ansa dell’anticodone, che contiene l’importante sequenza anticodone all’apice dell’ansa. Come abbiamo appreso nel Capitolo 3, l’appaiamento
dell’anticodone con il codone permette la decodificazione dell’mRNA. A
destra possiamo notare l’ansa T che prende nome dalla presenza di tre basi
conservate: TΨC. La Ψ indica il nucleoside modificato pseudouridina, simile all’uridina ma con la base legata al ribosio attraverso il carbonio-5 e non
all’azoto-1. La regione tra il braccio dell’anticodone e il braccio T in Figura
17.24 è chiamata ansa variabile perché cambia di lunghezza da 4 a 13 nucleotidi a seconda del tRNA; alcune delle anse variabili più lunghe possiedono
regioni di complementarità capaci di formare anch’esse steli.
Oltre al diidrouracile e alla pseudouridina i tRNA posseggono molti nucleosidi modificati. Molte delle modificazioni sono semplici mutilazioni. Altre sono invece molto elaborate, come la conversione della guanosina in un
Ribosomi e tRNA
•
W27
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W28 • La traduzione
Parte VI
D
G
G
G20
A
m22G
C —
C —
A —
G 30—
A —
Cm
U
Gm
G
—
—
C
—
G A G C25
—
D
G15
5
pG —
C —
G —
G
A5 —
U —
U —
U
A
A
C U C m2G10
—
—
—
—
—
A—OH 3
C75
C
A
C
G
C70
U
U
A
A
C60 U m A
G65 A C A C
G
C
m5C U50 G U G
T Ψ55
(a)
(b)
5
pG
A—OH 3
C
C
Braccio accettore
Braccio T
Braccio D
Braccio variabile
U m7G
A G45
G
U
m5C40
Ψ
A
Braccio anticodone
Anticodone
Y
A35
A
Figura 17.24 Due schemi della struttura a trifoglio del tRNA. (a) Sequenza di basi del t-RNAphe di lievito, che mostra la
forma a trifoglio. I nucleotidi conservati sono in rosso. I nucleotidi che sono sempre purine e/o sempre pirimidine sono in blu.
(b) Struttura a trifoglio del t-RNAphe. In alto è presente il braccio accettore (in rosso), dove l’amminoacido è legato al 3’ terminale dell’adenosina. A sinistra è mostrata l’ansa diidro U (ansa D, in blu), contenente almeno una base diidrouracile. In basso è
mostrata l’ansa anticodone (in verde), che contiene l’anticodone. L’ansa T (a destra in grigio), contiene la sequenza conservata
TΨC. Ogni ansa è definita dall’appaiamento di basi dello stelo e mostrata con lo stesso colore di quest’ultimo. (Da: (a) Kim,
S.H., F.L. Suddath, G.J. Quigley, A. McPherson, J.L. Sussman, A.H.J. Wang, N.C. Seeman e A. Rich, Three-dimensional tertiary
structure of yeast phenylalanine transfer RNA, Science 185: 435, 1974. Copyright ©1974 American Association for the Advancement of Science, Washington, DC. Riproduzione autorizzata.)
nucleoside chiamato Yosina una base composta da tre anelli chiamata base
Y (Figura 17.25). Alcune modificazioni dei tRNA sono più generiche; per
esempio, virtualmente tutti i tRNA hanno una pseudouridina nella stessa posizione nel braccio T, e molti tRNA hanno nucleosidi ipermodificati come la
Yosina, che è prossimale all’anticodone. Altre modificazioni sono specifiche
per alcuni tRNA. La Figura 17.25 illustra alcune delle comuni modificazioni
dei tRNA.
La modificazione di alcuni nucleosidi del tRNA porta a una domanda:
I tRNA nascono con le basi modificate, o le modificazioni avvengono al
termine della trascrizione? La risposta è che i tRNA nascono, analogamente
agli altri tipi di RNA, composti dalle quattro basi canoniche. Al termine della
trascrizione un sistema di enzimi multipli modifica le basi. Che effetto hanno
le modificazioni delle basi, se ne hanno, sulla funzione dei tRNA? Almeno
due tRNA sono stati sintetizzati in vitro, ed essendo composti dalle quattro
basi canoniche non sono stati capaci di legare gli amminoacidi. Quindi, almeno in questo caso, tRNA con assenza di modificazioni non sono funzionali. Questi esperimenti hanno quindi evidenziato come l’insieme delle varie
modificazioni sia critica, mentre una singola modificazione ha probabilmente
uno scarso effetto nell’efficienza di caricamento del tRNA.
Negli anni ’70 Alexander Rich e colleghi hanno adottato tecniche di diffrazione a raggi X per comprendere la struttura terziaria dei tRNA. Poiché
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
S
O
H
H
H
H
HN
O
N
HN
N
O
Ribosio
Diidrouridina
(D)
NH 2
NH 2
CH 3
O
N
Ribosio
4-Tiouridina
(s4U)
N
O
N
Ribosio
3-Metilcitidina
(m3C)
O
CH 3
N
O
CH 3
HN—CH3
Ribosio
HN
O
N
N
O
N
H
Pseudo-uridina
(ψ)
Ribosio
ribotimidina
(rT)
HN—CH2—CH
N
N
N
N
N
Ribosio
N6 Metiladenosina
(m6A)
C
N
HN
COOCH3
HC
COOCH3
CH2
N
N
Ribosio
5-Metilcitidina
(m5C)
O
HN
N
HN
W29
•
Ribosio
Inosina
(I)
H 3C
CH3
CH3
CH2
O
N
N
N
CH3
Yosina
(Y)
N
N
N
Ribosio
Ribosio
N6 Isopenteniladenosina
Figura 17.25 Alcuni nucleosidi modificati nei tRNA. Il rosso indica la variazione di uno dei quattro nucleosidi canonici. L’inosina è un caso speciale; infatti è un precursore sia dell’adenosina che della guanosina.
tutti i tRNA possiedono essenzialmente la stessa struttura secondaria, rappresentata dal modello a trifoglio; non è sorprendente che questi abbiano anche
una struttura terziaria simile. In Figura 17.26 è mostrata la struttura terziaria
del tRNAphe di lievito definita ripiegamento a L. La principale caratteristica
di questo ripiegamento è la massimizzazione della lunghezza delle regioni di
complementarietà degli steli, dovuta all’accoppiamento di due steli, a formare
una lunga regione di appaiamento. Una di queste due regioni giace orizzontalmente, all’estremità superiore della molecola, e comprende il braccio accettore e il braccio T. L’altra regione costituisce l’asse verticale della molecola e
include il braccio D e il braccio anticodone. Anche se i due segmenti di ogni
braccio non sono allineati perfettamente, e tendono a piegarsi leggermente,
l’allineamento permette alle coppie di basi di impilarsi l’un l’altra, conferendo quindi stabilità alla struttura terziaria. Gli steli sono costituiti da doppie
eliche di RNA. Come abbiamo visto nel Capitolo 2, le doppie eliche di RNA
assumono una forma ad A-elica con circa 11 coppie di basi per giro d’elica e
la cristallografia a raggi X ha confermato questa predizione.
Braccio T
54
64
1
56
Braccio
accettore
Braccio
accettore
Braccio T
4
72
1
69
7
2
4
12
2
Braccio T
44
Braccio D
26
38
(a)
1
Braccio D
Braccio D
20
Braccio
accettore
3
Anticodone
32
(b)
Braccio
anticodone
3
(c)
Braccio
anticodone
Braccio
anticodone
Figura 17.26 Struttura tridimensionale del tRNA. (a) Una proiezione planare della struttura del tRNAphe di lievito. Le varie
regioni della molecola hanno il codice di colori come in (b) e (c). (b) La familiare struttura a trifoglio dei tRNA con il codice dei
colori come in (a). Le frecce indicano i ripiegamenti assunti dalla struttura a trifoglio, per raggiungere la forma approssimativa
del vero tRNA, illustrata in (c). (Source: adapted from Quigley, G.J. and A. Rich, structural domains of transfer RNA molecules,
Science 194:197 Fig. 1b,1976.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W30 • La traduzione
Parte VI
La Figura 17.27 è un’immagine stereo della molecola di tRNAphe di lievito.
Le regioni di appaiamento sono particolarmente semplici da identificare in
tre dimensioni, ma è possibile comunque visualizzarle anche in due dimensioni, nella regione del braccio T-braccio accettore perché sono posizionate
perpendicolarmente al piano della pagina, in questo modo appaiono quasi
come linee parallele.
Come abbiamo visto, un tRNA è stabilizzato principalmente da interazioni
secondarie che si formano tra regioni di appaiamento di basi, ma è anche stabilizzato da dozzine di interazioni terziarie tra le varie regioni. Queste includono
interazioni base-base, base-scheletro e scheletro-scheletro. Molte di queste avvengono tra basi conservate o semi-conservate (le basi semi-conservate identificano basi che sono sempre purine o pirimidine). Poiché queste interazioni
permettono al tRNA di ripiegarsi in una struttura caratteristica, sembra logico
che queste basi tendano a essere conservate; ogni variazione infatti tenderebbe a influenzare la forma del tRNA, compromettendone la funzionalità. Una
sola interazione base-base coinvolge un canonico appaiamento Watson-Crick
(G19-C56). Tutte le altre interazioni sono non canoniche. Consideriamo per
esempio la coppia di basi G15-C48, che connette l’ansa D all’ansa variabile.
Questa non può essere un appaiamento Watson-Crick perché i due filamenti sono paralleli e non antiparalleli. Chiameremo questo tipo di interazione
appaiamento in trans. Esistono, inoltre, molti esempi di interazioni che avvengono tra una base singola e altre due basi. Un’interazione di questo tipo
coinvolge U8, A14 e A21. Ora che le interazioni terziarie sono state descritte,
riesaminando la Figura 17.26a è possibile vederla in forma più realistica. Da
notare per esempio, le interazioni tra la base 18 e la 55 e tra la base 19 e la 56.
In prima analisi sembrano basi che si appaiano nel braccio T; studiandole più
attentamente è possibile notare come queste connettano l’ansa T all’ansa D.
Un aspetto interessante della struttura terziaria dei tRNA è la disposizione
dell’anticodone. La Figura 17.27 mostra che le basi dell’anticodone sono impilate e proiettate verso destra, dalla parte opposta dello scheletro del tRNA.
Questo le mette in posizione per interagire con le basi del codone dell’mRNA. Infatti lo scheletro dell’anticodone è avvolto in una disposizione a elica
parziale, che presumibilmente facilita l’appaiamento con il codone corrispondente (vedi Figura 17.2).
3 del braccio
accettore
Anticodone
Figura 17.27 Immagine stereo del tRNA. Per osservare la molecola in tre dimensioni,
usate un visore tridimensionale, oppure forzate le due immagini a fondersi, rilassando gli
occhi come se dovessero mettere a fuoco qualcosa a distanza (tecnica dell’occhio magico), o incrociando leggermente gli occhi. Occorre un po’ di tempo affinché l’effetto tridimensionale abbia luogo. (Da: Quigley, G.J e A. Rich, Structural domains of transfer RNA
molecules. Science 194 (19 Nov 1976) f. 2, p. 798. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
•
W31
SOMMARIO Tutti i tRNA possiedono una comune struttura secondaria
a forma di trifoglio. Sono strutturati in 4 steli formati da appaiamenti di
basi, che definiscono tre regioni stelo-ansa (l’ansa D, l’ansa dell’anticdone e l’ansa T), e un braccio accettore costituito da uno stelo dove sono
aggiunti gli amminoacidi in fase di caricamento. I tRNA mostrano, inoltre,
una comune struttura terziaria, che assomiglia a una L invertita. Questo
ripiegamento stabilizza l’accoppiamento di basi allineando l’ansa D con
l’ansa anticodone e tra l’ansa T e il braccio accettore. L’anticodone del
tRNA protrude dall’ansa dell’anticodone ed è avvolto in modo da facilitare
l’appaiamento con il corrispondente codone sull’mRNA.
Riconoscimento dei tRNA da parte delle amminoacil-tRNA
sintetasi: il Secondo Codice Genetico
Nel 1962, Fritz Lipmann, Seymour Benzer, Günter von Ehrenstein e colleghi
dimostrarono che il ribosoma riconosce il tRNA e non l’amminoacido, in un
amminoacil-tRNA. L’esperimento è stato eseguito riducendo un cisteinil-tRNACys tramite nikel Raney ad alanil-tRNACys come mostrato in Figura 17.28.
(Notare la nomenclatura: nel cisteinil-tRNACys [Cys- tRNACys] la prima Cys
denota l’effettivo amminoacido caricato sul tRNA. La seconda Cys [in apice]
indica quale amminoacido dovrebbe essere attaccato al tRNA. Quindi alaniltRNACys è il tRNA che dovrebbe legare la cisteina, ma che in questo caso lega
l’alanina.) Hanno quindi introdotto questo tRNA alterato in un sistema di
traduzione in vitro, utilizzando un mRNA sintetico costituito da una sequenza casuale di U e G, in proporzione di 5 : 1. Questo mRNA possedeva molti
codoni UGU, i quali codificano la cisteina, quindi incorporano la cisteina
nella catena polipeptidica. Non possono inoltre causare l’incorporazione
dell’alanina perché il codone corrispondente a questo amminoacido è GCN,
dove N indica qualsiasi base e il polimero UG non contiene C. Comunque,
in questo caso l’alanina veniva incorporata perché legata al tRNACys. Questo
dimostra che i ribosomi non discriminano l’amminoacido attaccato al tRNA,
ma riconoscono solo la parte tRNA dell’amminoacil-tRNA.
Questo esperimento ha sottolineato l’importanza del corretto caricamento sul tRNA dell’amminoacido da parte dell’amminoacil-tRNA sintetasi.
Visto che i ribosomi riconoscono unicamente la parte tRNA dell’amminoacil-tRNA, un errato caricamento dell’amminoacido sul tRNA da parte della
sintetasi porterà a una proteina con un amminoacido errato. Questo potrebbe
Cys
Ala
nickel
Raney
ACG
UGU
Inserisce
Cys
ACG
UGU
Inserisce
Ala
Figura 17.28 Il ribosoma riconosce il tRNA e non l’amminoacido caricato dell’amminoacil-tRNA. Lipmann, Ehrenstein,
Benzer e colleghi partirono da un cisteinil-tRNACys, che inserisce
una cisteina (Cys, in blu) nella catena polipeptidica nascente,
come mostrato a sinistra della figura. Trattarono successivamente questo amminoacil-tRNA con nickel Raney, che riduce la
cisteina ad alanina (Ala, in rosso), ma non ha effetti sul tRNA.
Questo alanil-tRNACys inserisce un’alanina in una catena polipeptidica, in una posizione normalmente occupata da una cisteina,
come mostrato a destra. Quindi è importante la natura del tRNA
e non il tipo di amminoacido caricato, in quanto è la sequenza nucleotidica del suo anticodone che riconosce il codone
dell’mRNA.
Biologia molecolare 2/ed
Robert F. Weaver
W32 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
essere molto dannoso in quanto una proteina con sequenza amminoacidica
errata potrebbe essere non funzionale. Quindi non è sorprendente osservare
un’alta specificità delle amminocil-tRNA sintetasi nel legare tRNA e amminoacidi. Questo porta a una interessante questione relativa alla struttura dei
tRNA: vista la struttura secondaria e terziaria conservata in tutti i tRNA,
quale sequenza di basi del tRNA riconosce la sintetasi, per selezionare un
tRNA tra più di 20 diversi? Questa sequenza di nucleotidi è stata nominata “secondo codice genetico”, per sottolinearne l’impotanza. La domanda è
complicata anche dall’esistenza di specie isoaccettori di tRNA che possono
essere caricate con lo stesso amminoacido dalla stessa sintetasi, avendo comunque sequenze differenti e spesso differenti anticodoni.
Se volessimo indovinare quali sequenze vengono riconosciute dalla sintetasi sul tRNA, dovremmo prendere in considerazione in particolar modo
due siti. Il primo, il braccio accettore, sembra la scelta più logica, in quanto
è la regione dove l’amminoacido viene legato, quindi la regione più vicina al
sito attivo della sintetasi. Visto che l’enzima ha presumibilmente uno stretto contatto con il braccio accettore, potrebbe identificare delle sequenze in
questa regione tra i tRNA che ne identifichino il tipo. Comunque almeno le
tre basi CCA sono irrilevanti in questo tipo di riconoscimento, perché presenti in tutti i tRNA. Secondo, l’anticodone è una scelta ragionevole, perché
differente nei diversi tRNA, ed è di correlazione diretta con l’amminoacido
che deve essere caricato sul tRNA. Vedremo che, in molti casi, entrambe le
ipotesi sono corrette; inoltre, altre aree in alcuni tRNA svolgono un ruolo nel
riconoscimento da parte dell’amminoacil-tRNA sintetasi.
Il braccio accettore Nel 1972 Dieter Soll e i suoi collaboratori osservarono
in molti tRNA, un profilo particolare nella quarta base a partire dall’estremità 3’ in posizione 73. Questa base tende a essere la stessa in molti tRNA
specifici per una certa classe di amminoacidi. Per esempio, tutti gli amminoacidi idrofobici sono virtualmente accoppiati a tRNA con la base A nella
posizione 73, indifferentemente dalla specie in esame. Tuttavia una singola
base non può fornire una variazione strutturale adeguata per permettere un
caricamento specifico nelle 20 differenti classi di tRNA. Per lo più, potrebbe
svolgere un ruolo in una discriminazione approssimativa.
Bruce Roe e Bernard Dudok hanno utilizzato un altro tipo di approccio;
hanno esaminato, in molte specie, la sequenza di basi di tutti i tRNA che
potevano essere caricati da una singola sintetasi. Questi includevano alcuni tRNA che erano stati caricati con l’amminoacido errato, in un processo
che viene definito “caricamento eterologo errato”. Questo termine si riferisce
all’abilità di una sintetasi di una specie di caricare un tRNA non corretto di
un’altra specie. Tuttavia il caricamento eterologo è poco efficiente e richiede una maggiore concentrazione di enzima rispetto alle normali condizioni.
Per esempio la fenilalanil-tRNA sintetasi di lievito (PheRS) può caricare il
tRNAphe di E. coli, di lievito e di germe del grano, e può anche caricare il
tRNAVal di E. coli con la fenilalanina.
Dato che tutti questi tRNA possono essere caricati dalla stessa sintetasi,
potrebbero avere degli elementi in comune che informano l’enzima su quale
tRNA devono caricare. Così Roe e Dudock hanno confrontato le sequenze
di questi tRNA cercando degli elementi in comune tra loro, ma non comuni
a tutti i tRNA. Sono stati evidenziati due elementi caratteristici: la base 73 e
nove nucleotidi del braccio D.
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Nel 1973 J.D. Smith e Julio Celis studiarono un tRNA soppressore che
inseriva Gln al posto di Tyr. In altre parole il tRNA soppressore selvatico era
caricato dalla GlnRS, ma alcuni cambiamenti nella sequenza causavano il
suo caricamento con la TyrRS. L’unica differenza tra il tRNA mutante e il
selvatico era un cambiamento nella base 73 da G ad A.
Nel 1988, Ya-Ming Hou e Paul Schimmel hanno usato un approccio genetico per dimostrare l’importanza, nel caricamento specifico, di una singola
coppia di basi nel braccio accettore. Utilizzando un tRNA Ala con l’anticodone
mutato a 5’-CUA-3’ questo veniva trasformato in un soppressore ambra in
grado di inserire un’alanina in risposta al codone ambra UAG. Successivamente hanno osservato le mutazioni nel tRNA che cambiavano la specificità
di caricamento. Hanno quindi costruito un gene trpA con una mutazione
ambra nel codone 10. Questa mutazione poteva essere soppressa unicamente
da un tRNA capace di inserire un’alanina (o una glicina) in risposta al codone ambra. Tutti gli altri amminoacidi in posizione 10 producevano una
proteina inattiva. Successivamente hanno selezionato i loro mutanti in un
terreno mancante del triptofano. Se il mutante poteva sopprimere la mutazione ambra nel gene trpA, probabilmente possedeva un tRNA soppressore che
poteva essere caricato con alanina. In alternativa il tRNA soppressore sarebbe
stato probabilmente caricato con un altro amminoacido. I ricercatori hanno
osservato che le cellule in grado di crescere in assenza di triptofano possedevano una G in posizione 3 del tRNA soppressore e una U in posizione 70; in
questo modo potrebbe formarsi un appaiamento vacillante tra G3-U70 nel
braccio accettore a tre basi dalla fine dello stelo.
Questo esperimento suggerisce che l’appaiamento G3-U70 sia fondamentale nel caricamento operato da AlaRS. Se questo è vero, utilizzando un altro
tRNA soppressore in grado di inserire un amminoacido differente, e cambiando la posizione delle basi 3 e 70 rispettivamente in G e U, sarebbe possibile convertire la specificità di caricamento del tRNA soppressore ad Alanina.
Sono stati quindi prodotti due tRNA soppressori: tRNACys/CUA e tRNA Phe/
CUA
, dove la designazione CUA si riferisce alla sequenza dell’anticodone, che
riconosce il codone ambra UAG. Entrambi i tRNA possedevano l’appaiamento C3-G70 nel loro braccio accettore. Quando Hou e Schimmel hanno
cambiato questo appaiamento in G3-U70, hanno convertito i due tRNA in
tRNA Ala/CUA, come suggerito dalla loro abilità di sopprimere una mutazione
ambra nel codone 10 del gene trpA.
Questi tRNA soppressori ambra alterati inseriscono davvero un’alanina
nella proteina TrpA? Il sequenziamento amminoacidico ha rivelato che ciò
effettivamente avviene; inoltre questi tRNA alterati possono essere caricati
con un’alanina in vitro. Quindi se questi due tRNA differiscono dal tRNAAla/CUA
naturale, rispettivamente nelle basi 38 e 31, cambiando soltanto una
coppia di basi da C-G a G-U si cambia la specificità di caricamento da Cys
o Phe ad Ala.
Nel 1989, Christopher Francklyn e Schimmel presentarono un’ulteriore evidenza che implicava il braccio accettore, e in particolare l’appaiamento
G3-U70, nella specificità di caricamento dell’AlaRS. Essi mostrarono che una
“minielica” sintetica di 35-nt, che assomigliava alla parte superiore della forma
a L del tRNA Ala e che includeva il braccio accettore e l’ansa TΨC, poteva essere
efficientemente caricata con alanina. Infatti, finché l’appaiamento G3-U70 era
presente, il caricamento con alanina avveniva, anche quando molte altre basi
erano state modificate. È interessante notare che l’Ala-minielica si lega al sito
Ribosomi e tRNA
•
W33
Biologia molecolare 2/ed
Robert F. Weaver
W34 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
P del ribosoma, e partecipa come l’intero Ala-tRNA Ala alla reazione peptidiltransferasica con puromicina. Queste osservazioni conducono all’ipotesi che
la parte superiore della molecola di tRNA si sia evoluta prima, e potrebbe aver
partecipato, insieme a un antenato dell’rRNA 23S, a una versione grezza della
sintesi proteica in un “mondo a RNA” prima della comparsa dei ribosomi.
SOMMARIO Esperimenti biochimici e genetici hanno messo in luce l’importanza del braccio accettore nel riconscimento di un tRNA da parte della specifica amminoacil-tRNA sintetasi. In alcuni casi il cambiamento di una singola
base nel braccio accettore può cambiare la specificità di caricamento.
L’anticodone Nel 1973 LaDonne Schulman fu pioniera di una tecnica in cui
trattò il tRNAf Met con il bisolfito in grado di convertire le citosine in uracile.
Schulman e colleghi scoprirono che molte di queste alterazioni delle basi non
avevano effetto, mentre alcune mutazioni abolivano la capacità del tRNA di
essere caricato con la metionina. Uno di questi cambiamenti consisteva nella
mutazione C→U nella base 73; un’altra era la mutazione C→U nell’anticodone. Fino ad allora Schulman e colleghi avevano raccolto un gran numero di
evidenze che indicavano l’importanza dell’anticodone nella specificità di caricamento.
Nel 1983 Schulman e Heike Pelka svilupparono un metodo per variare in
modo specifico una o più basi alla volta nell’anticodone del tRNA iniziatore,
tRNA f Met. I ricercatori hanno prima tagliato il tRNA selvatico in due con
una digestione parziale a opera dell’RNasi pancreatica. Ciò rimuove l’anticodone dal frammento 5’
del tRNA e inoltre rimuove almeno due nucleotidi
Tabella 17.1 Tasso di amminoacilazione
iniziale dei derivati tRNAfMet
dalla sequenza terminale CCA del frammento 3’.
Hanno quindi utilizzato l’RNA ligasi di T4 per
Mol Met-tRNA/mol
Tasso
legare un piccolo oligonucleotide, con una o più
Met-tRNA sintetasi
relativo,
basi alterate, al frammento 5’ in modo tale da potRNA*
per min
CAU/altri
ter sostituire l’anticodone eliminato; hanno quindi
28,45
0,8
tRNAfMet
ligato le due parti del tRNA e inserito la sequenza
tRNAfMet (gel)†
22,80
1
terminale CA mediante tRNA-nucleotidiltransfeCAU
22,15
1
rasi. Infine, hanno analizzato i tRNA con il codoCAUA
1,59
14
ne alterato in una reazione di caricamento in vitro.
CCU
4,0 ⋅ 10 –1
55
850
CUU
2,6 ⋅ 10 –2
La Tabella 17.1 mostra come la variazione di una
CUA
2,0 ⋅ 10 –2
1100
base dell’anticodone del tRNA f Met sia sufficiente
–2
CAG
1,7 ⋅ 10
1300
per diminuire il tasso di caricamento con Met di
CAC
1,2 ⋅ 10 –3
18500
CA
0,5 ⋅ 10 –3
44000
almeno un fattore 105. La prima base nell’anticoC
<10 –4
>105
done (la posizione vacillante) risultò la più sensiACU
<10 –4
>105
bile; cambiando questa base si osserva infatti un
UAU
<10 –4
>105
AAU
<10 –4
>105
drastico effetto sul caricamento. Quindi l’anticoGAU
<10 –4
>105
done sembra essere necessario per il caricamento
del tRNA in vitro.
* Sono indicati gli oligonucleotidi inseriti nel braccio dell’anticodone
del derivato tRNA .
Nel 1991 Schulman e Leo Pallanck hanno rafCampione di controllo isolato da gel di poliacrilammide denaturante
forzato
quanto osservato in vitro con esperimenti
derivato sintetizzato.
in parallelo con il tRNA
Da: Schulman, L.H. e H. Pelka, Anticodon loop size and sequence
in vivo sull’alterazione degli anticodoni. Hanno
requirements for recognition of formylmethionine tRNA by methionylmodificato l’anticodone del tRNA f Met e analizzatRNA synthetase, Proceedings of the National Academy of Sciences
80: 6755-59, November 1993. Riproduzione autorizzata.
to l’abilità di questo tRNA nell’essere caricato in
Met
f
†
Met
f
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
•
W35
modo errato con un amminoacido corrispondente al nuovo anticodone. Hanno analizzato il caricamento errato utilizzando un gene reporter codificante per la deidrofolato reduttasi (DHFR), enzima semplice da isolare con un
elevato grado di purezza. Qui è riportato un esempio di come l’esperimento
è stato svolto: è stato alterato il codone del tRNA f Met da CAU a GAU, un anticodone per l’isoleucina (Ile). Quindi hanno trasferito il gene mutato in cellule
di E. coli insieme al gene DHFR con il codone d’inizio mutato in AUC.
Di norma, il codone AUC non è efficiente come codone d’inizio, ma lo è
in presenza di tRNA f Met con un codone complementare (vedi Tabella 17.2). La
sequenza risultante della proteina DHFR mostrò che l’amminoacido iniziale
era principalmente un’isoleucina. Tuttavia in alcuni casi il primo amminoacido era una metionina; questo dimostra che il tRNA f Met selvatico endogeno
può riconoscere il codone AUC come codone d’inzio.
Pallanck e Schulman utilizzarono la stessa procedura per cambiare l’anticodone del tRNA f Met in GUC (Valina, Val) o in UUC (Fenilalanina, Phe). In
ciascun caso generarono una variazione nel codone d’inizio del gene DHFR
per renderlo complementare al tRNA iniziatore modificato. La Tabella 17.2
mostra che in entrambi i casi il gene funzionava meglio in presenza del tRNA fMet
complementare. Significativamente, la tabella mostra anche che la natura
dell’amminoacido iniziale può cambiare con l’alterazione dell’anticodone del
tRNA. Infatti con il tRNA f Met, che presentava l’anticodone della valina, questo amminoacido era il solo presente all’N-terminale della proteina DHFR.
Questo significa che il cambiamento dell’anticodone del tRNA f Met da CAU
a GAC alterava la specificità di cambiamento di questo tRNA da metionina
a valina. In questo caso l’anticodone sembra essere un fattore determinante
nella specificità di caricamento del tRNA.
È da notare, inoltre, che il cambiamento dell’anticodone del tRNA f Met riduce sempre l’efficienza di sintesi. Infatti le alterazioni elencate in basso nella
Tabella 17.2 mostrano delle molecole di tRNA f Met la cui efficienza è troppo
bassa per essere analizzata anche in presenza del codone d’inizio complementare. Quindi alcune amminoacil-tRNA sintetasi possono caricare dei tRNA
non corretti con anticodoni modificati mentre altre non possono. Questi ultimi enzimi sembrano richiedere più elementi del semplice anticodone.
Tabella 10.2
Codone
iniziatore
5’-AUG-3’
AUC
GUC
UUC
UGC
AAC
ACC
UUA
GGC
GAC
Sintesi di DHFR da codoni di inizio Metionina e Nonmetionina, in presenza
o assenza dell’anticodone tRNAfMet complementare
Unità DHFR
Anticodone
+ tRNAfMet
tRNAfMet
complementare
5’-CAU-3’
GAU
GAC
GAA
GCA
GUU
GGU
UAA
GCC
GUC
1085 ± 94
298 ± 26
201 ± 3
61 ± 11
5,0 ± 0,3
4,1 ± 0,1
3,8 ± 0,4
3,4 ± 0,3
2,8 ± 0,2
0,7 ± 0,3
– tRNAfMet
complementare
Efficienza
relativa per
AUG (%)
1109 ± 133
104 ± 10
2,7 ± 0,1
2,0 ± 0,5
0,5 ± 0,08
0,5 ± 0,04
0,5 ± 0,05
1,2 ± 0,2
0,5 ± 0,04
0,4 ± 0,03
100
23
18
4,3
0,4
0,3
0,3
0,2
0,2
0,03
amminoacidi
NH2-terminali (%)
Met (100)
Ile (84), Met (16)
Val (100)
Phe (76), Met (21),Ile (3)
—
—
—
—
—
—
Da: Pallanck, L. e L.H. Schulman, Anticodon-dependent aminoacylation of a noncognate tRNA with isoleucine, valine e phenylalanine in vivo,
Proceedings of the National Academy of Sciences 88: 3872-76, May 1991. Riproduzione autorizzata.
Biologia molecolare 2/ed
Robert F. Weaver
W36 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
SOMMARIO Esperimenti biochimici e genetici hanno mostrato che sia
l’anticodone che il braccio accettore sono elementi importanti nella specificità di caricamento. In alcuni casi l’anticodone può essere il determinante
assoluto della specificità.
Strutture dei complessi sintetasi-tRNA Studi di cristallografia a raggi X
dei complessi tra tRNA, e le loro specifiche tRNA sintetasi, hanno mostrato che sia il braccio accettore che l’anticodone hanno dei siti di ancoraggio
sulle sintetasi. Questi studi sottolineano l’importanza del braccio accettore e
dell’anticodone nel riconoscimento della sintetasi. Nel 1989 Dieter Söll, Thomas Steitz e collaboratori utilizzarono la cristallografia a raggi X per determinare la prima struttura tridimensionale di un’amminoacil-tRNA sintetasi
(E. coli GlnRS) legata al suo tRNA specifico. La Figura 17.29 mostra questa
struttura. Nelle parte alta della figura possiamo osservare un profondo solco
dell’enzima, che accoglie il braccio accettore includendo la base 73 e le basi
accoppiate 3-70. In basso a sinistra si può notare un solco minore all’interno
dell’enzima che accoglie l’anticodone del tRNA. Questo potrebbe essere necessario per un riconoscimento specifico dell’anticodone dalla sintetasi. Inoltre si può notare che la maggior parte della regione a sinistra dell’enzima è in
stretto contatto con l’interno della struttura a L del tRNA che include l’ansa
D e il solco minore del braccio accettore.
Circa la metà delle sintetasi, inclusa GlnRS, fanno parte della classe
I. Queste presentano una struttura simile, inoltre la prima reazione svolta
consiste nell’amminoacilazione del gruppo 2’−OH dell’adenosina terminale
del tRNA. Il resto delle sintetasi appartengono alla classe II; anche queste
Figura 17.29 Immagine tridimensionale del complesso
glutamil-tRNA sintetasi con
tRNA e ATP. La sintetasi è mostrata in blu, il tRNA in giallo e
marrone e l’ATP in verde. Notare
le tre aree di contatto tra enzima
e tRNA: (1) il profondo solco in
alto che lega il braccio accettore
del tRNA e l’ATP; (2) la tasca più
piccola in basso a destra, dove si
inserisce l’anticodone del tRNA;
(3) l’area tra i due solchi, in cui
l’enzima contatta un’estesa regione della porzione interna della
struttura a L del tRNA. (Cortesia
di T.A. Steitz; da: Rould, Perona,
Vogt e Steitz, Science 246 (1 Dec
1989) cover. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
sono strutturalmente simili tra loro, ma abbastanza differenti dalle sintetasi
di classe I; inoltre l’amminoacilazione avviene sul gruppo 3’−OH dei loro
tRNA. Nel 1991 D. Moras e collaboratori ottennero la struttura cristallografica di un membro di classe II, l’AspRS di lievito, con il tRNA Asp. La
Figura 17.30 mette a confronto le strutture tridimensionali dei complessi
sintetasi-tRNA di classe I e II. Si possono notare molte differenze. In primo
luogo, sebbene la sintetasi contatti il tRNA all’interno della sua struttura
a L, questo avviene dalla parte opposta del tRNA, cioè includendo l’ansa
variabile e il solco maggiore del braccio accettore. Anche il braccio accettore stesso, compresi i nucleotidi terminali CCA, è in una conformazione
a elica canonica. Nelle sintetasi di classe I invece, il primo appaiamento di
basi al 3’ della molecola è rotto, cosa che induce una struttura a forcina. La
struttura cristallografica è concorde con le conclusioni principali apportate
dai dati genetici e biochimici sull’interazione delle sintetasi con i tRNA: sia
l’anticodone che il braccio accettore sono in stretto contatto con l’enzima;
inoltre il posizionamento di questi elementi determina la specificità delle
interazioni tra enzima e tRNA.
SOMMARIO La cristallografia ai raggi X ha mostrato come le interazioni
sintetasi-tRNA differiscano tra le due classi di amminoacil-tRNA sintetasi. Le
sintetasi di classe I presentano delle tasche per il braccio accettore e l’anticodone dei loro tRNA specifici e interagiscono con i tRNA dalla parte dell’ansa
D e del solco minore del braccio accettore. Anche le sintetasi di classe II
presentano delle tasche per il braccio accettore e l’anticodone, ma interagiscono con i loro tRNA dal lato opposto rispetto alle sintetasi di classe I, che
include l’ansa variabile e il solco maggiore del braccio accettore.
Correzione delle bozze e controllo dell’amminoacil-tRNA
sintetasi
Le tRNA-sintetasi riconoscono con facilità il loro tRNA specifico, ma hanno
maggiori difficoltà nel riconoscimento dell’amminoacido complementare. Il
(a)
(b)
Figura 17.30 Modelli (a) del complesso di classe I: GlnRS-tRNAGln e (b) del complesso
di classe II: AspRS-tRNA Asp di lievito (b). Per semplicità sono mostrati solo lo scheletro
dei fosfati del tRNA (in rosso) e lo scheletro di carbonio-α della sintetasi (in blu). Da notare
il legame delle due sintetasi da parti opposte dei tRNA. (Da: Ruff, M., S. Krishnaswamy, M.
Boeglin, A. Poterszman, A. Mitschler, A. Podjarny, B. Rees, J.C. Thierry e D. Moras, Class II
aminoacyl transfer RNA synthetases: Crystal structure of yeast aspartyl-tRNA synthetase
complexed with tRNA Asp. Science 252 (21 June 1991) f. 3, p. 1686. Copyright ©AAAS.)
•
W37
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W38 • La traduzione
Parte VI
motivo è chiaro: i tRNA sono molecole grandi, complesse e variabili tra loro
sia nella sequenza nucleotidica sia nelle modificazioni dei nucleosidi, mentre
gli amminoacidi sono molecole semplici e molto simili tra loro. Consideriamo per esempio l’isoleucina e la valina: i due amminoacidi sono identici a
eccezione del gruppo metilenico (CH2) extra dell’isoleucina. Nel 1958 Linus
Pauling, utilizzando nozioni termodinamiche, calcolò che l’isoleucil-tRNA
sintetasi (IleRS) nella reazione di caricamento dovrebbe generare circa un
quinto di Val-tRNA Ile, un accoppiamento errato, insieme all’accoppiamento
corretto Ile-tRNA Ile. Ciò invece non avviene, infatti solo un amminoacido
attivato da IleRS su 150 è una valina, e solo uno su 3000 amminoacil-tRNA
prodotti da questo enzima è un Val-tRNA Ile. Come può l’isoleucil-tRNA sintetasi arginare la formazione di Val-tRNA Ile ?
Nel 1977 Alan Fersht propose un meccanismo a doppio filtro, che impedisce la produzione di un tRNA caricato con un amminoacido errato. La
Figura 17.31 illustra questo meccanismo. Il primo filtro è compiuto dal sito
attivo dell’enzima, che rifiuta substrati troppo grandi. Ma substrati piccoli
come la valina possono entrare nel sito attivo, essere trasformati in forma
amminoacilata e in ultima analisi caricati su tRNA errati. Qui entra in gioco
il secondo tipo di filtro. L’amminoacido attivato o, meno frequentemente, gli
amminoacil-tRNA troppo piccoli, sono idrolizzati da un altro sito dell’enzima: il sito di correzione.
Per esempio, IleRS utilizza il primo filtro per escludere amminoacidi
troppo grandi, o amminoacidi con forme diverse da quelle corrette. L’enzima
esclude la fenilalanina perché troppo grande e la leucina perché di forma
errata (uno dei gruppi metile terminali della leucina non può entrare nel sito
Sito di attivazione
Amminoacidi
più grandi
rifiutati
Sito di correzione
Amminoacil-AMP
più piccoli accettati
Tyr
Phe
Val-AMP
Val + AMP
Ala
Ala-AMP
Ala + AMP
Gly
Gly-AMP
Gly + AMP
Val
IIe
Ile-AMP
Ile-tRNAIle
Figura 17.31 Il doppio filtro dell’isoleucil-tRNA sintetasi. Il sito di attivazione è un filtro
grossolano, nel quale gli amminoacidi di maggiori dimensioni, come Tyr e Phe, sono esclusi perché non possono entrare. Il sito di correzione (idrolitico) e il filtro più fine, che accetta amminoacidi attivati più piccoli dell’Ile-AMP, come Val-AMP, Ala-AMP e Gly-AMP, ma
rifiuta Ile-AMP perché troppo grande. Quindi gli amminoacidi attivati di minori dimensioni
sono idrolizzati ad AMP e amminoacido, mentre Ile-AMP viene trasformato in Ile-tRNAIle.
(Da: Fersht, A.R., Sives in sequence. Science 280:541, 1998. Copyright ©1998 American
Association for the Advancement of Science. Riproduzione autorizzata.)
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
Capitolo 17
Ribosomi e tRNA
•
W39
attivo dell’enzima). Che cosa succede ad amminoacidi come la valina? Questi
possono entrare nel sito di attivazione di IleRS, e quindi essere attivati; quando però sono trasportati al sito di correzione dell’enzima vengono riconosciti
come non corretti e degradati. Il secondo meccanismo di filtro viene chiamato anche sito di correzione delle bozze.
Shigeyuki Yokoyama e colleghi hanno ottenuto la struttura cristallografica dell’IleRS di Thermus thermophilus da solo, accoppiato con l’amminoacido isoleucina e con l’amminoacido errato valina. Questi risultati
hanno chiaramente verificato l’elegante ipotesi di Fersht. La Figura 17.32
mostra la struttura molecolare del sito di attivazione, a cui sono legate rispettivamente l’isoleucina (a) e la valina (b). Si può notare come entrambi
gli amminoacidi vengano agevolmente accolti dal sito di attivazione, anche se la valina forma contatti più deboli con i due residui amminoacidici
idrofobici (Pro46 e Trp558) che circondano il sito di attivazione dell’enzima; è chiaro invece come questo sito sia troppo piccolo per accogliere
amminoacidi grandi come la fenilalanina, o come possano intervenire
forti impedimenti sterici nel legame della leucina dovuti a uno dei due
residui metilici terminali dell’amminoacido. Questo quadro è quindi in
perfetto accordo con il primo meccanismo di filtro predetto dall’ipotesi
del doppio filtro.
L’enzima possiede un secondo solco, delle dimensioni di quello del sito di
attivazione, a una distanza da quest’ultimo di 34 Å. Questo secondo solco è
stato ipotizzato essere il sito di correzione, in quanto frammenti di enzima che
possiedono il solco mantengono attività di correzione; la struttura cristallografica dell’enzima conferma questa ipotesi: quando Yokoyama e colleghi hanno ottenuto la struttura cristallografica di IleRS con la valina, hanno trovato
l’amminoacido all’interno del solco. Tuttavia, quando sono stati preparati cristalli con isoleucina, nel solco non è stato trovato alcun amminoacido. Poiché
il solco sembra essere specifico per la valina, si ritiene che questo possa essere il
sito di correzione. L’analisi della tasca nella quale si trova la valina mostra che
lo spazio tra le catene laterali di Trp232 e di Tyr386 è grande abbastanza per
(a)
(b)
Figura 17.32 Immagine stereo del sito di attivazione di IleRS con l’isoleucina o la valina. Lo scheletro dell’enzima è rappresentato da un filamento turchese, con gli atomi di carbonio delle catene laterali in giallo. Gli atomi di carbonio dei substrati
(isoleucina in (a), valina in (b) sono mostrati in verde. Gli atomi di ossigeno di tutti gli amminoacidi sono in rosso e gli atomi di
azoto in blu. Notare come il sito di attivazione possa accogliere sia la valina che l’isoleucina. (Da: Nureki, O., D.G. Vassylyev, M.
Tateno, A. Shimada, T. Nakama, S. Fukai, M. Konno, T.L. Henrickson, P. Schimmel e S. Yokoyama, Enzyme structure with two
catalytic sites for double-sieve selection of substrate. Science 280 (24 Apr 1998) f. 2, p. 579. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
W40 • La traduzione
Copyright © 2009 - The McGraw-Hill Companies srl
Parte VI
accogliere la valina (Figura 17.33) ma è troppo piccolo per permettere l’accesso
all’isoleucina.
Se questo fosse effettivamente il sito di correzione, la sua rimozione dovrebbe abolire questo meccanismo. Infatti, quando Yokoyama e colleghi
hanno rimosso 47 amminoacidi da questa regione, tra cui Trp232, l’attività
di correzione era abolita, mentre si mantenevano la capacità di attivazione
dell’amminoacido; il secondo solco sembra quindi essere effettivamente il
sito di correzione. Diversi residui amminoacidici nel solco sono particolarmente vicini alla valina: Thr230 e Asn237 sono in buona posizione per
prendere parte alla reazione di idrolisi, essenziale per l’attività di correzione
delle bozze. Per valutare questa ipotesi Yokoyama e collaboratori hanno
cambiato Thr243 e Asn250 dell’ IleRS di E. coli, corrispondenti a Thr230 e
Asn237 dell’enzima di T. thermophilus. Il cambiamento di questi amminoacidi in alanina ha portato alla perdita della capacità di correzione dell’enzima, mantenendo la sua capacità di attivazione. Questi risultati sono coerenti con l’ipotesi secondo cui il secondo solco è il sito di correzione e l’idrolisi
degli amminoacil-AMP non corretti, come per esempio Val-AMP, avviene
in questo sito.
SOMMARIO La selettività amminoacidica, almeno di alcune amminoaciltRNA sintetasi, è controllata da un meccanismo a doppio filtro. Il primo
filtro, più grossolano, comporta l’esclusione degli amminoacidi troppo
grandi. L’enzima svolge questa attività grazie alle dimensioni del sito di
attivazione che permette l’accesso all’amminoacido corretto escludendo
quelli di maggiori dimensioni. Il secondo filtro è più selettivo e permette
la degradazione degli amminoacil-AMP troppo piccoli. Questo secondo
meccanismo è svolto da un secondo sito attivo dell’enzima (il sito di correzione) che accoglie gli amminoacil-AMP troppo piccoli e li idrolizza. L’amminoacil-AMP corretto, troppo grande per entrare nel sito di correzione,
viene rilasciato prima di subire idrolisi; l’enzima trasferisce quindi l’amminoacido attivato al suo specifico tRNA.
Figura 17.33 Immagine stereo della valina legata al sito di correzione di IleRS. I colori sono gli stessi utilizzati in Figura 17.32. (Da: Nureki, O., D.G. Vassylyev, M. Tateno, A.
Shimada, T. Nakama, S. Fukai, M. Konno, T.L. Henrickson, P. Schimmel e S. Yokoyama,
Enzyme structure with two catalytic sites for double-sieve selection of substrate. Science
280 (24 Apr 1998) f. 2, p. 579. Copyright ©AAAS.)
Biologia molecolare 2/ed
Robert F. Weaver
Capitolo 17
Copyright © 2009 - The McGraw-Hill Companies srl
Ribosomi e tRNA
•
W41
RIASSUNTO
Studi di cristallografia a raggi X sui ribosomi batterici
con e senza tRNA hanno mostrato che i tRNA occupano
il solco tra le due subunità. Essi interagiscono con la
subunità 30S tramite il loro anticodone e con la subunità
50S attraverso il braccio accettore. I siti di legame per i
tRNA sono composti principalmente di rRNA. Gli anticodoni dei tRNA nei siti A e P si avvicinano per permettere
il legame con i codoni adiacenti nell’mRNA legato alla
subunità 30S, grazie alla torsione di 45 gradi dell’mRNA
tra i due codoni. I bracci accettori dei tRNA nei siti A e
P si inseriscono nella tasca peptidil-transferasica della
subunità 50S con una distanza reciproca di circa 5 Å.
Sono visibili dodici contatti tra le subunità ribosomali.
La struttura cristallografica del ribosoma di E. coli
contiene due strutture che differiscono per i movimenti
del corpo rigido dei domini del ribososma, relativamente l’uno rispetto all’altro. In particolare, la testa della
particella 30S ruota di 6 gradi, e addirittura di 12 gradi
se confrontata con il ribosoma di T. thermophilus. Questa rotazione è probabilmente parte dell’azione rotante
del ribosoma che avviene durante la traslocazione.
La subunità 30S di E. coli contiene l’rRNA 16S e 21
proteine (S1-S21). La 50S presenta l’rRNA 5S, 23S e
34 proteine (L1-L34). I ribosomi citoplasmatici degli eucarioti sono più grandi e contengono più molecole di
rRNA e proteine rispetto ai procariotici. In vitro l’assemblaggio della subunità ribosomale 30S inizia con l’rRNA
16S. Le proteine, quindi, si legano sequenzialmente e
cooperativamente; le proteine che si legano precocemente aiutano le proteine tardive alla formazione della
particella.
Lo studio della sequenza dell’rRNA 16S ha permesso di proporre una struttura secondaria (dovuta
all’accoppiamento intramolecolare delle basi) di questa
molecola. Esperimenti di cristallografia a raggi X hanno
confermato la conclusione di questi studi. Gli esperimenti mostrano la subunità 30S con accoppiamento
estensivo tra le basi dell’rRNA 16S, la cui forma tratteggia quella della particella. Gli studi di cristallografia a
raggi X hanno inoltre confermato la localizzazione della
maggior parte delle proteine della subunità 30S.
La subunità ribosomale 30S ha due ruoli: facilita
la decodificazione tra codoni degli mRNA e anticodoni degli amminoacil-tRNA, inclusa la correzione delle
bozze, e partecipa alla traslocazione. Le strutture cristallografiche della subunità 30S con tre antibiotici che
interferiscono in queste due attività ne hanno rivelato i
meccanismi coinvolti. La spectinomicina lega la subunità 30S in prossimità del collo dove può interferire con
il movimento della testa necessario per la traslocazione. La streptomicina si lega vicino al sito A della subunità 30S, stabilizzando lo stato ram del ribosoma. Ciò
riduce la fedeltà di traduzione, permettendo a un amminoacil-tRNA non corretto di legarsi facilmente al sito A
impedendo il passaggio allo stato restrittivo necessario
per l’attività di correzione delle bozze. La paromomicina
si lega al solco minore dell’rRNA 16S nell’elica H44, in
prossimità del sito A. Ciò espone le basi A1492 e A1493,
stabilizzando l’appaiamento tra codone e anticodone,
includendo gli anticodoni non corretti e diminuendo la
fedeltà di traduzione.
La struttura cristallografica di IF1 legato alla subunità 30S mostra che questa proteina si lega al sito A. In
questa posizione blocca fMet-tRNA dal legame al sito
A, e può inoltre attivamente promuovere il legame di
fMet-tRNA al sito P tramite una presunta interazione tra
IF1 e IF2. IF1 inoltre interagisce strettamente con l’elica 44 della subunità 30S; ciò spiega come IF1 possa
accelerare sia l’associazione che la dissociazione delle
due subunità ribosomali.
La struttura cristallografica della subunità ribosomale 50S è stata ottenuta con una risoluzione di 2,4
Å. Questa struttura ha rivelato poche proteine presenti
all’interfaccia tra le subunità ribosomali e assenza di
proteine a 18 Å dal centro attivo peptidil-trasferasico
marcato con un analogo dello stato di transizione.
Il gruppo 2’OH del tRNA nel sito P è molto ben posizionato per formare un legame idrogeno con il gruppo
amminico dell’amminoacil-tRNA nel sito A, e quindi per
aiutare la reazione peptidil-transferasica. In accordo
con questa ipotesi, la rimozione del gruppo idrossile
elimina quasi completamente l’attività peptidil-transferasica. Allo stesso modo la rimozione del gruppo 2’-OH
dell’ A2451 dell’rRNA 23S inibisce fortemente l’attività
peptidil-transferasica. Anche questo gruppo può partecipare alla catalisi attraverso un legame idrogeno, oppure aiutando i reagenti a posizionarsi correttamente
per la catalisi.
Il tunnel di uscita della subunità ribosomale 50S è
grande abbastanza da permettere un ripiegamento ad
α-elica della proteina nascente. Le pareti del canale di
uscita sono costituite da RNA, il quale grazie alla sua
idrofilicità permette ai residui idrofobici della catena polipeptidica nascente di scivolare facilmente attraverso
di esso.
La maggior parte degli mRNA è tradotta da più di
un ribosoma simultaneamente; ciò risulta in una struttura chiamata polisoma. Negli eucarioti i polisomi sono
localizzati nel citoplasma. Nei procarioti la trascrizione
di un gene e la traduzione del suo mRNA sono processi
che avvengono simultaneamente; quindi molti polisomi
sono associati a geni attivamente trascritti.
I tRNA sono stati identificati come piccole specie
di RNA indipendenti dai ribosomi, che possono essere caricati con un amminoacido e trasferirlo alla catena
polipeptidica nascente. Tutti i tRNA mostrano la stessa
struttura secondaria a forma di trifoglio; possiedono
quattro regioni di appaiamento intramolecolare che formano tre strutture stelo-ansa, che prendono il nome di
ansa D, ansa anticodone e ansa T, inoltre formano uno
stelo chiamato braccio accettore, sito in cui viene legato l’amminoacido nel passaggio di caricamento. I tRNA
mostrano anche una comune struttura terziaria, che
assomiglia a una L rovesciata. Questo ripiegamento
rafforza la stabilità della molecola allineando gli appaiamenti di basi dell’ansa D con quelli dell’ansa anticodone
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl
W42 • La traduzione
e dell’ansa T con il braccio accettore. L’anticodone dei
tRNA protrude dall’ansa anticodone e assume un ripiegamento, necessario per l’appaiamento con il codone
complementare dell’mRNA.
Il braccio accettore e l’anticodone sono importanti siti di riconoscimento del tRNA da parte della sua
amminoacil-tRNA sintetasi. In alcuni casi questi due
elementi sono gli assoluti determinanti della specificità
di caricamento. Studi di cristallografia a raggi X hanno
dimostrato che le interazioni tra tRNA e sintetasi sono
differenti tra le amminoacil-tRNA sintetasi di classe I e
II. Le sintetasi di classe I hanno una tasca che accoglie
il braccio accettore e l’anticodone del loro specifico
tRNA, e legano tRNA dalla parte del braccio D e del
solco minore del braccio accettore; anche le sintetasi di classe II possiedono una tasca dove accolgono il
Parte VI
braccio accettore e l’anticodone, ma legano i tRNA dalla parte opposta, cioè dal braccio variabile e dal solco
maggiore del braccio accettore.
La selettività di caricamento di alcune sintetasi è
controllata da una meccanismo a doppio filtro. Il primo
filtro è più grossolano ed esclude gli amminoacidi troppo grandi. L’enzima raggiunge questo obiettivo in quanto possiede un sito di attivazione grande abbastanza da
accogliere l’amminoacido corretto, ma non amminoacidi di dimensioni maggiori. Il secondo filtro è più fine ed
è capace di degradare amminoacidi più piccoli di quello
corretto. L’enzima infatti possiede un secondo sito attivo (sito di correzione) che accoglie amminoacil-AMP
più piccoli e li idrolizza. L’amminoacil-AMP corretto è
invece troppo grande per entrare nel sito di correzione,
così sfugge all’idrolisi.
PER IL RIPASSO
1. Disegnare uno schizzo delle subunità ribosomali 30S e 50S
di E. coli e mostrare come esse si adattano insieme a formare il ribosoma 70S.
2. Disegnare uno schizzo dell’interfaccia tra le subunità ribosomali 50S e 30S. Far notare la posizione approssimativa
degli tRNA nei siti A, P ed E.
3. Quali parti degli tRNA interagiscono con la subunità 30S? E
con la subunità 50S?
4. Perché è importante che gli anticodoni siano posti in stretta
vicinanza nei siti A e P?
5. Perché è importante che i bracci accettori degli tRNA siano
posti in stretta vicinanza nei siti A e P?
6. Descrivere la tecnica di elettroforesi bi-dimensionale esposta in questo capitolo. In che modo la bi-dimensionale è
superiore all’elettroforesi mono-dimensionale?
7. Presentare ipotesi plausibili che spieghino come i seguenti
antibiotici interferiscono con la traduzione. Presentare l’evidenza sperimentale per ciascuna ipotesi.
a. Streptomicina
b. Paromomicina.
8. In che modo i dati di diffrazione a raggi X possono escludere le proteine ribosomali come il sito attivo nella peptidil
trasferasi?
9. Descrivere l’evidenza sperimentale per l’importanza nella
trnspeptidazione del gruppo 2’-OH dell’adenosina terminale del peptidil-tRNA nel sitoP. Come sembra partecipare alla
reazione di transpeptidazione questo gruppo idrossile?
10. Descrivere l’evidenza sperimentale per l’importanza nella
trnspeptidazione del gruppo 2’-OH dell’ A2451 dell’rRNA
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
23S. Come sembra partecipare alla reazione di transpeptidazione questo gruppo idrossile?
Come sappiamo che la base di A2451 (A2486 in H. marismortui) non è importante nella transpeptidazione?
Descrivere gli esperimenti che hanno condotto alla scoperta del tRNA.
Com’è stata scoperta la struttura secondaria a “trifoglio”
del tRNA?
Disegnare la struttura secondaria a “trifoglio” ed evidenziare gli elementi strutturali importanti.
Descrivere e fornire i risultati di un esperimento che mostri
che il ribosoma riconosce la parte tRNA, non la parte amminoacidica, di un amminoacil-tRNA.
Descrivere e fornire i risultati di un esperimento che mostri che l’appaiamento G3-U70 nel braccio accettore di un
tRNA è un determinante chiave nel caricamento del tRNA
con alanina.
Presentare almeno una linea di evidenza per l’importanza
dell’anticodone nel riconoscimento di un tRNA da parte di
un’amminoacil-tRNA sintetasi.
Sulla base di studi di cristallografia a raggi X, quali parti del
tRNA prendono contatto con la specifica amminoacil-tRNA
sintetasi?
Schematizzare il meccanismo a doppio filtro che assicura
la selettività amminoacidica dell’amminoacil-tRNA sintetasi.
Presentare l’evidenza sperimentale per il doppio filtro della
isoleucina-tRNA sintatasi che esclude gli amminoacidi più
grandi e quelli più piccoli.
PER L’APPROFONDIMENTO
1. Disegnare uno schema di un ipotetico polisoma eucariotico
in cui siano visibili le catene proteiche nascenti. Identificare
le estremità terminali 5’ e 3’ dell’mRNA ed utilizzare una
freccia per indicare la direzione dei ribosomi in movimento
lungo l’mRNA. Utilizzare N e C per indicare l’estremità ammino e carbossi terminali di uno dei polipeptidi crescenti.
2. Disegnare lo schema di un ipotetico gene procariotico che
viene trascritto e tradotto simultaneamente. Mostrare gli
mRNA nascenti con i ribosomi attaccatti, senza mostrare le
proteine nascenti. Con una fraccia, indicare la direzione di
trascrizione.
3. State studiando un tRNAPhe la cui specificità di caricamento sembra essere influenzata dall’appaiamento C11-G24
nell’ansa D. Disegnare due esperimenti che mostrino che
cambiando quest’appaiamento cambia la specificità di caricamento del tRNA. Il primo esperimento dovrebbe essere
biochimico utilizzando una reazione in vitro. Il secondo dovrebbe essere genetico ed effettuato in vivo.
Biologia molecolare 2/ed
Robert F. Weaver
Capitolo 17
Copyright © 2009 - The McGraw-Hill Companies srl
Ribosomi e tRNA
•
W43
LETTURE CONSIGLIATE
Cech, T.R. 2000. The ribosome is a ribozyme. Science 289:
878-79.
Dahlberg, A.E. 2001. The ribosome in action. Science 292:
868-69.
Fersht, A.R. 1998. Sieves in sequence. Science 280: 541.
Hill, W.E., P.B. Moore, A. Dahlberg, D. Schlessinger, R.A. Garrett e J.R. Warner eds. 1990. The Ribosome: Structure,
Function and Evolution. Washington, D.C.: American Society for Microbiology.
Noller, H.F. 1990. Structure of rRNA and its functional interactions in translation. In Hill, W.E., et al., eds. The Riboso-
me: Structure, Function and Evolution. Washington, D.C.:
American Society for Microbiology, c. 3, pp. 73-92.
Pennisi, E. 2001. Ribosome’s inner workings come into sharper view. Science 291: 2526-27.
Saks, M.E., J.R. Sampson e J.N. Abelson. 1994. The transfer
RNA identity problem: A search for rules. Science 263:
191-97.
Waldrop, M.M. 1990. The structure of the “second genetic
code.” Science 246: 1122.
Zimmermann, R.A. 1995. Ins and outs of the ribosome. Nature
376: 391-92.
Biologia molecolare 2/ed
Robert F. Weaver
Copyright © 2009 - The McGraw-Hill Companies srl