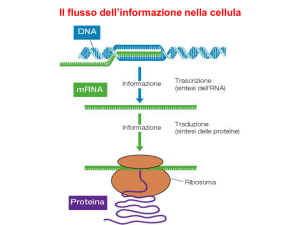
I R I BOSOM I
I RIBOSOMI sono organuli citoplasmatici presenti in tutte le
cellule, sia procariotiche che eucariotiche. Sono visibili al M.O.
solo quando presenti in gran numero, (come capita nelle cellule
nervose), ed appaiono come ammassi citoplasmatici di
materiale basofilo.
Nelle sezioni istologiche esaminate al M.E. si presentano come
particelle dense con diametro di circa 15-30 nm e possono essere
distribuiti nella matrice ialoplasmatica (RIBOSOMI LIBERI)
oppure essere adesi alla superficie esterna delle membrane del
RER (RIBOSOMI ASSOCIATI ALLE MEMBRANE).
Possono trovarsi liberi nel citoplasma
oppure adesi al R.E. In entrambi i casi si
possono trovare associati in gruppi,
denominati Polisomi o Poliribosomi,
costituiti da un esile filamento di RNA
messaggero
I POLISOMI possono assumere una
caratteristica forma a spirale. Le ricerche
con isotopi radioattivi hanno dimostrato
che le sintesi proteiche avvengono soltanto
SUI RIBOSOMI ASSOCIATI NEL
COMPLESSO POLISOMALE E NON
SUI RIBOSOMI SINGOLI, i poliribosomi
quindi rappresentano le unità attive di
sintesi della cellula.
Si tratta di
corpiccioli di natura
ribonucleoproteica
che nel citoplasma di
tutte le cellule
presiedono ai
processi di sintesi
proteica.
L’importanza che
essi rivestono è
notevole dato che le
interazioni tra RNA
messaggero, RNA
transfer e
amminoacidi,
sembrano dipendere
in larga misura
dall’organizzazione
molecolare del
ribosoma.
I ribosomi possono essere
isolati mediante
centrifugazione e la loro analisi
ha dimostrato che essi
presentano una considerevole
uniformità di dimensioni,
struttura e composizione. Sui
RIBOSOMI ISOLATI è
possibile valutare mediante
ultracentrifugazione il
coefficiente di sedimentazione
che è espresso in unità
Svedberg o unità S.
Mediante queste tecniche si è
potuto stabilire che i ribosomi dei
batteri hanno un coefficiente di
sedimentazione di 70S che
corrisponde ad un peso molecolare
di 2,6x106dalton, mentre quelli
delle cellule superiori sia animali
che vegetali hanno un coefficiente
di sedimentazione di circa 80S con
un peso molecolare di 4,5x 106
dalton.
Gli esperimenti hanno inoltre
dimostrato che i ribosomi sono
formati da due subunità
diseguali. Il ribosoma 70S è
costituito da una subunità
maggiore di 50S e una
subunità minore di 30S; il
ribosoma 80S da due subunità
di 60S e di 40S. L’associazione
tra le due subunità è
mantenuta dalla presenza di
MAGNESIO fino a
concentrazioni di 1mM.
Abbassando tale
concentrazione il ribosoma si
dissocia, elevando di nuovo la
concentrazione si riforma la
particella ribosomiale
completa. Se la
concentrazione di magnesio
aumenta notevolmente due
ribosomi si combinano per
formare un dimero.
Figura
3.45
Mediante metodi di centrifugazione su gradiente è possibile separare non soltanto
ribosomi singoli ma anche poliribosomi. Questi appaiono al M.E. come aggregati di
un numero variabile da tre a varie decine, di singole unità, (collegate, come già detto
prima, da un esile filamento di mRNA). Tale filamento è digerito dalla ribonucleasi
che dissocia il poliribosoma nei ribosomi singoli e si ritiene che sia una molecola di
mRNA che porta l’informazione genetica per la sintesi proteica dal DNA ai
ribosomi.
È stata avanzata l’ipotesi che la molecola di mRNA sia interposta tra le due
subunità del ribosoma; tale configurazione può spiegare come circa 25 nucleotidi
dell’mRNA polisomale siano protetti dall’azione della ribonucleasi quando il
polisoma è trattato con questo enzima. La lunghezza dell’mRNA e il numero di
monomeri del polisoma possono variare in misura notevole e dipendono dalla
lunghezza della catena polipeptidica che ogni molecola specifica di mRNA può
codificare.
Sebbene non esista una
dimostrazione diretta
al M.E., le indagini di
biologia molecolare
fanno ritenere che
nella cellula i ribosomi
singoli siano sempre
dissociati nelle due
subunità e che queste
si uniscano tra loro e
con la molecola di
mRNA formando il
polisoma soltanto
durante il processo di
sintesi proteica, per
dissociarsi al termine
di esso.
L’analisi chimica dei
ribosomi isolati ha
dimostrato che
entrambe le subunità
sono composte da
molecole di acido
ribonucleico ribosomiale
(rRNA) e da proteine in
proporzioni pressoché
uguali.
Nei procarioti il rapporto in peso tra rRNA e proteine è di 2:1, la subunità
minore 30S contiene un’unica molecola di RNA 16S alla quale sono associate
circa 21 proteine diverse.
La subunità maggiore 50S ha una molecola di RNA 23S ed una di 5S ad
esse sono legate circa 35 proteine.
I ribosomi degli eucarioti sono più complessi: la subunità minore 40S
contiene una molecola di RNA 18S e la subunità maggiore 60S tre
molecole di RNA: una 28S, una5,8S ed una 5S le proteine ribosomiali
ammontano a 80 distribuite nelle due subunità (circa 50 nella sub
maggiore e 30 in quella inferiore). La maggior parte di queste proteine è
di carattere basico.
Nei mitocondri e nei cloroplasti si trovano
ribosomi simili a quelli procariotici. Nei lieviti i
ribosomi mitocondriali (o MITORIBOSOMI),
sono di 70S, nei protozoi sono di 80S, ma con
due subunità entrambe di 55S.
Nei mitoribosomi di 55S sono presenti solo due
RNA, uno di 12 S nella subunità minore ed uno
di 21 S in quella maggiore.
La struttura secondaria delle molecole di rRNA è
poco conosciuta, ma si ritiene che esse siano
costituite da una CATENA UNICA RIPIEGATA IN
ALCUNE ZONE SU SE STESSA A FORMARE
TRATTI A DOPPIA ELICA CON APPAIAMENTO
COMPLEMENTARE DELLE BASI.
La caratteristica dell’rRNA è di avere un
contenuto in guanina+citosina più elevato(60%)
del contenuto in adenina+uracile (40%) e di
essere relativamente stabile.
Tutte le molecole di rRNA, 28S,5,8S,5S e 18S
sono caratterizzate dalla presenza, in aggiunta
alle 4 principali basi azotate, di basi metilate, di
pseudouridina e di 2-0-metilribosio
L’RNA ribosomiale rappresenta la frazione più grande di RNA nel citoplasma
costituendo il 70-85% dell’RNA totale.
Sia nei batteri che nelle
cellule superiori, la subunità
maggiore del ribosoma
contiene anche una molecola
di RNA di modeste
dimensioni 5S costituita da
120 nucleotidi. LA SUA
STRUTTURA È
COSTITUITA DA UN
FILAMENTO UNICO
RIPIEGATO IN ALCUNI
PUNTI SU SE STESSO A
FORMARE TRATTI A
DOPPIA ELICA.
La funzione di tale struttura è ancora
sconosciuta. La modalità di interazione tra
RNA e proteine nelle subunità non è
completamente nota. Tuttavia si ritiene che la
molecola di RNA e proteine ribosomiche siano
tra loro intimamente associate.
Le proteine interagiscono con la molecola di RNA principalmente nelle
regioni a catena singola, formando un filamento nucleoproteico che a sua
volta fittamente avvolto su se stesso a formare una particella compatta.
È attualmente ben stabilito che i ribosomi si originano nel nucleolo.
Figura 3.47
INNESCO O INIZIO DELLA
SINTESI PROTEICA
La fase iniziale della biosintesi
proteica è definita come la
sequenza di eventi che porta alla
formazione di un complesso tra
un ribosoma 80S, una molecola di
metionina-t-RNA(met-t-RNA) ed
una molecola di mRNA. Diverse
proteine non ribosomiche sono
necessarie per la formazione di
questo complesso di inizio ed esse
sono dette Fattori di inizio(IF).
Un ribosoma contiene 4 siti di legame per
molecole di RNA: uno per l’mRNA e tre
(sito E, sito P e sito A) per il tRNA
ALLUNGAMENTO DELLA CATENA POLIPEPTIDICA
Dopo che si è formato il complesso
d’inizio, il met-t-RNA è sul sito
peptidico (sito P) del ribosoma. Un
secondo aminoacil-t-RNA
corrispondente alla seconda tripletta
dell’mRNA si colloca sulla subunità
maggiore del ribosoma legandosi al
sitoaminoacidico (sito A) .
Sul ribosoma sono ora localizzate due
molecole di tRNA affiancate e legate
a due codon consecutivi dell’mRNA;
esse sono disposte l’una in posizione
prossimale rispetto all’estremità
5’dell’mRNA e l’altra in posizione
distale. L’attacco dell’aminoacil-t-RNA
al sito A del ribosoma richiede GTP
come fornitore di energia ed è
promosso dal cosiddetto “fattore di
allungamento”(EF1).
La metionina (e nelle tappe successive la catena polipeptidica nascente) si
stacca dal suo tRNA per rottura del legame estere tra l’adenosina
terminale dell’RNA di trasporto ed il gruppo carbossilico dell’aminoacido;
tra questo gruppo ed il gruppo aminico del secondo aminoacil-t-RNA
affiancato al codon successivo si forma il legame peptidico.
L’AMINOACIL-T-RNA
LEGATO AL SITO A
RICEVENDO LA
CATENA
POLIPEPTIDICA
DIVENTA PEPTIDILT-RNA. La formazione
del legame peptidico è
catalizzata dall’enzima
peptiltransferasi che è
localizzato sulla
subunità 60S ed è
forse parte integrante
del sito P.
Il peptidil-tRNA si dissocia dal sitoA, rimanendo tuttavia
Il sito A è in questo
ancorato al suo codon, ed il ribosoma si sposta rispetto al
momento vacante. Il
complesso tRNA –mRNA di un codon verso l’estremità 3’
dell’mRNA, oppure l’mRNA scorre relativamente al ribosoma
in direzione opposta. Come conseguenza il met-tRNA deacilato
processo di
traslocazione dell’mRNA
relativamente al
esce dal ribosoma ed il peptidil-tRNA sempre ancorato alla
ribosoma e del peptidil-
sua tripletta dell’mRNA viene a collocarsi sul sito P.
tRNA dal sito A al sito P
è forse il risultato di un
cambiamento
conformazionale del
ribosoma che avviene a
spese dell’idrolisi del
GTP e richiede
l’intervento del fattore
proteico EF2 o
traslocasi; per ogni
tappa di traslocazione si
forma un complesso
EF2-GTP che si lega al
ribosoma.
Un terzo aminoacil-tRNA complementare alla terza tripletta dell’mRNA si
attacca al ribosoma collocandosi sul suo sito aminoacidico vacante. Sul ribosoma
sono di nuovo situati fianco a fianco due tRNA legati a due codon contigui
dell’mRNA : il peptidil-tRNA che porta la catena polipeptidica nascente ed è
inserito sul sito P e l’aminoacil-tRNA che porta l’ultimo aminoacidico incorporato
ed è associato al sito A.
Con l’attacco del nuovo
aminoacil-tRNA, la
situazione è ritornata
a quella di partenza,
con la differenza che
la catena polipeptidica
si è accresciuta di un
aminoacido e la
traduzione dell’mRNA
è progredita di un
codon.
Il ciclo costituito dalle precedenti fasi
si ripete per ogni codon, incorporando
un aminoacido per volta, finché tutto
l’mRNA è tradotto.
sitoP
sitoA
TERMINAZIONE DELLA TRADUZIONE
Quando l’ultima tripletta
dell’mRNA alla sua estremità
3’ è stata letta, la traduzione
si arresta e la catena
polipeptidica si libera dal
ribosoma e dal tRNA,
contemporaneamente alla
dissociazione del ribosoma
nelle due subunità. Un fattore
di natura proteica detto
Fattore di dissociazione
interviene nella separazione
delle due subunità. Il segnale
di terminazione è dato da una
opiù triplette NON SENSO
(UAA, UGA, UAG) cioè non
traducibili mancando il
rispettivo tRNA.
TERMINAZIONE DELLA TRADUZIONE
Quando il peptidil-tRNA è nel
sito P ed una tripletta non senso
di terminazione è nel sito A, un
fattore proteico di rilascio RF si
lega al sito A in presenza di GTP
e catalizza la reazione di
terminazione. Questa reazione
consiste nell’idrolisi del legame
che unisce il peptide al tRNA ,
nel loro rilascio, nel distacco
dell’mRNA dal ribosoma ed
infine nell’idrolisi del GTP.