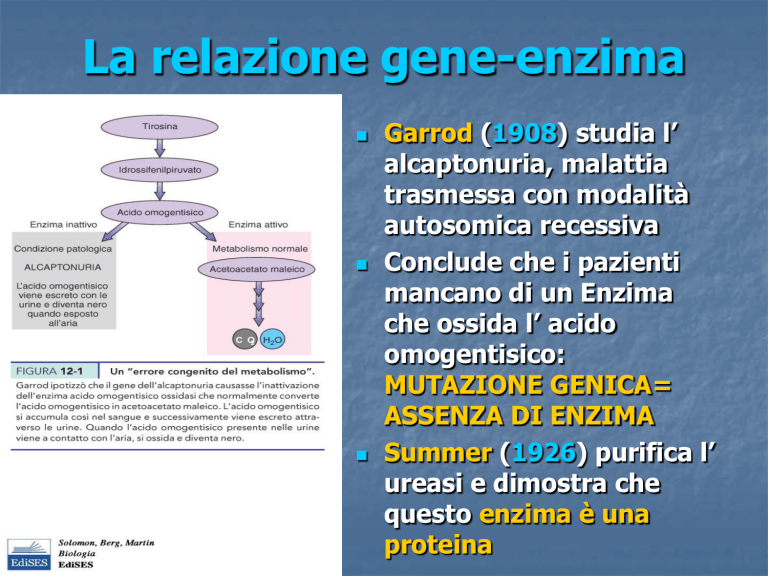
La relazione gene-enzima
Garrod (1908) studia l’
alcaptonuria, malattia
trasmessa con modalità
autosomica recessiva
Conclude che i pazienti
mancano di un Enzima
che ossida l’ acido
omogentisico:
MUTAZIONE GENICA=
ASSENZA DI ENZIMA
Summer (1926) purifica l’
ureasi e dimostra che
questo enzima è una
proteina
Ipotesi un gene-un enzima
Beadle e Tatum (1940): cercarono le mutazioni
che interferiscono con le reazioni metaboliche
Usarono ceppi Wilde-Type di Neurospora
(cresce come aploide) capaci di crescere in
terreno minimo (zucchero, sali e biotina).
Tramite radiazioni ionizzanti ottennero dei
mutanti, capaci di crescere in un terreno
completo o in un terreno minimo + il composto
richiesto per la crescita (essenziale)
Ogni ceppo mutante presentava una mutazione
in un solo locus genico e ciascun locus genico
influenzava solo un enzima (un gene – un
enzima)
Pauling (1949): la struttura dell’ Hb può essere
alterata dalla mutazione di un singolo locus
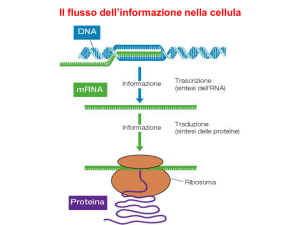
L’ RNA
E’ l’ intermediario fra
DNA e proteine
Fornisce l’ informazione
per dirigere la sintesi
proteica
E’ a singolo filamento
Contiene il ribosio
L’ Uracile sostituisce la
Timina (UA= basi
complementari)
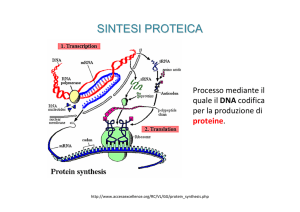
Trascrizione e traduzione
Un RNA messaggero (mRNA) a singola elica viene
trascritto da una semielica del DNA (filamento
stampo) e porta l’ informazione per la sintesi proteica
Nel processo di traduzione la sequenza nucleotidica
viene tradotta nella sequenza aminoacidica: la
sequenza di tre nucleotidi (tripletta) dell mRNA
chiamata codone determina l’ incorporazione di un AA
Gli RNA di trasferimento (tRNA) sono degli adattatori:
da una parte tramite l ‘anticodone si legano per
complementarietà al codone del mRNA e dall’ altra
portano lo specifico AA
Gli AA portati dai tRNA devono legarsi tramite legami
peptidici nell’ ordine dettato dal messaggero e a
questo sono deputati i ribosomi. Questi organuli fatti
da Pt. e RNA ribosomale (rRNA) si attaccano all’
estremità del mRNA e permettono ai tRNA di decifrare
il messaggio.
Il codice genetico (I)
Come fanno 4 nucleotidi a codificare per 20 AA?
Nel 1961 Crick e Brenner ipotizzarono che il codice
fosse basato su triplette non sovrapposte: 4 basi si
possono unire a 3 a 3 dando 64 combinazioni possibili
Nirenberg e Matthaei (1968) provano
sperimentalmente che specifiche triplette codificano
per specifici AA: l’ mRNA poli U codifica per Phe; il
poli A per Lys; il poli G per Gly; il poli C per Pro etc.
UAA, UGA, UAG non codificano per alcun AA, ma sono
codoni di stop o terminazione
Il codice genetico si riferisce all’ mRNA, mentre le
sequenze sul DNA o dell’ anticodone del tRNA sono
complementari
Il codice genetico (II)
E’ universale (solo in alcuni
protozoi UAA e UGA
codificano per Glu e non
sono di Stop) e quindi si è
evoluto molto
precocemente
E’ ridondante o degenerato
(solo Met e Trp sono
codificati da una sola
tripletta, mentre per gli
altri AA ci sono da due a sei
triplette-sinonimi che in
genere differiscono per il
terzo nucleotide)
La trascrizione (I)
Le RNA polimerasi DNA
dipendenti sintetizzano
mRNA, tRNA e rRNA su
uno stampo di DNA
Utilizzano nucleosidi
trifosfati che vengono
idrolizzati liberando PP
(tranne il primo all’
estremità 5’ che resta
trifosfato)
La sintesi di RNA avviene
in direzione 5’→3’ su uno
stampo di DNA letto in
direzione 3’→5’.
La trascrizione (II)
Il promotore è la sequenza di DNA non trascritta alla
quale si lega l’ RNA polimerasi per iniziare la
trascrizione della regione codificante per la proteina
Quando la RNA Polimerasi riconosce il promotore si
srotola la doppia elica ed avviene la trascrizione senza
bisogno di primer
Il termine della trascrizione è controllato da sequenze
di terminazione che determinano il distacco della DNA
pol dal DNA stampo e dall’ RNA neosintetizzato
In genere uno solo dei due filamenti di una regione
codificante viene trascritto
Uno stesso filamento può fungere da filamento
trascritto per alcuni geni e da filamento non trascritto
per altri
L’ mRNA contiene sequenze non codificanti
L’ RNA polimerasi inizia la trascrizione di un
gene a monte della sequenza codificante
L’ mRNA ha al 5’ una sequenza leader non
codificante fondamentale per l’ interazione
con il ribosoma
La sequenza codificante contiene il
messaggio per la proteina e termina con un
codone di stop (UAA,AGA,UAG)
Infine ci sono sequenze non codificanti
trailing al 3’ di lunghezza variabile
La traduzione
Negli eucarioti l’ mRNA va dal nucleo al
citoplasma dove avviene la traduzione
della sequenza nucleotidica in sequenza
aminoacidica
Sono necessari degli adattatori molecolari,
i tRNA
Le aminoacil tRNA sintetasi legano i
rispettivi AA ai t RNA con consumo di ATP
Si originano gli aminoacil tRNA in grado di
legarsi all’ mRNA nel giusto ordine per
originare la Pt
RNA transfer
I tRNA sono costituiti da 70-80 nucleotidi
con sequenze comuni e sequenze uniche
Alcune regioni si appaiano, mentre altre
formano anse
L’ ansa 2 costituisce l ‘anticodone che
riconosce il codone dell’ mRNA
Al 3’ OH c’è l’estremità che lega il gruppo
COOH dell’ AA (mentre il gruppo NH2
partecipa alla formazione del legame
peptidico)
Sono riconosciuti dalle specifiche aminoacil
tRNA sintetasi
Sono riconosciuti dai ribosomi
I ribosomi (I)
Eucariotici (80S) e Procariotici (70S) sono
costituiti da due subunità fatte da rRNA e
Proteine
L’ rRNA non trasferisce informazioni ma ha
funzioni catalitiche, le Pt ribosomali hanno
funzione strutturale nel ribosoma , ma non
funzione catalitica
La subunità maggiore contiene una
depressione sulla quale si adatta la subunità
minore
Fra le superfici di contatto si forma una
scanalatura nella quale scorre l’ mRNA
I ribosomi (II)
I ribosomi hanno 1 sito di legame per l’
RNA e 3 siti di legame per i tRNA
Le molecole di t RNA si attaccano a tre siti
(depressioni) presenti sul ribosoma
Sito P (peptidilico): è occupato dal tRNA
che porta la catena polipeptidica crescente
Sito A (aminoacilico): a cui si lega l’
aminoacil-tRNA che porta l’ AA da inserire
nella catena polipeptidica
Sito E (di uscita): sito in cui i tRNA che
hanno fornito alla catena l’ AA, escono dal
ribosoma
Inizio della traduzione nei
procarioti (I)
Tre fattori di inizio proteici si attaccano alla
subunità ribosomale minore
La subunità ribosomale minore lega l’ mRNA a
livello del codone di inizio (AUG)
La sequenza leader, a monte dell’ AUG, aiuta il
ribosoma ad identificare l’ inizio della regione
codificante dell’ mRNA
Il tRNA che porta il primo AA è il tRNA iniziatore
e lega una metionina formilata (fMet) come
primo AA che in seguito può essere rimossa
Inizio della traduzione nei
procarioti (II)
Il fMet –tRNA iniziatore ha l’ anticodone
UAC che si lega al codone AUG rilasciando
uno dei fattori di inizio
La subunità ribosomale maggiore si
assembla con la minore rilasciando i
rimanenti fattori di inizio
Il complesso di inizio è ora completo
Inizio della traduzione negli
eucarioti
La Met legata al t RNA iniziatore non
è formilata
Il codone di inizio sull’ mRNA non è
identificato grazie alla sequenza
leader
Il complesso di inizio è più complesso
e coinvolge almeno dieci fattori
proteici
Allungamento (I)
Gli AA sono aggiunti al polipeptide in crescita
L’ Aminoacil t RNA riconosce il codone nel
sito A e vi si lega mediante l’ appaiamento
del codone (mRNA) e dell’ anticodone (tRNA)
La formazione del legame peptidico richiede
proteine (fattori di allungamento) ed energia
(GTP)
Il gruppo NH2 del nuovo AA sul sito A è
allineato al gruppo COOH dell’AA precedente
(sito P) e si forma il legame peptidico
Allungamento (II)
L’AA sul sito P viene rilasciato dal suo tRNA e
legato all’ Aminoacil t RNA posto sul sito A
Questa reazione è spontanea ma richiede l’
intervento della peptidil transferasi un
catalizzatore (ribozima) costituente dell
rRNA della subunità ribosomale maggiore
Segue la traslocazione del ribosoma (che
richiede GTP) processo durante il quale il
ribosoma scorre sul mRNA di un codone
Ora il codone dell’ mRNA che codifica per l’
AA successivo si trova sul sito A
Il tRNA non carico viene rilasciato dal sito E
La terminazione
In presenza del codone di stop sull’
mRNA non si lega alcun t RNA ed il
sito A può legare un fattore di rilascio
proteico
Quindi il legame fra l’ ultimo AA e il t
RNA nel sito P si rompe e si libera il
polipeptide
Si disassembla il complesso di
traduzione: mRNA, fattore di rilascio,
tRNA del sitoP, subunità ribosomali