LEZIONE IX
SINTESI PROTEICA
Dott. Paolo Cascio
L’ESTREMITA’
3’
DEL
tRNA
SI
LEGA
AD
UN
DETERMINATO
AMINOACIDO CON UN LEGAME COVALENTE AD ALTA ENERGIA. IL
PRODOTTO DI QUESTA REAZIONE E’ UNA MOLECOLA DI AMINOACILtRNA.
LE MOLECOLE DI AMINOACIL-tRNA SONO SPESSO DETTE
MOLECOLE
DI
tRNA
CARICHE.
IL
TIPO
SPECIFICO
DI
tRNA
AMINOACILATO VIENE INDICATO CON UN PREFISSO: AD ESEMPIO
ALANIL-tRNAALA.
GLI ENZIMI CHE CATALIZZANO L’ATTACCO DEGLI AMINOACIDI ALLE
SPECIE CORRISPONDENTI DI tRNA SONO DETTI AMINOACIL-tRNASINTETASI.
POICHE’ ESISTONO 20 DIVERSI AMINOACIDI, IN CIASCUNA CELLULA
SONO PRESENTI 20 DIVERSE AMINOACIL-tRNA-SINTETASI (NON DI PIU’,
NONOSTANTE CHE IL CODICE GENETICO SIA DEGENERATO, PERCHE’
UNA SINTETASI E’ CAPACE DI RICONOSCERE LE DIVERSE MOLECOLE DI
tRNA CHE SI LEGANO ALLO STESSO AMINOACIDO “tRNA isoaccettori”).
1 - UNO STESSO TRNA PUÒ RICONOSCERE PIÙ CODONI DIVERSI (CHE
CODIFICANO PERÒ PER LO STESSO AMINOACIDO)
ES:
mRNA
tRNAALA
C
G
I
G
C
U
G
C
C
G
C
A
G
G
G
Codoni per l’alanina
2 – TRNA DIVERSI (CHE PORTANO PERÒ LO STESSO TIPO DI
AMINOACIDO E CHE VENGONO QUINDI RICONOSCIUTI DA UNA STESSA
AMMINOACIL-TRNA SINTETASI) POSSONO LEGARSI AD UN UNICO
CODONE
ES:
tRNAHIS
G
U
I
tRNAHIS
G
U
A
tRNAHIS
G
U
G
mRNA
C
A
U
Codone per l’istidina
N.B. LA COPPIA DI BASI G-U È COMPATIBILE QUASI QUANTO LA COPPIA
STANDARD G-C
LE VARIE AMINOACIL-tRNA-SINTETASI PUR CATALIZZANDO TUTTE LO
STESSO TIPO DI REAZIONE DIFFERISCONO NOTEVOLMENTE NELLA
CONPOSIZIONE IN SUBUNITA’ E NEL PESO MOLECOLARE: ALCUNE
SONO MONOMERI, ALTRE SONO GROSSI COMPLESSI MULTIMERICI
FORMATI DA UN SOLO TIPO DI MONOMERO O DA PIU’ MONOMERI
DIVERSI.
LA REAZIONE COMPLESSIVA CATALIZZATA DA UNA AMINOACIL-tRNASINTETASI E’:
AMINOACILtRNA-SINTETASI
AMINOACIDO+tRNA+ATP
AMINOACIL-tRNA+AMP+PP
L’AMINOACIDO VIENE LEGATO AL tRNA TRAMITE UN LEGAME ESTERE
TRA IL PROPRIO GRUPPO CARBOSSILICO E IL GRUPPO OSSIDRILICO IN
2’ O 3’ DEL RIBOSIO CHE SI TROVA ALL’ESTREMITA’ 3’ DELLA
MOLECOLA DI tRNA. QUESTA REAZIONE AVVIENE IN DUE FASI BEN
SEPARATE.
NELLA PRIMA FASE IL GRUPPO CARBOSSILICO DELL’AMINO-ACIDO SI
ATTACCA AL FOSFORO IN α DELL’ATP LIBERANDO UN PIROFOSFATO. IL
PRODOTTO DI QUESTA REAZIONE E’ UN AMINOACILADENILATO CHE E’
UNA MOLECOLA AD ALTA ENERGIA (PER QUESTO MOTIVO SI DICE CHE
SI ATTIVA L’AMINOACIDO).
L’ENERGIA
NECESSARIA
PER
LA
FORMAZIONE
DELL’
AMINOACILADENILATO E’ FORNITA DALL’IDROLISI DEL PIROFOSFATO.
NELLA SECONDA FASE IL GRUPPO AMINOACILICO DELL’AMINOACILADENILATO VIENE TRASFERITO AL GRUPPO OSSIDRILICO 2’ O 3’ DEL
RIBOSIO PRESENTE ALL’ESTREMITA’ 3’ DEL tRNA (QUALE DEI DUE
OSSIDRILI VIENE UTILIZZATO DIPENDE DAL TIPO DI ENZIMA).
UNA VOLTA CHE IL RESIDUO AMINOACILICO SI E’ LEGATO AL RIBOSIO
TERMINALE DEL tRNA ESSO SI SPOSTA SPONTANEAMENTE TRA LE
POSIZIONI 2’ E 3’. DURANTE LA SINTESI PROTEICA, PERO’, GLI
AMINOACIDI DEVONO TROVARSI NELLA POSIZIONE 3’ PER POTER
ESSERE
TRASFERITI
SULLA
CATENA
POLIPEPTIDICA
IN
ALLUNGAMENTO.
NELLA TRADUZIONE DEL MESSAGGIO GENETICO, IL RICONOSCIMENTO
DI UN AMINOACIDO SPECIFICO E DI UNA MOLECOLA SPECIFICA DI tRNA
DA PARTE DELL’AMINOACIL-tRNA-SINTETASI E’ UNA FASE CRITICA. SE
QUESTO PROCESSO NON SI SVOLGESSE IN MANIERA ACCURATA NELLA
PROTEINA VERREBBERO INCORPORATI AMINOACIDI ERRATI. PER
QUESTO MOTIVO LE AMINOACIL-tRNA-SINTETASI ATTACCANO GLI
AMINOACIDI ALLE CORRISPONDENTI MOLECOLE DI tRNA CON UNA
NOTEVOLE SPECIFICITA’.
OGNI AMINOACIL-tRNA-SINTETASI SELEZIONA IL PROPRIO SUBSTRATO
AMINOACIDICO IN BASE ALLE DIMENSIONI E ALLA CARICA.
SULLA SUPERFICIE DI OGNI AMINOACIL-tRNA-SINTETASI E’ PRESENTE
UNA TASCA DI LEGAME CHE ACCOGLIE IN MANIERA SPECIFICA UN
SOLO TIPO DI AMINOACIDO. UNA PRIMA SELEZIONE VIENE OPERATA
SULLA BASE DELLE DIMENSIONI DEGLI AMINOACIDI: AMINOACIDI
TROPPO GROSSI RISPETTO ALLA TASCA NON POSSONO ESSERE
ACCOLTI E VENGONO COSI’ ESCLUSI PER MOTIVI STERICI.
ANCHE AMINOACIDI TROPPO PICCOLI POSSONO PERO’ ESSERE
SFAVORITI NELL’INTERAGIRE CON LA TASCA, PERCHE’ POSSONO
STABILIRE MENO LEGAMI DI VAN DER WAALS RISPETTO AD UN
AMINOACIDO
AVENTE
LE
L’ISOLEUCIL-tRNA-SINTETASI
GIUSTE
E’
100
DIMENSIONI.
AD
VOLTE
AFFINE
L’ISOLEUCINA CHE PER LA PIU’ PICCOLA VALINA.
PIU’
ESEMPIO
PER
GLI AMINOACIDI VENGONO POI SELEZIONATI IN BASE ALLA
LORO
CARICA E IDROFOBICITA’. AD ESEMPIO LA TIROSIL tRNA-SINTETASI
LEGA LA TIROSINA E NON LA FENILALANINA PERCHE’ LA TIROSINA HA
UN OSSIDRILE CON IL QUALE L’ENZIMA PUO’ FORMARE DEI PONTI H.
LE
AMINOACIL-tRNA-SINTETASI
RICONOSCONO
IN
MANIERA
ALTAMENTE SPECIFICA NON SOLO I DIVERSI AMINOACIDI, MA ANCHE
LE DIVERSE MOLECOLE DI tRNA. QUESTO RICONOSCIMENTO E’
MEDIATO
DALLA
FORMAZIONE
DI
LEGAMI
SPECIFICI
TRA
L’AMINOACIL-tRNA-SINTETASI E L’ANSA DELL’ANTICODONE DEL tRNA.
ALTRE BASI IMPORTANTI PER IL RICONOSCIMENTO SI TROVANO SULLO
STELO ACCETTORE.
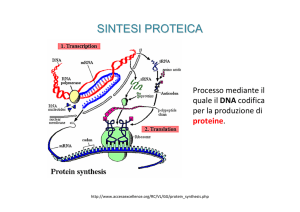
SINTESI PROTEICA NEI PROCARIOTI
LA SINTESI PROTEICA PUO’ ESSERE DISTINTA IN TRE FASI: INIZIO,
ALLUNGAMENTO DELLA CATENA E FINE.
L’INIZIO COINCIDE CON LA DISSOCIAZIONE DEL RIBOSOMA 70S NELLE
DUE SUBUNITA’ 30S E 50S. LA SUBUNITA’ 30S SI LEGA QUINDI
ALL’mRNA E LA SUBUNITA’ 50S SI RIASSOCIA A QUELLA 30S PER
FORMARE UN RIBOSOMA 70S ATTIVO.
IL RIBOSOMA 70S ATTIVO RIMANE ASSOCIATO ALL’mRNA DURANTE
TUTTA LA FASE DI ALLUNGAMENTO DELLA CATENA.
ALLA FINE DELLA SINTESI DELLA CATENA POLIPEPTIDICA IL
RIBOSOMA 70S SI DISSOCIA NUOVAMENTE, SI SEPARA DALL’mRNA E LE
DUE SUBUNITA’ 30S E 50S POSSONO RIFORMARE UN RIBOSOMA 70S
INATTIVO.
L’AVVIO DELLA SINTESI PROTEICA RICHIEDE LA FORMAZIONE DI UN
COMPLESSO D’INIZIO 70S, FORMATO DALLE SUBUNITA’ 30S E 50S,
DALL’mRNA E DA UN PARTICOLARE METIONIL-tRNA (tRNA INIZIATORE
O tRNAiMet). IL tRNA INIZIATORE VIENE RICONOSCIUTO DA UNA
TRASFERASI
CHE
TRASFERISCE
UN
GRUPPO
FORMILICO
ALLA
METIONINA FORMANDO N-FORMIL METIONINA. PER QUESTO LA NFORMILMETIONINA E’ IL PRIMO AMINOACIDO DI QUASI TUTTE LE
PROTEINE BATTERICHE.
IL COMPLESSO D’INIZIO NON SI FORMA SPONTANEAMENTE, MA
RICHIEDE L’INTERVENTO DI 3 FATTORI D’INIZIO: IF-1, IF-2 E IF-3.
IF-3 SI LEGA AL RIBOSOMA 70S E DETERMINA LA SEPARAZIONE DELLE
DUE SUBUNITA’ 30S E 50S.
IF-1 E IF-2 SI LEGANO ALLA SUBUNITA’ 30S E COSI’ LEGATA LA
SUBUNITA’ 30S PUO’ LEGARSI ALL’fMET-tRNAiMet E ALL’mRNA.
PER ULTIMA SI LEGA LA SUBUNITA’ 50S E SI FORMA COSI’ IL
COMPLESSO D’INIZIO ATTIVO. LA FORMAZIONE DEL COMPLESSO
D’INIZIO
ATTIVO
70S
RICHIEDE
ENERGIA
CHE
E’
FORNITA
DALL’IDROLISI DEL GTP LEGATO A IF-2.
IL CORRETTO POSIZIONAMENTO DEL RIBOSOMA IN PROSSIMITA’ DEL
CODONE D’INIZIO AUG E’ RESO POSSIBILE DALL’APPAIAMENTO TRA
UNA SERIE DI TRE-NOVE PURINE DELL’mRNA LOCALIZZATE A CIRCA 10
NUCLEOTIDI DALL’ESTREMITA’ 5’ DEL CODONE D’INIZIO (SEQUENZA
DI SHINE-DELGARNO) E UNA SEQUENZA DI PIRIMIDINE PRESENTI
NELL’rRNA 16S DEL RIBOSOMA.
L’APPAIAMENTO DI QUESTI NUCLEOTIDI COMPLEMENTARI PERMETTE
IL POSIZIONAMENTO DEL RIBOSOMA NELLA POSIZIONE CORRETTA PER
POTER INIZIARE LA SINTESI PROTEICA.
GLI mRNA POLICISTRONICI CONTENGONO VARIE SEQUENZE DI SHINEDALGARNO (UNA PER OGNI PROTEINA) A LIVELLO DELLE QUALI SI
ASSOCIA IL COMPLESSO D’INIZIO 70S E INIZIA LA TRADUZIONE DI
CIASCUNA PROTEINA
L’ALLUNGAMENTO DI UNA CATENA PROTEICA AVVIENE MEDIANTE UN
MINICICLO RIPETUTO DI TRE FASI NEL CORSO DEL QUALE OGNI
ULTERIORE
AMINOACIDO
VIENE
AGGIUNTO
ALLA
CATENA
POLIPEPTIDICA NASCENTE.
LE TRE FASI DI QUESTO CICLO SONO:
1)
POSIZIONAMENTO
SUL
COMPLESSO
RIBOSOMIALE
DELL’AMINOACIL-tRNA APPROPRIATO
2) FORMAZIONE DEL LEGAME PEPTIDICO
3) SPOSTAMENTO DEL RIBOSOMA DI UN CODONE RISPETTO ALL’mRNA
IL RIBOSOMA CONTIENE UN SITO ATTIVO PER LA FORMAZIONE DI UN
LEGAME PEPTIDICO E DUE SITI CHE LEGANO I SUBSTRATI NECESSARI
PER LA REAZIONE (SITO PEPTIDILICO E SITO ACCETTORE).
IL SITO PEPTIDILICO (O SITO P) TRATTIENE LA CATENA POLIPEPTIDICA
IN FASE DI ALLUNGAMENTO ANCORA ATTACCATA MEDIANTE UN
LEGAME AMINOACILICO
AL tRNA DELL’ULTIMO AMINOACIDO
AGGIUNTO.
IL
SITO
ACCETTORE
(SITO
A)
LEGA
L’AMINOACIL-tRNA
DEL
SUCCESSIVO AMINOACIDO CHE DEVE ESSERE AGGIUNTO ALLA
CATENA.
GLI ANTICODONI DEI tRNA IN AMBEDURE I SITI, P E A, FORMANO
LEGAMI IDROGENO CON I CODONI DELL’mRNA.
ALL’INIZIO DEL CICLO IL SITO A E’ VUOTO MENTRE IL SITO P E’
OCCUPATO DAL tRNA DELL’ULTIMO AMINOACIDO AGGIUNTO ALLA
CATENA. QUESTO tRNA (DETTO PEPTIDIL-tRNA) E’ LEGATO ALLA
CATENA POLIPEPTIDICA NASCENTE.
IL tRNA DELL’AMINOACIDO SUCCESSIVO SI LEGA INNANZITUTTO CON
IL FATTORE DI ALLUNGAMENTO EF-TU.
EF-TU E’ UN MONOMERO CON UN SITO DI LEGAME PER IL GTP.
EF-TU:GTP-AMINOACIL-tRNA FORMANO UN COMPLESSO TERNARIO CHE
SI ADATTA AL SITO A DEL RIBOSOMA.
EF-TU RICONOSCE LA STRUTTURA DELLO STELO ACCETTORE DEL tRNA
(LEGA TUTTI GLI AMINOACIL-tRNA ECCETTO L’N-FORMILMETIONILtRNAiMet).
UNA DELLE FUNZIONI DI EF-TU:GTP E’ LA STABILIZZAZIONE DEL
LEGAME ESTERE NELLA POSIZIONE 3’ DELL’AMINOACIL-tRNA.
SE L’APPAIAMENTO TRA L’ANTICODONE DELL’AMINOACIL-tRNA DEL
COMPLESSO TERNARIO E IL CODONE CHE SI TROVA NEL SITO A E’
CORRETTO, SI HA L’IDROLISI DEL GTP IN GDP+Pi AD OPERA DI DUE
PROTEINE RIBOSOMIALI (L7 E L12).
LA COMPONENTE PEPTIDILTRANSFERASICA DELLA SUBUNITA’ 50S DEL
RIBOSOMA CATALIZZA IL TRASFERIMENTO DEL GRUPPO PEPTIDILICO
DAL PEPTIDIL-tRNA CHE SI TROVA NEL SITO P AL GRUPPO AMINICO
DELLA MOLECOLA DI AMINOACIL-tRNA CHE SI TRIVA NEL SITO A,
ALLUNGANDO COSI’ LA CATENA PEPTIDICA DI UN RESIDUO.
LA MOLECOLA DI tRNA DEAMINOACILATA VIENE ESPULSA DAL SITO P.
IL PEPTIDIL-tRNA APPENA SINTETIZZATO SI SPOSTA DAL SITO A AL
SITO P MENTRE IL RIBOSOMA SI SPOSTA VERSO IL CODONE
SUCCESSIVO. UNA NUOVA MOLECOLA DI AMINOACIL-tRNA OCCUPA IL
SITO A VACANTE E IL CICLO PUO’ COSI’ RICOMINCIARE.
IL GRUPPO AMINICO LIBERO DELL’AMINOACIL-tRNA FORMA UN
LEGAME PEPTIDICO CON IL CARBONIO CARBONILICO DELLA CATENA
PEPTIDICA NASCENTE.
L’ATTIVITA’
PEPTIDILTRANSFERASICA,
RESPONSABILE
DELLA
FORMAZIONE DEL LEGAME PEPTIDICO, E’ LOCALIZZATA SU ALMENO 5
PROTEINE DELLA SUBUNITA’ 50S DEL RIBOSOMA E SULL’rRNA 23S.
L’ENERGIA NECESSARIA ALLA FORMAZIONE DEL LEGAME PEPTIDICO
VIENE FORNITA DALL’IDROLISI DEL LEGAME ESTERE AD ALTO
CONTENUTO DI ENERGIA TRA LA CATENA PEPTIDICA NASCENTE E
L’OH IN 3’ DEL RIBOSIO DEL tRNA.
LO SCORRIMENTO DEL RIBOSOMA DI UN CODONE IN DIREZIONE 3’ (IN
CONCOMITANZA CON L’ESPULSIONE DEL tRNA DEAMINOACILATO E
CON LO SPOSTAMENTO DEL PEPTIDIL-tRNA DAL SITO A AL SITO P)
PRENDE IL NOME DI TRASLOCAZIONE.
LA TRASLOCAZIONE RICHIEDE L’INTERVENTO DI UNA PROTEINA
CHIAMATA EF-G CHE LEGA ANCH’ESSA IL GTP. L’IDROLISI DI QUESTO
GTP PERMETTE A EF-G DI OPERARE L’ESPULSIONE DEL tRNA
DEAMINOACILATO E LO SPOSTAMENTO DEL PEPTIDIL-tRNA DAL SITO A
AL SITO P.
TRE FATTORI DI RILASCIO, RF-1, RF-2 E RF-3 PARTECIPANO ALLA
CONCLUSIONE DELLA SINTESI PROTEICA. QUANDO NEL SITO A VIENE A
TROVARSI UNO DEI TRE CODONI DI STOP (UGA, UAG, UAA) LA SINTESI
PROTEICA HA TERMINE. INFATTI IL SITO A NON PUO’ ESSERE
OCCUPATO DA NESSUN AMINOACIL-tRNA (NON ESISTONO ANTICODONI
COMPLEMENTARI PER I 3 CODONI DI STOP) MA VIENE OCCUPATO DAI
FATTORI DI RILASCIO:
RF-1 RICONOSCE UAA E UAG
RF-2 RICONOSCE UAA E UGA
RF-3 SI LEGA CON IL GTP E SI ASSOCIA, POI, CON RF-1 O RF-2
I COMPLESSI RF-3:GTP-RF-1 E RF-3:GTP-RF-2 ALTERANO L’ATTIVITA’
DELLA
PEPTIDILTRANSFERASI
DEL
RIBOSOMA,
FACENDOLE
IDROLIZZARE IL LEGAME ESTERE DEL PEPTIDIL-tRNA. IL POLIPEPTIDE
VIENE COSI’ RILASCIATO NEL CITOPLASMA.
SINTESI PROTEICA NEGLI EUCARIOTI
LE TRE FASI DELLA SINTESI PROTEICA (INIZIO, ALLUNGAMENTO
DELLA CATENA E FINE) SONO SIMILI NEI PROCARIOTI E NEGLI
EUCARIOTI.
LE DIFFERENZA PIU’ MARCATE SI HANNO NELLA FASE D’INIZIO,
PERCHE’ L’mRNA EUCARIOTICO PRESENTA ALL’ESTREMITA’ 5’ IL
CAPPUCCIO DI 7-METILGUANINA. INOLTRE NEGLI EUCARIOTI L’mRNA
FORMA
STRUTTURE
RICOPERTE
DA
SECONDARIE
SVARIATE
ESTESE
PROTEINE.
CHE
VARI
VENGONO,
FATTORI
POI,
D’INIZIO
RIMUOVONO QUESTE PROTEINE E PERMETTONO COSI’ L’INIZIO DELLA
TRADUZIONE.
LA MAGGIOR PARTE DEGLI mRNA EUCARIOTICI HANNO UN UNICO SITO
D’INIZIO SITUATO VICINO AL CAPPUCCIO DI 7-METIL-GUANOSINA.
L’INTERAZIONE DELLA SUBUNITA’ MINORE DEL RIBOSOMA CON IL
CAPPUCCIO DI 7mG RICHIEDE UN GRUPPO DI PROTEINE CHIAMATE eIF4
DOPO AVER RICONOSCIUTO IL CAPPUCCIO 7mG (GRAZIE A Eif4F) LA
SUBUNITA’ MINORE DEL RIBOSOMA COMINCIA A SCORRERE LUNGO
L’mRNA FINO A RAGGIUNGERE IL CODONE D’INIZIO AUG (SITUATO A
NON PIU’ DI CIRCA 100 NUCLEOTIDI DAL CAPPUCCIO 7mG). IL
RICONOSCIMENTO DEL CODONE D’INIZIO VIENE FACILITATO DA
SEQUENZE SPECIFICHE AD ESSO VICINE: SEQUENZE DI KOZAK.
A QUESTO PUNTO ARRIVANO IL Met-tRNAiMet E LA SUBUNITA’
RIBOSOMIALE MAGGIORE E LA TRADUZIONE PUO’ COSI’ COMINCIARE.
QUESTO
MECCANISMO
D’INIZIO
IMPEDISCE
AL
RIBOSOMA
DI
RICONOSCERE CODONI AUG ALL’INTERNO DELL’mRNA. PERCIO’ TUTTE
LE MOLECOLE DI mRNA NEGLI EUCARIOTI CODIFICANO PER UN SOLO
POLIPEPTIDE E SONO DEFINITE MONOCISTRONICHE.
TUTTAVIA
ALCUNI
mRNA
VIRALI,
CHE
VENGONO
TRADOTTI
DALL’APPARATO DELLE CELLULE EUCARIOTICHE INFETTE, SONO PRIVI
DI CAPPUCCIO DI 7mG. IN QUESTI CASI LA TRADUZIONE VIENE
INIZIATA A LIVELLO DI SITI INTERNI PER L’INGRESSO DEI RIBOSOMI
(IRES, INTERNAL RIBOSOME ENTRY SITES) CON UN MECCANISMO NON
ANCORA BEN CHIARO.
COSTO ENERGETICO DELLA SINTESI PROTEICA
PER OGNI AMINOACIDO AGGIUNTO ALLA CATENA POLIPEPTIDICA
VENGONO SCISSI 4 LEGAMI FOSFODIESTERE AD ALTA ENERGIA:
ATP
AMP + PP
P+P
(SINTESI AMINOACIL-tRNA)
GTP
GMP + PP
(EF-TU)
GTP
GMP + PP
(EF-G)
COSTO ENERGETICO MOLTO ELEVATO: IN E. COLI
LA SINTESI
PROTEICA UTILIZZA DAL 30 AL 50% DELL’ATP TOTALE
LA VELOCITA’ DI SINTESI DI UNA PROTEINA E’ DI CIRCA 3-5
AMINOACIDI ADDIZIONATI AL SECONDO.
PICCOLE PROTEINE DI 100-200 AMINOACIDI VENGONO SINTETIZZATE IN
CIRCA UN MINUTO.
LA SINTESI DELLA PROTEINA PIU’ GRANDE ATTUALMENTE NOTA, LA
TITINA
FORMATA
DA
30.000
AMINOACIDI,
RICHIEDE
2-3
ORE.