THE S CHECKPOINT
S arrest
unreplicated DNA
1
Durante la fase S il DNA viene replicato
E’ importante controllare:
l’inizio della replicazione del DNA
il completamento della replicazione
che avvenga un solo ciclo di replicazione del
DNA
la presenza di eventuali danni
2
Danni al DNA dovuti a
replicazione incompleta
Danni al DNA dovuti a
eccesso di replicazione
3
Meccanismo di controllo per evitare la
rireplicazione del DNA
4
Le origini di replicazione vengono attivate una volta
sola in ogni ciclo cellulare
Esportazione
dal nucleo
ORC (origin recognition complex) si
lega ad una sequenza di 11 bp
caratteristica delle origini di
replicazione in S. cerevisiae
P
P
Cdc6
Ubiquitinazione
e degradazione
Durante la G1 altre proteine si
associano ad ORC formando il
complesso di pre-replicazione
SPFs (Cdc28-Clb5,6):
fosporilano la chinasi eterodimerica Cdc7-Dbf4 che induce i complessi di
replicazione ad iniziare la sintesi di DNA;
inibiscono l’assemblaggio di nuovi complessi di pre-replicazione
5
Anche l’ingresso e l’uscita
dalla Mitosi sono punti di
controllo importanti
6
La presenza di DNA non
impedisce l’ingresso in
replicato
Mitosi
Recentemente sono stati isolati mutanti di lievito difettivi
in questo punto di controllo, ma le molecole coinvolte
(inibitori di MPF) sono tuttora sconosciute
7
Effetto della uscita prematura dalla
Mitosi (anafase anticipata)
Trisomia del cromosoma 21
Sindrome di Down
8
DNA Damage Checkpoints:
G1 and G2 arrest
9
DNA-damage checkpoint
Punto di controllo attivato da un danno al genoma
E’ realizzato da un apparato di sorveglianza che rapidamente
ed efficientemente riconosce anche un singolo danno di piccola
entità avvenuto in un punto qualsiasi del genoma
Il danno al DNA può essere determinato:
• da stimoli esterni (es. agenti genotossici, UV, IR)
• da stimoli endogeni (es. produzione di ROS dal normale
metabolismo cellulare)
La cellula può fermarsi nella fase G1 o nella fase G2 del ciclo
cellulare
10
Varie componenti che controllano il ciclo
cellulare a livello di G1/S o G2/M sono geni
oncosoppressori
11
DNA
Damage
p53
p53
Transient cell cycle arrest
Repair
p53
Re-entry in the
cell cycle
p53
Apoptosis
12
Upstream and downstream
Upstream
Signal transduction
pathways
+ ..
+
p53 functions as
sensor of upstream
signals reflecting
DNA-damage
/cellular stress
p53
Downstream
+
.
.
activation
13
p53
Guardian of the Genome
p53 signaling is normally “off”
Activated by cell stress or
damage
p53 shuts down the multiplication
of stressed cells and induces
apoptosis
14
Signaling that activates p53
DNA damage (double strand breaks)
Aberrant growth signals (such as
overactivation of ras or myc)
Drugs (chemotherapeutics) and UV light
All 3 pathways inhibit the degradation of
p53, allowing it to activate transcription
of genes that induce cell death or inhibit
cell division
15
p53 Blocks the Cell
Cycle if DNA is
Damaged
There are two DNA damage
checkpoints: late G1 and
late G2.
Note that a block to p53
proteolysis increases p53
levels.
p53 mutations occur in at
least half of all cancers.
16
p53 tumor suppressor is downregulated by Mdm2
p53
p53
Blocks the ability of p53 to activate
transcription.
Mdm2
Serves as a ubiquitin ligase (E3) that
promotes p53 degradation.
Involved in the nuclear export of p53
Mdm2
Cell cycle arrest
Apoptosis
Inhibition of tumor growth
Activation and increase of p53
p53
p53
p53
p53
The mdm2 gene has been found amplified
overexpressed in many human malignancies.
or
17
G and S Phases of the Cell Cycle
1
18
G2 and M Phases of the Cell Cycle
19
The p53 Signaling Pathway
20
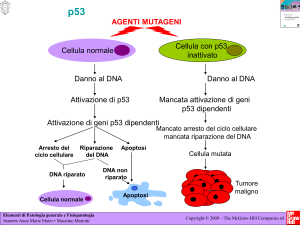
Il danneggiamento del DNA attiva p53
L’esito finale dipende dalla
fase del ciclo in cui si trova la
cellula:
Nelle fasi iniziali, p53
attiva il blocco in G1 o G2 fino
a che non è stato riparato il
danno
Se la fase è tardiva per
effettuare il blocco, p53
scatena l’apoptosi.
21
La proteina p53 wt è necessaria per limitare la crescita
cellulare.
La sua sua attività può andare perduta per delezione di
entrambi gli alleli o per una mutazione dominante in un
allele
22
Caratteristiche biochimiche di p53
Il gene p53 è localizzato sul cromosoma umano 17 ed è
composto di undici esoni, per una lunghezza complessiva di
circa 20 Kb.
Questo gene è espresso praticamente in tutti i tessuti
ed è altamente conservato in tutte le specie di vertebrati.
Il prodotto proteico di questo gene è una fosfoproteina
nucleare formata da 393 amminoacidi (PM 53 Kd) che è
coinvolta nella regolazione della proliferazione cellulare.
23
p53 domains
C-terminal allosteric domain
TAD
Binds to MDM2
DNA contact in major and
minor grove
Most of the p53 mutations that cause cancer are found in the DNA-binding
domain
–most common mutation changes arginine 248 (red), snaking into the minor
groove of the DNA - a strong stabilizing interaction.
–Other key sites of mutation are shown in pink, including arginine residues 175,
249, 273 and 282, and glycine 245. Some of these contact the DNA directly,
and others are involved in positioning other DNA-binding amino acids. 24
p53 domains
•binds DNA as tetramer (dimer of dimer)
•DNA recognition sequence reflects this: 4x RRRCW arranged like this:
C-terminal allosteric domain
•Tetramerization domain aa 324-355
–2β + 2α structure
–forms tetramers
–linked with DBD via 37aa flexible
linker [aa 287-323]
25
The human p53 protein
Mutations in human tumors that inactivate the function of p53
Structural organization of the p53 protein. Phosphorylation by various
kinases at the sites indicated by P stabilize p53. MDM2 protein binds at the
indicated site and represses transcription activation by p53 as part of the
normal control of p53 function. The activity of p53 also is inhibited by
binding of viral proteins such as E6 from human papillomavirus and E1b from
adenovirus
26
Stato conformazionale e funzione
Un fattore che influenza lo stato conformazionale della proteina p53, e quindi
la sua funzione biologica, è il suo stato di fosforilazione.
Varie chinasi, tra cui la Cdk1 e la proteina-chinasi C, aggiungono gruppi
fosfato ai vari residui di serina della p53.
La fosforilazione aumenta la capacità dei dimeri di p53 in forma latente di
legarsi al DNA
Conformazione attiva o latente?
La conversione da una forma all’altra dipende da modificazioni del sito
regolatorio carbossi-terminale.
Si è visto che utilizzando un anticorpo monoclonale, il PAb421, che si lega al
dimero p53 in
corrispondenza del sito bersaglio della protein-chinasi C si
passa da uno stato latente a uno stato attivo.
27
p53: dimero regolato in modo allosterico
Normalmente
attivata con
si
trova
in una forma latente che poi
può essere
un meccanismo ATP-dipendente dalla proteina-chinasi
C.
Lo stato
di
attivazione
è
reversibile,
ciò
è
di
enorme
importanza dal punto di vista terapeutico
Caratteristiche dei mutanti p53
Concentrazione intracellulare più elevata
Conformazione diversa
Non si legano in modo efficace al DNA
28
Effect of mutation of p53 on G1 DNA-damage checkpoint control
By 8 hours following exposure of wild-type cells to γ-radiation, cells that were in the
S phase (red shading) had completed DNA synthesis, entered G2, and then arrested,
accounting for the rise in the G2 peak. The absence of S-phase cells indicates that
irradiation prevented new cells from entering the S phase, causing them to arrest in
G1. (b) The presence of an S peak 8 hours after irradiation of p53 mutant cells
indicates that the G1 checkpoint does not operate in these cells. The increase in the
G2 peak indicates that the checkpoint blocking entry of irradiated cells into mitosis
still operates in these mutant cells.
29
Effect of ionizing radiation on wt and p53-cells
30
p53 e morte cellulare programmata
31
p53 mutata e carcinogenesi
Il gene p53 è uno dei geni che si trova più frequentemente
mutato nei tumori umani.
In alcuni casi la perdita della funzione oncosoppressiva della
p53 è un evento tardivo associato con la progressione del
tumore da benigno a maligno; in altri casi, è un evento precoce
dello sviluppo tumorale.
32
Classificazione delle cellule tumorali con
mutazione nel gene p53
Cellule tumorali che non esprimono affatto la proteina
p53
Cellule che hanno una delezione ereditaria di un allele
p53 e una mutazione somatica nel secondo allele
Cellule che esprimono un mutante p53 dominantenegativo
Mutazioni che portano ad un “acquisto di funzione” da
parte della proteina p53
33
Percentuale di mutazione del gene p53 in
vari tumori umani
34
Geni regolati positivamente da p53
Gene MCK (muscle creatine kinase)
Oncogene MDM2 (codifica per una proteina capace
di formare un complesso stabile con la p53)
Gene c-erbA (codifica per il recettore dell’ormone
tiroideo)
Gene Thy-1 (marker di differenziamento)
Gene GADD45 (indotto da danni al DNA)
Gene p21 (codifica per una proteina inibitrice delle
chinasi ciclina-dipendenti)
35
An overwiew on P53 role
The p53 protein is essential for the checkpoint control
that arrests human cells with damaged DNA in G1.
Replication of such cells would tend to perpetuate
mutations.
p53 functions as a transcription factor to induce
expression of p21, an inhibitor of G1 Cdk-cyclin
complexes.
Normal cell [p53] is low. Is degraded and replenished
regularly
Because p53 is a tetramer, a point mutation in one p53
allele can be sufficient to inhibit all p53 activity.
36
An overwiew on P53 role
If DNA is damaged then increase in [p53] stops
cell division till DNA repaired. If damage can’t be
repaired apoptosis (programmed cell death)
p53 gene damage increase in cancers
p53 damage is not inherited but acquired by exposure
to mutagens eg benzopyrene – cigarettes
p53 – associated with more then 60% of all known
cancers
MDM2, a protein that normally inhibits the ability of
p53 to restrain the cell cycle or kill the cell, is
overexpressed in several cancers.
37
LE BASI GENETICHE
DEL CANCRO
ONCOGENI E
ONCOPROTEINE
38
I geni coinvolti nello
sviluppo
del cancro sono geni
coinvolti
nel controllo della
PROLIFERAZIONE
CELLULARE
39
CONTROLLO DELLA PROLIFERAZIONE CELLULARE
DIRETTO
INDIRETTO
AGISCE SUI PUNTI DI
CONTROLLO CELLULARE
(G1/S, G2, M)
•DIFFERENZIAMENTO
•SENESCENZA
•APOPTOSI
GENI COINVOLTI
PROMOTORI DELLA
PROLIFERAZIONE CELLULARE
INIBITORI DELLA
PROLIFERAZIONE CELLULARE
MUTAZIONI GENICHE
Iperattività del gene,
mutazione dominante:
il gene mutato induce
tumori
⇒
ONCOGENE
Prodotto genico inattivo,
proliferazione cellulare,
mutazione recessiva
GENE SOPPRESSORE DI TUMORI
40
(Rb e p53)
Geni soppressori dei tumori e oncogeni
41
“Gatekeeper” genes
• The “gatekeeper” gene monitor cell proliferation
and death. They include:
– “oncogenes” which are cell growth promoters,
where
dominant
mutations
lead
to
uncontrolled growth, and
– “tumor suppressor” genes which are negative
regulators of cell growth, where recessive
mutation can lead to uncontrolled cell growth
(e.g., p53).
42
“Caretaker” genes
• The “caretaker” genes include genes that regulate
DNA repair and chromosome segregation in mitosis.
Mutation of of these genes can cause abnormal
gatekeeper function because of mutation or
abnormal gene content.
The “caretaker” genes
include:
– DNA repair genes, whose mutations
genetic instability (more mutations), and
lead
to
– genes that regulate chromosome segregation,
whose mutations lead to abnormal chromosome
content, or chromosomal instability.
43
Proto-oncogenes
Proto-oncogenes (c-onc) are genes that possess
normal gene products and stimulate normal cell
development.
Oncogenes
Oncogenes arise from mutant proto-oncogenes.
Oncogenes are more active than normal or active at
inappropriate times and stimulate unregulated cell
proliferation.
44
Some tumor viruses that infect cells possess
oncogenes (v-onc):
•RNA tumor viruses = possess viral oncogenes
(derived form cellular proto-oncogenes) capable
of transforming cells to a cancerous state.
•DNA tumor viruses = another class of tumor
viruses; do not carry oncogenes, but induce
cancer by activity of viral gene products on the
cell (no transformation per se).
45
Conversione di un
proto-oncogene in
oncogene
Effetti della
trasformazione di un
proto-oncogene in
oncogene
46
Trasformazione da proto-oncogene (c-onc) a oncogene
MECCANISMI
Amplificazione genica
Riarrangiamento
cromosomico delle
regioni di regolazione
Mutazioni
Delezioni
Inserzioni
Traslocazioni
EFFETTI
Alterazione quantitativa
Alterazione qualitativa
47
Kinds of Tumor-promoting Mutations
Defects in the regulation through the cell cycle can
contribute to cancer (leads to overproliferation of cells)
WT Function
Mutation
Promotes cell division
Oncogene (gain-of-function)
Inhibit cell cycle
Tumor suppressor gene (loss-of-function)
Promotes cell death (apoptosis)
Tumor suppressor gene (loss-of-function)
Inhibit apoptosis
Oncogene (gain-of-function)
Promote DNA repair
Tumor suppressor gene (loss-of-function)
48
Oncogenes as Signal Transducers
EXTRACELLULAR
Growth Factors
v-sis, int-1, int-2, hst, fgf-5
Growth Factors Receptors
C
Y
T
O
P
L
A
S
M
NUCLEUS
v-erb-B, v-fms, v-kit, v-ros,
Signal Transducers
v-ras, v-src, v-raf/mil, v-abl, v-mos, v-crk
Transcription Factors
v-ets, v-myc, v-myb, v-rel, v-ski, v-erb-A49
Le principali classi di proteine oncogeniche
CLASSE
Protoncogèni
CLASSE I
sis, fgf, int1
int2 …
CLASSE II
CLASSE III
CLASSE IV
Fms, erbB, neu,
ros
Famiglia ras,
Src, abl,yes
Mos,raf.
myc, fos,
myb, erb-A, ..
Funzione
proteica
Localizzazione
Fattori di crescita
Extracellulare
Recettori
Versante esterno e
interno della membrana
Trasduttori
intracellulari (ras, crk,
Proteine Tyr-chinasi
Proteine Thr/Serchinasi)
Membrana, citoplasma
Fattori di trascrizione
Nucleo
50
Normal cell cycle is controlled by signal
transduction:
Growth factors bind to surface receptors on the cell;
Transmembrane proteins relay signals into the cell.
Two types of growth factors:
Growth factors
stimulate cell division
Growth-inhibiting factors
inhibit cell division
Healthy cells divide only when growth factor and growthinhibiting factor balance favors cell division
Cancer cells divide without constraint (e.g., mutations in
growth and growth-inhibiting factor genes)
51
Regulation of cell division by signal transduction
52
Growth Factors as Oncogenes
v-sis carried by simian sarcoma virus is derived from
PDGF,and c-sis is amplified in glioma.
int-2 was identified as a MMTV integration is related to
FGF and is amplified in human breast carcinoma.
hst was isolated from stomach cancer and is amplified
(along with int-2) in head and neck tumors.
TGF-α (transforming growth factor-α) binds EGFreceptor
53
Growth Factor Receptors as Oncogenes
I.
Epidermal Growth Receptor Family:
Erb 2 in breast cancer
EGF receptor in glioblastoma multiforme
Neu in neuroblastoma
II.
Ret Oncogene:
involved in multiple endocrine neoplasm type
2 (MEN2)
•
Chimeric receptors found in leukemias:
NPM-ALK
TEL-PDGFR
54
Structural Change in Acquired vOnc
c-Erb B (EGFR)
v-Erb B
Epidermal growth factor Transduced retroviral
receptor
version
Ligand binding
domains
Viral gag
membrane
Kinase
domain
P
P
P
P
P
Regulatory
domain
P
P
P
P
Altered v-Erb B
functions as
constitutively
activated EGFR
55
Signal Transducers as Oncogenes
Non-receptor Tyrosine kinases
BCR-ABL and TEL-ABL (Philadelphia
Chromosome)
TEL-JAK
Src-family
Ras Family of G-proteins
Cytoplasmic Kinasesmil/Raf
56
Activating Ras Mutations
p21ras
12,13
GTP Hydrolysis
**
GTP Exchange
59,61
***
**
*
GTP/GDP
Binding
Switch
Region
GTP
Structures from
Sprang S.R.,
Annu. Rev. Biochem 1997. 66:639-78
GDP
57
Transcription Factors as Oncogenes
c-Myc plays a role in many human cancers
Translocations (8;14) c-myc and Ig genes (µ, λ and k)
-Burkitt’s Lymphoma
-Low-grade follicular lymphomas (sometimes with
BCL-2)
-Diffuse large cell lymphomas
Amplifications of c-myc
-Breast carcinoma
-neuroblastoma (involve the related N-myc gene)
-Small cell lung cancer (involve the related L-myc)
58
Myc Target Genes
α -Prothymosin
ODC
Cdc25A
Cyclin D1
Unkown function
Polyamine biosynthesis
Cell cycle
Cell cycle
eIF-2
eIF4E
Protein Synthesis
Protein syntheis
ARF or p19
Cdc2
Cyclin A
Cyclin E
Apoptosis/Checkpoint control
Cell cycle
Cell cycle
Cell cycle
59
MAPPA DELLE PRINCIPALI VIE DI
SEGNALAZIONE CHE PORTANO AL CANCRO
NELLE CELLULE UMANE
60
Retroviral Oncogenes (partial list)
Oncogene(v-onc)
Prototype Retrovirus
src
Rous sarcoma virus
myc
Avian myelocytomatosis virus
erb A, B
Avian erythroblastosis virus
myb
Avian myeloblastosis virus
H-ras
Harvey rat sarcoma virus
K-ras
Kirsten murine sarcoma virus
abl
Abelson murine leukemia virus
fes
Feline sarcoma virus
sis
Simian sarcoma virus
61
DNA tumor viruses target
tumor suppressors
Virus
Gene Product
Cellular target
Adenovirus
E1A
E1B
Rb
p53
SV40
Polyomavirus
Large T antigen
Large T antigen
Middle T antigen
Rb, p53
Rb
Src, PI3K
Papillomavirus
E7
E6
E5
Rb
p53
PDGF receptor
62
Mechanism of Rb inactivation
E2F
Rb
E1A
T ag
E7
E2F
E1A
Transcription of
E2F responsive
genes
Release of Rb
cell cycle brake
Rb
• Investigation on mode of action of E1A lead to the
discovery of E2F transcription factor and its
interactions with Rb.
• Important for transcription of Adenovirus E2 gene
63
Model for pRB function
Repressed
pRB
pRB
E2F
E2F
Activated
Adenovirus
X
E1A pRB
E2F
TTTCGCGC GCGCGAAA
P
P pRB P
pRB
E2F
E2F
Cyclin/CDK
X
TTTCGCGC GCGCGAAA
E2F
Kovesdi et al. 1986, Cell 45
Chellappan et al. 1991, Cell 65
Bagchi et al. 1991, Cell 65
Chittenden et al. 1991, Cell 65
Bandara et al. 1991, Nature 351
TTTCGCGC GCGCGAAA
P
pRB
E1A pRB
E2F
P
P
P pRB P
E2F
TTTCGCGC GCGCGAAA
Cloning of E2F:
Helin et al. 1992, Cell 70
Kaelin et al. 1992, Cell 70
64
65
Mechanisms of p53 inactivation
p53
p53
p53
T ag
E6
E1B
Tag
p53
Ub Ub
Ub
p53
p53
E1B
Stabilizes p53 in
an inactive state
E6AP
E6
E6AP:
E3 Ub ligase
Converts p53
from activator to
repressor of
transcription
66
DNA tumor viruses are smart
Adenovirus
Human Papilloma Virus
Simian Virus 40
E1A pRB
E1B
p53
E7 pRB
E6
p53
p53
pRB
T
67