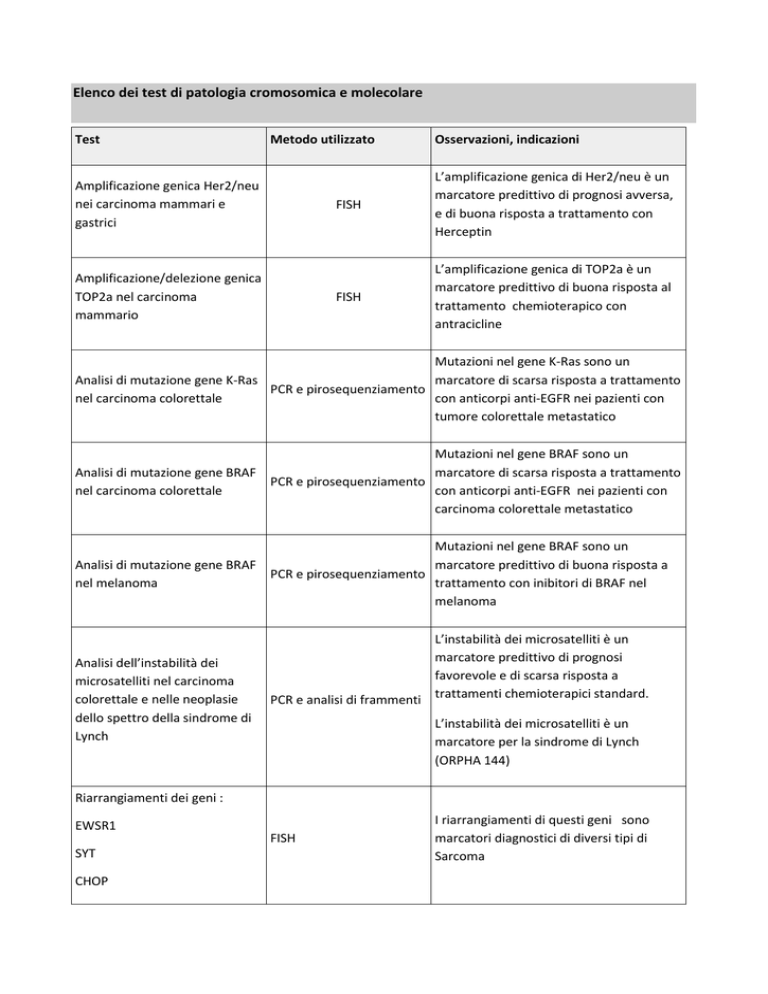
Elenco dei test di patologia cromosomica e molecolare
Test
Metodo utilizzato
Amplificazione genica Her2/neu
nei carcinoma mammari e
gastrici
Amplificazione/delezione genica
TOP2a nel carcinoma
mammario
Osservazioni, indicazioni
FISH
L’amplificazione genica di Her2/neu è un
marcatore predittivo di prognosi avversa,
e di buona risposta a trattamento con
Herceptin
FISH
L’amplificazione genica di TOP2a è un
marcatore predittivo di buona risposta al
trattamento chemioterapico con
antracicline
Mutazioni nel gene K-Ras sono un
Analisi di mutazione gene K-Ras
marcatore di scarsa risposta a trattamento
PCR e pirosequenziamento
con anticorpi anti-EGFR nei pazienti con
nel carcinoma colorettale
tumore colorettale metastatico
Mutazioni nel gene BRAF sono un
Analisi di mutazione gene BRAF
marcatore di scarsa risposta a trattamento
PCR e pirosequenziamento
con anticorpi anti-EGFR nei pazienti con
nel carcinoma colorettale
carcinoma colorettale metastatico
Analisi di mutazione gene BRAF
nel melanoma
Analisi dell’instabilità dei
microsatelliti nel carcinoma
colorettale e nelle neoplasie
dello spettro della sindrome di
Lynch
Mutazioni nel gene BRAF sono un
marcatore predittivo di buona risposta a
PCR e pirosequenziamento
trattamento con inibitori di BRAF nel
melanoma
L’instabilità dei microsatelliti è un
marcatore predittivo di prognosi
favorevole e di scarsa risposta a
PCR e analisi di frammenti trattamenti chemioterapici standard.
L’instabilità dei microsatelliti è un
marcatore per la sindrome di Lynch
(ORPHA 144)
Riarrangiamenti dei geni :
EWSR1
SYT
CHOP
FISH
I riarrangiamenti di questi geni sono
marcatori diagnostici di diversi tipi di
Sarcoma
FKHR
L’amplificazione di MDM2 e CDK4 è un
marcatore diagnostico per alcuni tipi di
sarcomi e Gliomi ed ha rilevanza
prognostica per i tumori GIST
Amplificazione di MDM2 e di
CDK4 nei sarcomi e nei Gliomi
FISH
Analisi di mutazione gene EGFR
nel carcinoma polmonare
Mutazioni nel gene EGFR sono un
PCR e pirosequenziamento marcatore predittivo di buona risposta a
trattamento con inibitori di EGFR
FISH
Il riarrangiamento nel gene ALK è
marcatore predittivo di risposta a
trattamento con inibitori di recettori
tirosin-chinasici
Riarrangiamento gene ROS1 nel
FISH
carcinoma del polmone
Il riarrangiamento nel gene ROS1 è
marcatore predittivo di risposta a
trattamento con inibitori di recettori
tirosin-chinasici
Riarrangiamento gene ALK nel
carcinoma polmonare e nei
linfomi
FISH
Il riarrangiamento nel gene c-MET è
marcatore predittivo di risposta a
trattamento con inibitori di recettori
tirosin-chinasici
FISH
Marcatore con significato prognostico nel
carcinoma polmonare
Amplificazione gene c-MET
nel carcinoma del polmone
Amplificazione gene EGFR
nell’adenocarcinoma del
polmone e nei Gliomi
Amplificazione gene FGFR1 nel
carcinoma squamo cellulare del FISH
polmone
Indicatore di risposta alla terapia con
inibitori di FGFR1
Metilazione del gene MGMT nei
PCR pirosequenziamento
Gliomi
La metilazione del promotore del gene
MGMT è un marcatore predittivo di
prognosi favorevole e di buona risposta a
trattamento con terapie alchilanti
Delezioni 1p, 19q, 17p, 10q nei
Gliomi
Delezioni dei cromosomi 1p, 19q, 17p, 10q
PCR e analisi di frammenti sono marcatori con significato diagnostico,
prognostico e predittivo nei gliomi
Mutazione del gene IDH1 nei
PCR pirosequenziamento
La mutazione del gene IDH1è un
marcatore con significato diagnostico e
gliomi
prognostico nei gliomi
Amplificazione del gene MYC
nei gliomi
FISH
L’amplificazione del gene MYC è un
marcatore con significato prognostico nei
gliomi
ISH
L’identificazione e la tipizzazione dei virus
HPV è un marcatore diagnostico e
prognostico nel carcinoma orofaringeo
PCR-ibridazione
L’identificazione e la tipizzazione dei virus
è un marcatore diagnostico per la
neoplasia intraepiteliale della cervice
uterina
FISH
Il riarrangiamento del gene CCND1 è un
marcatore diagnostico di linfoma
mantellare e un marcatore predittivo di
prognosi favorevole nel mieloma
plasmacellulare
Diagnosi HPV nel carcinoma
orofaringeo
Diagnosi HPV nelle neoplasie
intraepiteliali della portio
uterina
Riarrangiamento del gene
CCND1 nei Linfomi e nei
Mielomi multipli
Riarrangiamento del gene MYC
FISH
nei Linfomi
Il riarrangiamento del gene MYC è un
marcatore diagnostico di linfoma di Burkitt
e ha un significato prognostico nei linfomi
diffusi a grandi cellule B
Riarrangiamento del gene
MALT1 nei Linfomi
Il riarrangiamento del gene MALT1 è un
marcatore diagnostico dei linfomi tipo
MALT
FISH
Riarrangiamento del gene BCL2
FISH
nei Linfomi
Il riarrangiamento del gene BCL2 è un
marcatore diagnostico di linfoma
follicolare
Riarrangiamento del gene BCL6
nei Linfomi
FISH
Il riarrangiamento del gene BCL6 è un
marcatore prognostico dei linfomi B diffusi
a grandi cellule
Riarrangiamento gene ALK nei
Linfomi
FISH
Il riarrangiamento del gene ALK è un
marcatore diagnostico e predittivo di
prognosi favorevole
Riarrangiamento dei geni
TCRAD, TCRB, TCRG, TCL1
FISH
Questi riarrangiamenti sono marcatori
diagnostici di linfomi a cellule T
Riarrangiamento del gene IGH
nei Linfomi
FISH
Il riarrangiamento del gene IGH è un
marcatore diagnostico di Linfomi a cellule
B
Il riarrangiamento genico di IGH è un
Riarrangiamento genico IGH nei
PCR e analisi di frammenti marcatore diagnostico dei linfomi a cellule
Linfomi
B
Il riarrangiamento genico di TCRB e TCRG è
Riarrangiamento genico TCRB e
PCR e analisi di frammenti un marcatore diagnostico dei linfomi a
TCRG
cellule T
Delezione della regione 13q14
Delezione della regione ATM
Delezione della regione TP53
FISH su sangue periferico
e/o midollare
Anomalie di queste regioni cromosomiche
sono indicatori di diagnosi e di prognosi
nelle Leucemie Linfatiche Croniche
Trisomia del cromosoma 12
Traslocazione IGH/CCND1
Traslocazione IGH/FGFR3
Delezione della regione 13q14
FISH su cellule selezionate Anomalie di queste regioni cromosomiche
CD138+ da sangue
sono indicatori di diagnosi e di prognosi
midollare
nel Mieloma Multiplo
Delezione di TP53
Cariotipo su linfonodi, sangue
midollare, sangue periferico,
campioni di tumori solidi
Aneuploidie dei cromosomi 2,
7, 17 e delezione della regione
p16 nelle cellule del sedimento
urinario
Analisi citogenetica
convenzionale
La caratterizzazione di anomalie
citogenetiche identifica marcatori
diagnostici e prognostici in molte
neoplasie
FISH su cellule urinarie
Anomalie di queste regioni cromosomiche
sono indicatori di diagnosi di neoplasie
uroteliali
Mutazione ignota e nota dei
geni BRCA1 e BRCA2
PCR sequenziamento
Mutazione ignota e nota dei
geni MSH2, MLH1, MSH6
PCR sequenziamento
Analisi MLPA
Analisi MLPA
Le mutazioni dei geni BRCA1 e BRCA2
identificano la sindrome di suscettibilità
ereditaria ai tumori mammari e ovarici
(ORPHA145)
Le mutazioni dei geni MSH2,MLH1, MSH6
identificano la sindrome di Lynch
(ORPHA144)
Mutazione ignota e nota dei
geni APC e MutYH
PCR sequenziamento
Analisi MLPA
Le mutazioni dei geni APC e MutYH
identificano la Sindrome poliposica
famigliare (FAP ORPHA 733) e le poliposi
attenuate (AFAP e MAP ORPHA 247806 e
247798)
Mutazione ignota e nota di TP53 PCR sequenziamento
Le mutazioni del gene TP53 identificano la
Sindrome di Li Fraumeni (ORPHA524)
Mutazione ignota e nota di
PTEN
PCR sequenziamento
Le mutazioni del gene PTEN identificano la
sindrome di Cowden (ORPHA201)
Mutazione ignota e nota di
STK11 (LKB1)
PCR sequenziamento
Le mutazioni di STK11 (LKB1) identificano
la Sindrome di Peutz Jeghers (ORPHA2869)
Mutazione ignota e nota di
CDKN2A e CDK4
PCR sequenziamento
Le mutazioni di CDKN2A e CDK4
identificano la sindrome del Melanoma
famigliare (ORPHA618)
Mutazione ignota e nota di
PTCH1
PCR sequenziamento
Le mutazioni di PTCH1 identificano la
Sindrome di Gorlin (ORPHA377)
PCR sequenziamento
Le mutazioni di CDH1 identificano la
sindrome del cancro gastrico ereditario
(ORPHA26106)
Mutazione ignota e nota di
CDH1












