Diagnostica Biomolecolare:
Tecnologie e Applicazioni
Preparazione dei campioni:
(Estrazione del DNA o dell’RNA dal tessuto di interesse)
Analisi delle mutazioni:
SSCP – DHPLC – Dot blot - Southern - PCR (ARMS – Enzimi di
restrizione)
Real-Time PCR (Discriminazione allelica) - RT-PCR
Analisi HT
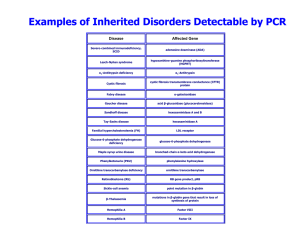
Examples of Inherited Disorders Detectable by PCR
Disease
Affected Gene
Severe-combined immunodeficiency,
SCID
adenosine deaminase (ADA)
Lesch-Nyhan syndrome
hypoxanthine-guanine phosphoribosyltransferase
(HGPRT)
α1-Antitrypsin deficiency
α1-Antitrypsin
Cystic fibrosis
cystic fibrosis transmembrane conductance (CFTR)
protein
Fabry disease
α-galactosidase
Gaucher disease
acid β-glucosidase (glucocerebrosidase)
Sandhoff disease
hexosaminidase A and B
Tay-Sachs disease
hexosaminidase A
Familial hypercholesterolemia (FH)
LDL receptor
Glucose-6-phosphate dehydrogenase
deficiency
glucose-6-phosphate dehydrogenase
Maple syrup urine disease
branched-chain α-keto acid dehydrogenase
Phenylketonuria (PKU)
phenylalanine hydroxylase
Ornithine transcarbamylase deficiency
ornithine transcarbamylase
Retinoblastoma (Rb)
RB gene product, pRB
Sickle-cell anemia
point mutation in β-globin
β-Thalassemia
mutations in β-globin gene that result in loss of
synthesis of protein
Hemophilia A
Factor VIII
Hemophilia B
Factor IX
Diagnosi attraverso l’analisi
molecolare
mRNA
DNA
Diagnosi attraverso l’analisi
molecolare
mRNA
a. RT-PCR
b. Protein Truncation Test (PTT)
a. RT-PCR
RT-PCR permette di evidenziare variazioni
nella lunghezza dell’mRNA (delezioni) oppure
modulazioni del livello dei trascritti (mutazioni
nei siti di regolazione dell’espressione).
Può essere vantaggiosa nel caso si analizzi un
gene di notevoli dimensioni (distrofina).
The dystrophin gene is the
largest gene found in nature,
measuring 2.4 Mb.
79 exons
L’analisi viene condotta operando inizialmente una reazione enzimatica di amplificazione del DNA,
conosciuta come Polymerase Chain Reaction (PCR), che consente di amplificare in vitro una
specifica regione della molecola, copiandola in varie fasi successive, fino ad ottenerne milioni di
copie.
In tale maniera vengono amplificati, mediante amplificazione genica fluorescente multipla , 18
esoni del gene della Distrofina, così ripartiti:
Il sistema, realizzato seguendo le linee guida del gruppo collaborativo europeo per la diagnosi
molecolare della Distrofia DMB/DMD (EMQN), permette di evidenziare circa il 98% delle delezioni
del gene della Distrofina.
Exon
exon 1
exon 3
exon 43
exon 50
exon 13
exon 6
exon 47
exon 60
exon 52
Size
(bp)
535
410
357
271
238
202
181
139
113
DYE
Exon
HEX
HEX
HEX
HEX
HEX
HEX
HEX
HEX
HEX
exon 45
exon 48
exon 19
exon 17
exon 51
exon 8
exon 12
exon 44
exon 4
Size
(bp)
547
506
459
416
388
360
331
268
196
DYE
FAM
FAM
FAM
FAM
FAM
FAM
FAM
FAM
FAM
b. Protein Truncation Test (PTT)
Il “PTT” è un test che permette di mettere in
evidenza la formazione di una proteina tronca.
Naturalmente non permetterà di conoscere il
tipo di mutazione che ha causato l’evento
PTT
“Protein Truncation Test”
È un test specifico per mutazioni frameshift,
nonsense, splice site che portano alla
produzione di una proteina tronca
(Neurofibromatosi, Distrofia di Duchenne)
Amplificazione del cDNA
Presenza al 5’:
• di un promotore,
• sito di inizio della trascrizione
• Una sequenza specifica che permetta il giusto frame di
lettura
Il prodotto di PCR viene messo in contatto con un
sistema di trascrizione e traduzione che costruisce il
polipeptide corrispondente, le cui dimensioni vengono
analizzate su gel di poliacrilamide con SDS.
Polipeptidi tronchi avranno dimensioni minori rispetto
a quelle di controllo.
Diagnosi attraverso l’analisi
molecolare
DNA
a. Mutazioni non necessariamente caratterizzate
b. Specifiche mutazioni
• Amplificazione
della
sequenza
da
analizzare (PCR)
• Individuazione dei prodotti mediante
sonde specifiche (Ibridazione)
a. Mutazioni non necessariamente caratterizzate
• Mobilità elettroforetica di heteroduplex
• Single-strand conformation polymorphism (SSCP)
• Denaturing gradient gel electrophoresis (DGGE)
• Mismatch cleavage
enzimatico
chimico
SSCP
Analisi di polimorfismi di DNA a singola elica
• Amplificazione mediante PCR del frammento di DNA
da analizzare.
• Denaturazione.
• Elettroforesi su gel di poliacrilamide non denaturante
del DNA da analizzare e di un DNA di controllo.
In presenza di mutazione la diversità di conformazione del
DNA a singola elica determinerà differenze nella velocità di
migrazione.
DGGE
“denaturing gradient gel electrophoresis”
DHPLC
“denaturing high performance liquid cromatography”
Sfruttano le proprietà del DNA eteroduplex, in gradiente di
denaturazione, per poter individuare sequenze che
differiscono anche per una sola base
Questi metodi non individuano la posizione
e il tipo di mutazione.
Non permettono di discriminare tra una
mutazione che provoca un’alterazione nel
prodotto proteico ed un semplice
polimorfismo del DNA.
Per conoscere quindi se la mutazione
messa in evidenza può causare un
fenotipo patologico occorre ricorrere al
sequenziamento
b. Specifiche mutazioni
• La mutazione che causa la malattia è
unica ( Falcemia )
• Effetto del fondatore o vantaggio degli
eterozigoti ( βTalassemia – Fibrosi cistica )
• La malattia è causata da uno specifico
meccanismo molecolare ( Corea di Huntington
– FraX )
b. Specifiche mutazioni
• Digestione con enzimi di restrizione di DNA amplificato
con PCR; controllo della grandezza dei frammenti su
gel
• Ibridazione di DNA amplificato
oligonucleotidi allele specifici (ASO)
con
PCR
ad
• Amplificazione di DNA usando primers allele specifici
(ARMS)
• PCR con primers localizzati agli estremi di una
delezione o di un breakpoint di traslocazione
• Individuazione del numero di triplette ripetute
Digestione con enzimi di restrizione di DNA
amplificato con PCR;
controllo della grandezza dei frammenti su gel
M
1
2
3
4
187 bp
165 bp
+-
+-
++
++
Analisi della mutazione N1303K (gene CFTR: 4041 C>G)
In questo caso la mutazione crea un sito di restrizione per l’enzima DdeI (CTNAG), perciò
l'enzima taglia il DNA con la mutazione
(+ presenza della mutazione; - mutazione assente;
M = marcatore di peso molecolare)
Scomparsa di un sito BclI nel gene del fattore VIII
Ibridazione di DNA amplificato con PCR ad
oligonucleotidi
allele
specifici
(ASO)
ARMS
“Amplification refractory mutation system”
(amplificazione allele specifica)
• Si eseguono due amplificazioni parallele.
• Uno dei primer è comune ad entrambe le
reazioni;
• l’altro in una reazione è specifico per
l’allele normale, nell’altra per l’allele
mutato
PCR allele specifica
Multiplex ARMS test per individuare 4 specifiche
mutazioni che causano la fibrosi cistica
PCR con primers localizzati agli estremi di una
delezione o di un breakpoint di traslocazione
Chromosomal rearrangements or translocations
Neoplasm
Translocation
Burkitt lymphoma
t(8;14) 80% of cases
t(8;22) 15% of cases
t(2;8)
5% of cases
c-myc1
Chronic myelogenous
leukemia
t(9;22) 90-95% of cases
bcr-abl2
Acute lymphocytic
leukemia
t(9;22) 10-15% of cases
bcr-abl2
1c-myc
Proto-oncogene
is translocated to the IgG locus, which results in its activated expression
fusion protein is produced, which results in a constitutively active abl kinase
2bcr-abl
34
La proteina, codificata da c-myc attiva la trascrizione di geni che
controllano la progressione del ciclo cellulare
dalla fase G1 alla fase S.
Normalmente, sia l’ mRNA trascritto dal protooncogene, che la
proteina sono molto instabili.
Le cellule il cui genoma contiene le mutazioni sono costitutivamente
indotte a proliferare.
35
sono tipiche le traslocazioni che interessano il cromosoma 8, dove è
localizzato il gene c-myc, ed uno dei tre cromosomi in cui sono
localizzati i geni che codificano per le catene pesanti e leggere delle
immunoglobuline.
La traslocazione più frequente è la [t (8:14)], che si riscontra nel
90% dei BL:
la traslocazione, spostando cMyc in prossimità dell’enhancer del
gene che codifica per una delle catene delle immunoglobuline, causa
la sua continua espressione e di conseguenza le cellule sono
costitutivamente stimolate a proliferare.
37
c-myc is translocated to the IgG locus,
which results in its activated expression
c-myc
IgG enhancer
IgG
c-myc is activated by
the IgG enhancer in
lymphocytes
bcr-abl fusion protein is produced,
which results in a constitutively active abl kinase
bcr
bcr-abl
abl
38
A chromosome translocation that forms
Bcr-Abl in a hematopoietic stem cell
forms the diagnostic “Philadelphia”
chromosome and results in the initial
chronic phase of human chronic
myelogenous leukemia (CML),
characterized by an expansion in the
number of well-differentiated
granulocytes, a type of white blood cell.
A second mutation in one such cell (e.g.,
in p53) leads to acute leukemia.
39
40
The chromosomal translocation results in fusion of a portion of the
bcr gene (whose function is unknown but whose N-terminal segment
forms a coiled-coil domain that links several bcr polypeptides
together) with part of the c-abl gene, which encodes a proteintyrosine kinase whose normal substrates are not known.
The chimeric polypeptides expressed from the resulting Bcr-Abl
oncogene form a tetramer that exhibits constitutive Abl kinase
activity.
Although Abl is normally localized to the nucleus, addition of the
Bcr segment causes the Bcr-Abl oncoprotein to be localized to the
cytosol.
Bcr-Abl binds to many intracellular signal-transduction proteins
and then phosporylates them, proteins that Abl would not normally
activate.
As a consequence, these signaling proteins become activated in
41 the
absence of growth factors.
42
X fragile
(309550)
Frequenza: 1/4000 maschi.
Ereditarietà: Legata al cromosoma X. Malattia causata da mutazione dinamica.
Genetica: Nel 1991 è stato identificato il gene responsabile. La mutazione è
caratterizzata dall’amplificazione di un tratto di DNA costituito da una
specifica sequenza ripetuta (CGG). Nei soggetti normali è presente un
numero di ripetizioni variabili da 6 a 55. Esistono due differenti tipi di
mutazione: la premutazione (56-200) e la mutazione completa (>200). La
probabilità di espansione aumenta con le dimensioni della premutazione e
quindi con il passare delle generazioni (Paradosso di Sherman).
Diagnosi: La diagnosi molecolare (Southern blot) permette di individuare anche
gli individui con la premutazione.
Diagnosi di malattie da espansione
di triplette ripetute
Real Time PCR
Per ogni campione da analizzare utilizzo 2
differenti sonde marcate con 2 differenti
fluorofori
1) Complementare all’allele “wild type”
2) Complementare all’allele mutato
e la stessa coppia di primers
Discriminazione allelica
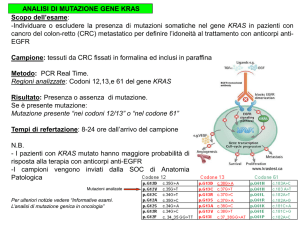
Le ricerche sono state condotte sul cetuximab, un anticorpo monoclonale utilizzato
per il trattamento del cancro del colon-retto metastatico ed è stato individuato un
gene (KRAS) che predice l'efficacia di questa molecola sul paziente.
I risultati mostrano che il cetuximab funziona meglio nei pazienti che non
presentano mutazioni in questo marcatore.
Questa scoperta è un altro decisivo passo avanti verso la messa a punto di terapie
sempre più mirate e su misura per il paziente.
Una mutazione puntiforme nel gene, che codifica per la
proteina Ras, riduce la sua attività GTPasica rendendola
costitutivamente attiva.
Cetuximab è un anticorpo
monoclonale che blocca il recettore
dell’EGF.
La proteina Ras si trova a valle di
EGFR, quindi anche bloccando il
recettore non si ottiene la risposta
desiderata (blocco della proliferazione
cellulare)
Ricerca delle mutazioni del gene K-Ras
nella biopsia del paziente
Codon
12
13
Mutation;
Aminoacid
change
Nucleotide substitution
GGT
TGT; G12C
G
T
GGT
GTT; G12V
G
T
GGT
GAT; G12D
G
A
GGT
GCT; G12A
G
C
GGT
AGT; G12S
G
A
GGT
CGT; G12R
G
C
GGC
GAC; G13D
G
A