COSA SONO LA FARMACOGENOMICA E LA FARMACOGENETICA
?
NON QUESTO !!!!
FARMACOGENOMICA: Applicazione delle tecnologie “genomiche” per la ricerca
di nuovi farmaci o per lo studio dell’azione di farmaci orfani
FARMACOGENETICA: Studio delle variazioni genetiche individuali che danno
luogo a differenti risposte alla assunzione di un farmaco
Uno degli aspetti più frustranti della
terapia con antidepressivi è la natura
“prova e sbaglia” della risposta clinica.
Ci vogliono spesso mesi di tentativi ed
errori prima di trovare farmaco e dose
efficace
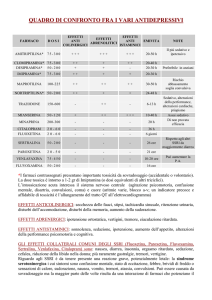
Il 25–40%
dei pazienti sotto
antidepressivi non rispondono alla loro
prima opzione farmacologica e tra i
rispondenti il 10–20% hanno effetti
collaterali clinicamente significativi
Una predizione su base genetica alla
suscettibilità di risposta potrebbe
aiutare a definire la scelta dell’agente
terapeutico e la dose migliore per
velocizzare la risposta e diminuire gli
effetti collaterali
Differenze di singole basi genomiche
sono la causa maggiore della variabilità
fenotipica tra gli uomini e quindi anche
della diversa risposta ai farmaci
Rappresentano circa lo 0,1% delle 3.300.000.000
basi nucleotidiche
Dopo la stessa dose di SSRI le concentrazioni plasmatiche possono variare
anche di 40 volte tra individui diversi.
Le varianti genetiche determinano
queste
differenze
nella
risposta
farmacocinetica……
...... Ed altre varianti modificano i parametri farmacodinamici
Farmacocinetica
Molte delle variazioni farmacocinetiche sono dovute a differenze nel
metabolismo dei farmaci a loro volta determinate da forme alternative
polimorfe dei geni che codificano alcuni enzimi epatici.
Il sistema meglio conosciuto è quello del citocromo P450
Alcuni enzimi P450 sono presenti anche
a livello delle pareti addominali, ma la
maggioranza sono negli epatociti dove
sono coinvolti nella inattivazione di
molti farmaci anche gli antidepressivi
Family
Function
Members
Names
CYP1
drug and steroid
(especially estrogen)
metabolism
3 subfamilies, 3
genes, 1 pseudogene
CYP1A1, CYP1A2,
CYP1B1
CYP2
drug and steroid
metabolism
13 subfamilies, 16
genes, 16
pseudogenes
CYP2A6, CYP2A7,
CYP2A13, CYP2B6,
CYP2C8, CYP2C9,
CYP2C18, CYP2C19,
CYP2D6, CYP2E1,
CYP2F1, CYP2J2,
CYP2R1, CYP2S1,
CYP2U1, CYP2W1
CYP3
drug and steroid
(including
testosterone)
metabolism
1 subfamily, 4 genes,
2 pseudogenes
CYP3A4, CYP3A5,
CYP3A7, CYP3A43
CYP4
CYP51
cholesterol
biosynthesis
Farmacocinetica
1 subfamily, 1 gene, 3
pseudogenes
6 subfamilies, 11
arachidonic acid or
genes, 10
fatty acid metabolism
pseudogenes
CYP51A1 (lanosterol
14-alpha demethylase)
CYP4A11, CYP4A22,
CYP4B1, CYP4F2,
CYP4F3, CYP4F8,
CYP4F11, CYP4F12,
CYP4F22, CYP4V2,
CYP4X1, CYP4Z1
Farmacocinetica
CYP2D6, CYP2C19, CYP3A4 e CYP1A2
sono gli enzimi più importanti per il
metabolismo degli antidepressivi
L’80%
degli
antidepressivi
sono
degradati
da
CYP2D6,
tranne
citalopram, roboxetina, moclobamide
ed amilsulpiride
La fluvoxamina viene
Ossidata da CYP1A2.
Un altro enzima CYP3A4, agisce sul
metabolismo di circa il 50% di
antidepressivi
(sertalina
e
norfluoxetina) ma non sono note
varianti importanti
Il gene CYP2D6 mostra
una enorme variabilità
genetica con
più
di
70
funzionali;
varianti
15 varianti inattive;
duplicazioni di alleli
attivi ed isoenzimi
iperattivi
In Europa il 7%
PM e 5% UM
della popolazione
è
La duplicazione ed espansione di copie
attive del gene CYP2D6 dona un
vantaggio evolutivo in condizioni di
ridotto apporto nutrizionale avendo un
aumentato valore di detossificazione di
alcuni componenti della dieta come la
frutta
Nell’Africa sub-Sahariana il fenotipo
UM è più frequente (10–20%) che nel
Nord Europa (5%)
La variabilità dei livelli plasmatici di
farmaco dopo la stessa dose orale sono
determinati quasi esclusivamente dal
genotipo CYP2D6
La maggioranza di effetti seri che si
hanno nel trattamento routinario con
SSRI è determinato in portatori con
copie inattive od iperattive di CYP2D6
Sarebbe raccomandabile ridurre il dosaggio del
30–70% nei PM ed aumentarlo del 135–180% agli
UM. Tuttavia, nessuno studio prospettico ha
dimostrato
la
superiorità
dell’aggiustamento
basato sul genotipo rispetto alla corrente pratica
clinica
Identificare i PM permetterebbe
inoltre di cambiare agente terapeutico,
selezionando farmaci che non sono
metabolizzati
dal sistema delle
CYPP450
L’identificazione
del
grado
di
metabolizzazione può essere fatto sia
con l’analisi genomica che con modalità
farmacologiche (test alla sparteina). Il
secondo approccio sembra più sensibile
FDA News
FDA Clears First of Kind Genetic Lab Test
Un uomo di 32 anni viene trattato con
venlafaxina
per
una
depressione
maggiore. La sua risposta alla dose
massima raccomandata di 225 mg/die
è solo parziale dopo un mese a questo
dosaggio.
La genotipizzazione dimostra che è
poratatore
di
varianti
multiple
CYP2D6*1 associate con uno stato UM.
La dose è aumentata a 375 mg/die con
un ritorno soddisfacente allo stato di
pre-morbidità.
La genotipizzaione di CYP2D6 per le varianti genetiche è anche importante
per il trattamento combinato con farmaci che interagiscono con lo stesso
sistema CYP
Parecchi SSRIs inibiscono CYP2D6 (forti inibitori sono la fluoxetina e la
paroxetina). La co-amministrazione di uno di questi farmaci può cambiare
lo stato di un individuo UM,IM,EM in PM, in particolare se il secondo
farmaco è metabolizzato esclusivamente da CYP2D6
Farmacocinetica
La barriera emato-encefalica contiene sistemi di trasporto di molte sostanze
dal sistema circolatorio al parenchima cerebrale e viceversa.
MDR1
(ABCB1/p-glycoprotein)
è
il
sistema di pompa meglio caratterizzato
che tende a riassorbire il farmaco dal
SNC.
Polimorfismi in MDR1 sono stati associati a differenze
nell’accesso di alcuni farmaci antidepressivi nei siti di azione a
livello del SNC
Certi individui hanno sistemi di pompa più rapidi e meno
farmaco riuscirà a penetrare nel SNC. Questi tenderanno
ad avere meno effetti sul SNC dovuti al farmaco anche di
fronte ad elevati livelli ematici
Farmacodinamica
SERT ricicla la serotonina alla
sinapsi.
La sua inibizione aumenta i livelli di
neurotrasmettotore
Farmacodinamica
Nell’uomo vi è un unico gene Sert posizionato sul cromosoma 17
Farmacodinamica
Gli alleli 5HTTLPR sono composti da 14
(“short”o “S”) o 16 (“long o “L”)
elementi ripetuti (da 20-23 bp).
Polimoirfismo al promotore
(5HTTLPR)
Esistono anche alleli “super-long”, XL e
XXL,
con fino a 20 ripetute, e
varianti con inserzioni, delezioni, o
sostituzioni ento le ripetute
La
popolazione
caucasica
mostra
frequenze alleliche del 57% per L e
43% per con genotipi 5HTTLPR
32% LL, 49% LS, and 19% SS
Gli Asiantici hanno
frequenze di SS
il
doppio
delle
Farmacodinamica
La variante S causa una diminuzione dell’espressione genica ed
una risposta minore all’attività SSRI comparata con la
variante L
I riscontri di associazione clinica tra genotipo e
risposta alla terapia sono controversi:
Una meta-analisi recente su tutti i trials fino al
2006 ha suggerito che il genotipo SS sia associato a
ridotta remissione rispetto as LL dopo trattamento
con SSRI
I due studi su 9 dove non si aveva
associiazione era a carico della popolazione
Giapponese e Coreana dove le frequenze del
genotipo LL è il 7% comparato al ~30% nei
Caucasici
Un incidenza di 5,3 volte più elevata di ipomania come effetto collaterale
di SSRI si è osservato in individui SS rispetto ad altri genotipi
Tendenza suicidaria
SS ed all’allele S
dopo trattamento è stata anche aasociata al genotipo
E’ stato anche associato un aumento di 3,5 volte per insonnia (78% vs 22%) e
9.5 volte di agitazione (67% vs. 7%) dopo trattamento con fluoxetina in
individui SS rispetto ad individui LL
I riscontri di associazione clinica tra
genotipo e risposta alla terapia sono
controversi:
Lo studio più grosso tuttavia (STAR*D)
non conferma queste associazioni in una
popolazione in trattamento con citalopram
Esiste una variante SNP nell’allele L che ne riduce
il funzionamento a livelli comparabili ad un S
Un altro SNP rs25531, nelle sequenze upstream
sembra avere rilevanza funzionale
ALTRE VARIANTI FUNZIONALI
Una delezione di ~380bp [del(17)(q11.2)] tra il 5HTTLPR e l’inizio di
trascrizione, che sembra instabile in vivo e conferisce un mosaicismo
nell’espressione del trasportatore
ALTRE VARIANTI FUNZIONALI
Una VNTR nell’introne 2 che contiene
9, 10, o 12 copie di una ripetuta a 16
o 17 basi, com maggiore attività degli
alleli con copie più lunghe
Farmacodinamica
6 tipi di recettori 5-HT (14 geni
differenti) sono stati identificati:
5-HT-1A, -1B, -1C, -1D, -2
Un polimorfismo nel recettore 5HT2A
(T102C), è associato ad intolleranza
alla paroxetina.
Soggetti con questa variante hanno
particolarmente severi effetti
collaterali
DISTROFIA DEI CINGOLI
(GOWER)
DISTROFIE MUSCOLARI DEI CINGOLI (LGMD)
Limb girdle dystrophies: Dominant
1A: Myotilin; 5q31; Dysarthria
1B: Lamin A/C; 1q21; + Cardiac
1C: Caveolin-3; 3p25; Child onset
1D: 7q
Dilated Cardiomyopathy (?1E): 6q23
1F: 7q32
Ankle contractures & High CK
Bethlem: Collagen VI; 21q22 & 2q37
Central core: Ryanodine receptor (19q13)
Cytoplasmic body: 2q24; 2q21 + Other
Distal myopathies
MPD2: 5q31; ? Same locus as LGMD1A
Emery-Dreifuss: Lamin A/C; 1q21
Facioscapulohumeral: 4q35
Myofibrillar (Desmin storage)
Desmin: 2q35; AD or AR
αB-crystallin: 11q22
Myotonic (DM1): DMPK; 19q13
Myotonic (DM2): ZNF9; 3q21
Oculopharyngeal: PABP2; 14q11
Paget disease + Myopathy: 9p13
Spheroid body
Limb girdle dystrophies: Recessive
2A: Calpain-3 ;15q15
2B: Dysferlin; 2p12
2C: γ-Sarcoglycan; 13q12
2D: α-Sarcoglycan; 17q21
2E: β-Sarcoglycan; 4q12
2F: δ-Sarcoglycan; 5q33
2G: Telethonin; 17q11-12
2H: TRIM32; 9q31-q33
2I: FKRP; 19q13.3
2J: Titin; 2q31
Merosin (Laminin α2)
Absent: 6q2
Reduced: 6q2
Abnormal: LGMD 2I
Caveolin-3 mutation (Gly55Ser)
Limb girdle dystrophies: X-linked
Barth: G4.5 (Tafazzins); Xp28
Becker: Dystrophin; Xp21
Duchenne: Dystrophin; Xp21
Emery-Dreifuss: Emerin; Xq28
McLeod Syndrome: XK; Xp21.1;
Vacuolar
Danon's disease: LAMP-2; Xq24
Excessive Autophagy: Xq28
Mental retardation & Cardiomyopathy