Riepilogo 1^ lezione
• I 20 amminoacidi che si trovano
comunemente nelle proteine sono uniti l’uno
all’altro da legami peptidici.
• La sequenza lineare degli amminoacidi legati
contiene l’informazione necessaria a generare
una proteina con una forma tridimensionale
esclusiva.
• La struttura di una proteina è complessa:
organizzazione in 4 livelli gerarchici
(struttura primaria, secondaria, terziaria,
quaternaria).
Gli amminoacidi possono unirsi tra loro con legami peptidici
Estremità amminica
Il ripetersi di questa reazione dà luogo a polipeptidi e
proteine.
Proprieta’ del legame peptidico:
Planare, ha una forza intermedia tra il legame semplice ed il
legame doppio.
R
O
H
C
N
C
H
O
R
C
C
N
H
+
OH
H
H
R
H
H
H
O
C
H
H
C
N
R
N
H
O
C
OH
C
OH
Il legame peptidico è rigido e planare
Gli atomi di Cα di amminoacidi adiacenti sono separati
da tre legami covalenti:
O H
Cα – C – N – Cα
PROPRIETA’ DEL LEGAME PEPTIDICO
I 6 atomi del gruppo peptidico giacciono sullo stesso piano →
l’ossigeno legato al carbonio del gruppo carbonilico e l’atomo di
idrogeno legato all’azoto amminico, si trovano in trans.
L’ossigeno carbonilico ha una parziale carica negativa e
l’azoto amminico ha una parziale carica positiva → ciò genera
un parziale dipolo elettrico.
I legami ammidici C-N hanno un parziale carattere di doppio
legame per effetto della risonanza→ non possono ruotare
liberamente.
La rotazione è permessa solo attorno ai legami N-Cα e Cα-C.
Il legame peptidico è rigido e planare
φ e ψ sono di 180° quando il polipeptide è nella conformazione complanare estesa e
tutti i gruppi peptidici sono sullo stesso piano.
φ e ψ possono assumere tutti i valori compresi tra -180° e +180°, ma molti valori
risultano proibiti per interferenze steriche tra gli atomi dello scheletro del polipeptide
e quelli delle catene laterali.
Caratteristiche del legame peptidico
• Ha il carattere di un doppio legame parziale (è più corto
di un legame singolo).
• E’ rigido e planare (non è possibile la rotazione attorno
al legame tra il carbonio carbonilico e l’azoto del legame
peptidico).
• In genere è un legame di tipo trans, a causa di
interferenze steriche tra i gruppi -R (i legami tra un Cα
e un gruppo α-amminico o α-carbossilico possono
ruotare!)
• I gruppi -C=O ed -NH del legame peptidico non hanno
una carica elettrica (a differenza del gruppo αamminico all’estremità N-terminale ed α-carbossilico al
C-terminale) ma sono polari e partecipano alla
formazione di legami a idrogeno.
• I singoli amminoacidi in una catena peptidica
sono chiamati residui amminoacidici.
• In genere le proteine sono composte da 502000 residui amminoacidi.
• La struttura primaria di una proteina è
definita dalla sequenza lineare dei
residui amminoacidici.
La peculiare sequenza amminoacidica di una catena
polipeptidica rappresenta la struttura primaria
Lisozima
Struttura secondaria
• Si riferisce alla conformazione locale della
catena polipeptidica.
• E’ determinata da interazioni di tipo legame a
idrogeno fra l’ossigeno di un gruppo
carbonilico del legame peptidico e l’idrogeno
del gruppo ammidico di un altro legame
peptidico.
• Esistono due tipi di strutture secondarie:
l’ α-elica ed il foglietto β.
proteine: struttura secondaria
strutture dovute ad interazioni “locali” di tipo ponte-H
α-elica
• ponte-H ogni 3,6
aminoacidi
•Il legame H si
instaura tra l’H
dell’azoto amidico
e l’O del gruppo
carbonilico
• residui esterni
alla spirale
β-foglietto
• legami idrogeno fra aminoacidi di
catene diverse
• foglietto piegato
Struttura secondaria (α
α-elica)
• E’ una struttura in cui la catena polipeptidica è
avvolta a spirale .
• Le catene laterali degli amminoacidi (-R) si
protendono verso l’esterno rispetto all’asse della
spirale.
• L’α-elica è stabilizzata da legami idrogeno
intracatena che si formano tra l’ossigeno
carbonilico di un legame peptidico e l’idrogeno
ammidico di un legame peptidico situato a 4 residui
di distanza sulla catena.
• La prolina interrompe l’α-elica!!!
• Gli amminoacidi con catene laterali (-R ) voluminose
o cariche possono interferire con la formazione
dell’α-elica.
Struttura secondaria: alfa elica
Legame idrogeno
Le proprietà idrofobiche o
idrofiliche di una alfa-elica
dipendono dalle catene
laterali degli aa
Champe et al., Le basi della biochimica, Ed. Zanichelli
Legame H
α-elica
• ponte-H ogni
3,6 aminoacidi
•Il legame H si
instaura tra l’H
dell’azoto
amidico e l’O del
gruppo
carbonilico
Esempio di proteina composta da alfa eliche
Struttura secondaria
(foglietto β)
• E’ una struttura ripiegata, formata da 2 o più
catene polipeptidiche (filamenti) quasi
completamente distese.
• I legami a idrogeno sono intercatena e
perpendicolari allo scheletro del peptide.
• Tutti i componenti di un legame peptidico
partecipano alla formazione di legami a idrogeno.
• Tali legami si realizzano tra l’ossigeno di un gruppo
carbonilico di un legame peptidico e l’idrogeno del
gruppo ammidico di un altro legame peptidico
appartenente ad un filamento diverso.
Struttura secondaria: foglietto beta
Nei foglietti pieghettati ci sono ancora dei
legami ad idrogeno,
ma stavolta sono tra fogli adiacenti (sheet)
Struttura secondaria
(foglietto β)
• I polipeptidi che formano un foglietto β
possono disporsi in modo parallelo o antiparallelo.
• Un foglietto β può essere formato anche da
una singola catena polipeptidica ripiegata su
se stessa: in tal caso i legami a H sono legami
intracatena.
• La superficie dei foglietti β è “pieghettata”.
β Sheet
Stabilizzata da legami H intercatena
tra N-H & C=O
2 Orientations
Parallel
Not optimum H-bonds;
less stable
Anti-parallel
Optimum H-bonds; more
stable
Struttura secondaria
(sequenze non ripetitive)
• Queste strutture non ripetitive non sono
“casuali”.
• Hanno una forma meno regolare rispetto all’
α-elica ed al foglietto β.
• La catena polipeptidica assume una
conformazione ad anse ed avvolgimenti.
•
•
•
•
•
•
•
•
•
Circa un terzo dei residui che costituiscono le proteine globulari sono coinvolti in
ripiegamenti "a gomito" che invertono la direzione della catena polipeptidica alla
superficie della molecola e rendono possibile la struttura globulare. Data la loro
frequenza, questi ripiegamenti vengono classificati come terzo tipo di struttura
secondaria oltre alle α-eliche e ai foglietti β.
α-elica
Foglietto-β
β-turns
Esistono diversi tipi di ripiegamento che coinvolgono diversi residui. I più frequenti
sono i β-turns (tradotti in italiano come curve β, ripiegamenti β, etc.), che spesso
uniscono due filamenti β antiparalleli a formare un'ansa a forcina. I β-turns sono
definiti da 4 residui che occupano le posizioni designate da i a i+3
i
i+1
i+2
i+3
Nell'ambito dei β-turns si possono identificare diversi tipi, ma i più frequenti sono
quelli cosiddetti di Tipo I e Tipo II, anche se il Tipo I è 2-3 volte più frequente del
Tipo II.
I ripiegamenti β sono frequenti nelle proteine. Le proteine globulari presentano circa 1/3 dei residui aa sotto
forma di ripiegamenti o anse, dove la catena proteica inverte la sua direzione, i ripiegamenti β sono i più diffusi,
collegano due estremità di un foglietto β antiparallelo, 4 residui con un angolazione di 180° e formazione di un
ponte di H tra il Carbonile del I a. e l’H dell’N del IV amminoacido. Gly e Pro sono frequenti in questa
conformazione.
Gly perché piccola e flessibile
Pro perché l’imminoacido promuove la forma cis degli aa.
coinvolti nel legame
Super-secondary Structure
β-turns in una proteina permettono che eliche e
foglietti si allineino
βαβ
αα
β meander
Qui sopra sono riprodotte le 2 conformazioni descritte per la stessa proteina, la proteina prionica, la cui
alterazione conformazionale provoca la cosiddetta malattia della Mucca Pazza(v.”Connessioni Biochimiche” ,
pag. 105 del Campbell-Farrell). A sinistra è illustrata la struttura a) della proteina prionica “buona”, che è
presente in moltissimi organismi, uomo compreso. A destra (b) è riportata la struttura della proteina prionica
“cattiva”. Come si può constatare nella forma a) esistono 2 tratti ad α-elica, 1 a sinistra (verde), ed 1 a destra
(rosso). Nel passaggio alla forma b) il tratto “verde” cambia ripiegamento e genera 2 tratti β antiparalleli. Pure
il tratto “rosso” fa altrettanto e genera i 2 tratti “rossi” β antiparalleli nella forma b),producendo , in definitiva,
un unico foglietto β formato da 4 tratti β contigui.
Le strutture primarie sono mantenute da legami peptidici.
Le strutture secondarie sono mantenute da legami idrogeno
tra atomi di residui aminoacidici.
Quattro livelli di struttura determinano la
forma di una proteina
alfa
Beta
• Primaria: la sequenza lineare degli amino acidi tenuti insieme
da legami peptidici.
• Secondaria: l’organizzazione di parti di una catena
polipeptidica (es.: l’α elica o il foglietto β), tenute da legami
a ponte di H.
• Terziaria: la struttura tridimensionale completa di una
catena polipeptidica, con molti tipi di legami e interazioni di
cui solo uno covalente il ponte dS.
• Quaternaria: l’associazione di due o più polipeptidi in una
struttura complessa multi-subunità
Per funzionare una proteina deve assumere una struttura
tridimensionale precisa
collagene
mioglobina
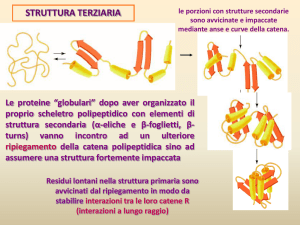
proteine: struttura terziaria
Determina la struttura 3D
• R apolari verso l’interno (eccetto in
Stabilizzata da
proteine integrali di membrana)
• ponti S-S
• R polari verso l’esterno (solvatati da H2O)
• interazioni idrofobiche
• interazioni elettrostatiche (legami ionici)
• legami di Wan der Waals
Suscettibile di denaturazione-rinaturazione
ponti disolfuro
Il Ponte Di-S si forma tra gruppi sulfidrilici adiacenti di
cysteine (-S-H).
La formazione avviene mediante ossidazione, il taglio in
residui disulfidici mediante riduzione.
Denatured inactive
ribonuclease
Struttura terziaria : relazione a lungo raggio tra amminoacidi anche molto distanti tra
di loro, I protagonisti sono i gruppi R che possono appartenere anche a filamenti con
diverse strutture secondarie.
proteine: struttura terziaria
effetto dell’interazione idrofobica
Legame a H
si può formare
sulla catena
laterale sul
lato esterno
della molecola
Non polare
Catena
laterale
Core idrofobico
contenente
catene laterali
non polari
struttura 3D
Polipeptide senza struttura
terziaria
Conformazione con struttura terziaria
in ambiente acquoso
ORIGINE
DELLA
STRUTTURA
TERZIARIA
L'effetto idrofobico è la forza motrice
del ripiegamento di una proteina.
La struttura terziaria si genera grazie
alle interazioni tra i gruppi R, i quali
si posizioneranno in risposta a
attrazione o repulsione, generando
la struttura finale.
Le strutture terziarie sono sempre
compatte
La superficie delle proteine è polare
mentre l'interno è prevalentemente
apolare eccezione per le proteine di
membrana, dove è l’opposto.
L'avvolgimento della catena deve
essere tale da esporre sempre al
solvente acquoso le catene laterali
idrofile. Catene laterali cariche
possono trovarsi all'interno di una
proteina solo se la loro carica netta
viene neutralizzata.
Legami responsabili della struttura
terziaria
Forze di Wan der Waals 1-2 Kcal/mole
Legami a H
3-7 Kcal/mole
Legami ionici
5 Kcal/mole
Legami S-S
50 Kcal/mole
Le proteine con peso
molecolare superiore a 50.000
sono OLIGOMERICHE
Sono costituite cioè da più
catene polipeptidiche
PROTOMERI O SUBUNITÀ
Le proteine oligomeriche presentano
un ulteriore livello di organizzazione
srutturale la struttura quaternaria
LA STRUTTURA QUATERNARIA
descrive il modo in cui le singole
catene polipeptidiche sono disposte
l'una rispetto all'altra.
Classificazione generale delle strutture
terziarie
Proteine con
predominanza
di α elica
Proteine
miste
Proteine con predominanza
di β sheets
Biofisica
Per la sintesi di una catena polipeptidica
di 4000 residui aminoacidici è necessario
un gene che contenga almeno 4OOO x 3
= 12.000 basi azotate.
Per la sintesi di 2O copie di una
stessa catena polipeptidica di
200 residui è sufficiente un gene
che contenga solo 200 x 3 = 600
basi
Emoglobina
Emoglobina e
collageno
Collageno
La struttura terziaria è generata dal ripiegamento e dalla
conformazione della catena polipeptidica.
La struttura quaternaria è l’organizzazione di polipeptidi
in un’unica unità funzionale che consiste di più di una
subunità polipeptidica.
Rappresentazioni grafiche differenti
della stessa proteina
In alcune catene polipeptidiche
particolarmente lunghe (più di 200
residui) si ritrovano 2 o più zone
distinte (30-150 residui) a struttura
globulare e compatta, congiunte da
segmenti di catena polipeptidica
relativamente flessibili.
Queste strutture si definiscono
DOMINI
LA STRUTTURA QUATERNARIA DI
UNA PROTEINA PUÒ SUBIRE
MODIFICHE CONFORMA ZIONALI
REVERSIBILI AD OPERA DI
LIGANDI, DEFINITI EFFETTORI
ALLOSTERICI.
LE MODIFICHE CONFORMAZIONALI
POSSONO ALTERARE LA FUNZIONE DI
UNA PROTEINA, REALIZZANDO IN TAL
MODO UN IMPORTANTE MECCANISMO
DI CONTROLLO DELLA SUA ATTIVITÀ
BIOLOGICA.
Riepilogo 2^lezione
Le proteine oligomeriche
presentano un ulteriore livello di
organizzazione srutturale la
struttura quaternaria
LA STRUTTURA QUATERNARIA
descrive il modo in cui le singole
catene polipeptidiche sono disposte
l'una rispetto all'altra.
La struttura quaternaria delle proteine
La struttura quaternaria riguarda proteine costituite da 2 o più
catene polipeptidiche o da più domini strutturali (es. proteine
regolatrici). E’ possibile classificare le proteine in due gruppi:
Proteine fibrose con catene disposte in lunghi fasci o foglietti e
Proteine globulari con catene polipeptidiche ripiegate a formare
forme globulari o sferiche
Esempio: la
emoglobina
Le interazioni tra le subunità consentono grandi variazioni
nell’attività catalitica
Biofisica
Per la sintesi di una catena polipeptidica
di 4000 residui aminoacidici è necessario
un gene che contenga almeno 4OOO x 3 =
12.000 basi azotate.
Per la sintesi di 2O copie di una stessa
catena polipeptidica di 200 residui è
sufficiente un gene che contenga solo
200 x 3 = 600 basi
Biofisica
In alcune catene polipeptidiche
particolarmente lunghe (più di
200 residui) si ritrovano 2 o più
zone distinte (30-150 residui) a
struttura globulare e compatta,
congiunte da segmenti di catena
polipeptidica relativamente
flessibili.
Queste strutture si definiscono
DOMINI
Biofisica
Struttura terziaria (i domini)
• Le catene polipeptidiche formate da più di 200 amminoacidi in
genere comprendono 2 o più piccole unità compatte: i domini.
• I domini sono le unità strutturali e funzionali di una proteina.
• Ciascun dominio è una regione globulare, compatta, che si
forma per la combinazione di più elementi strutturali secondari
(α-eliche, foglietti β, sequenze non ripetitive).
• Strutturalmente, ciascun dominio è indipendente da altri domini
della stessa catena polipeptidica.
• La struttura terziaria riguarda sia il ripiegamento di ciascun
dominio sia la disposizione reciproca finale dei domini di un
polipeptide.
Le proteine con peso
molecolare superiore a
50.000 sono
OLIGOMERICHE
Sono costituite cioè da più
catene polipeptidiche
PROTOMERI O SUBUNITÀ
Vantaggi della struttura quaternaria
Risparmio di DNA
Minimizzazione degli errori casuali
durante la biosintesi proteica
Presenza di interazioni allosteriche
La Ferritina ha un peso
molecolare di circa 480.000
daltons. Non è costituita da una
sola catena polipeptidica di 400
a.a. ma di 20 catene identiche
di circa 200 residui ciascuna.
LA GLICERALDEIDE-3-FOSFATO
DEIDROGENASI È COSTITUITA
DA 4 SUBUNITÀ IDENTICHE DI
330 RESIDUI (330 X 3 = 990
NUCLEOTIDI)
SE LA PROTEINA CONSISTESSE IN
1 CATENA DI (330 X 4) 1320
RESIDUI OCCORREREBBE UN
GENOMA DI 3 X 1320=3960
NUCLEOTIDI.
Generalmente nella formazione delle
proteine oligomeriche le subunità
difettose sono scartate.
Nelle cellule le proteine si sintetizzano
ad una velocità molto elevata.
Le cellule di E.Coli producono una
molecola proteica biologicamente attiva
contenente 100 residui aminoacidici in 5
sec a 37°.
COME FANNO LE PROTEINE AD
AVVOLGERSI NEL TEMPO DI POCHI
SECONDI?
Supponiamo che ciascuno dei 2 angoli
di torsione, φ ψ, di una proteina con n
residui possa assumere 3 conformazioni
stabili, le conformazioni possibili per
questa proteina saranno 32n circa 100n
Se la proteina può esplorare una
conformazione ogni 10-13 secondi
Il tempo in sec necessario per esplorare
tutte le conformazioni possibili sarà
t = 10 n / 1013
Per n= 100 t = 10 87 ( 20 miliardi di anni!)
Le proteine
non ricercano casualmente la
conformazione nativa fra le molte
possibili
si ripiegano seguendo vie dirette
PICCOLI TRATTI DI STRUTTURA
SECONDARIA SERVONO DA
MEDIATORI DEL PROCESSO DI
AVVOLGIMENTO.
LA FORMAZIONE DI CORTI SEGMENTI
DI STRUTTURA SECONDARIA È MOLTO
VELOCE.
Questi piccoli tratti (circa 15
residui) si stabilizzano formando
dei complessi
(es 2α
α, 2β,
β, αβ)
αβ che si chiamano
unità di avvolgimento.
Intorno a questi centri si
stabilizzano poi altri tratti di
struttura secondaria
L'avvolgimento spontaneo delle catene polipeptidiche nella loro
corretta struttura terziaria è un processo altamente cooperativo,
cooperativo in cui
la formazione di piccoli elementi accelera la produzione di altri più
grandi
Il processo di
ripiegamento
di una proteina
procede da
uno stato ad
alta energia ed
alta entropia
ad uno a bassa
energia e
bassa entropia
IL processo di avvolgimento delle
proteine può essere accelerato
dall’enzima “ proteina disolfuro
isomerasi”
Per alcune proteine a tale processo
partecipano gli chaperoni molecolari
che :
contribuiscono al corretto
avvolgimento di una proteina
nascente
consentono alle proteine ripiegate
in modo non corretto di raggiungere
la conformazione nativa
Esistono due classi di
chaperoni molecolari : la
famiglia Hsp70 e le
chaperonine (Hps60 o
GroEL ed o Hsp10 GroES )
Le chaperonine sono costituite da due tipi di
proteine HP60 (GroEL) e HP10 (GroES)
GroEL 14 subunità identiche (549 aa) disposte in due
anelli sovrapposti (7+7)
GroES 7 subunità identiche (97aa) formano un
anello eptamerico
Conformazione, modificazione e
degradazione delle proteine
• Una catena polipeptidica appena sintetizzata deve
conformarsi e spesso subire modificazioni chimiche per
generare la proteina finale
• Tutti i polipeptidi con la stessa sequenza amminoacidica
assumono, in condizioni standard, la stessa conformazione
(lo stato nativo), che è la più stabile conformazione che la
molecola può assumere.
L’ informazione per il “folding” della
proteina è contenuta nella sequenza
Proteine denaturare al calore, con acidi, o chimici perdono la
struttura terziaria e secondaria e la funzione biologica.
Il processo
è reversibile
Le chaperonine assistono le proteine nella fase di
folding, prevenendo il legame con ligandi inappropriati.
Molte malattie sono dovute al difettoso
ripiegamento di una proteina
Alcune patologie derivano da proteine che non sono in grado di
raggiungere la loro struttura funzionale e che tendono a formare
grossi aggregati (fibrille o forme amiloidi): Alzheimer, Parkinson,
encefalopatia spongiforme, diabete di tipo II.
In altri casi mutazioni puntiformi generano proteine che non
raggiungono la loro locazione finale o che non sono più in grado di
svolgere la loro funzione perché incapaci di legare i loro substrati.
La fibrosi cistica è un difetto nella proteina transmembrana che
agisce come un canale degli ioni cloro nelle cellule epiteliali (CFTR:
1480 amminoacidi). La mutazione più comune è la delezione di un
amminoacido (Phe 508) e la proteina mutata non si avvolge
correttamente.
Proteine conformate in modo
aberrante sono implicate nello
sviluppo di patologie
Una placca amiloide
nella malattia di
Alzheimer è un
agglomerato di
filamenti proteici
Comparazione sequenze
Hum αglobina:
Bovis:
Pig:
mvlspadktn vkaawgkvga hageygaeal
mvlsaadkgn vkaawgkvgg haaeygaeal
vlsaadkan vkaawgkvgg qagahgaeal
ermflsfptt ktyfphfdls hgsaqvkghg kkvadaltna vahvddmpna
ermflsfptt ktyfphfdls hgsaqvkghg akvaaaltka vehlddlpga
ermflgfptt ktyfphfnls hgsdqvkahg kvadaltka vghlddlpga
lsalsdlhah klrvdpvnfk llshcllvtl aahlpaeftp avhasldkfl
lselsdlhah klrvdpvnfk llshsllvtl ashlpsdftp avhasldkfl
lsalsdlhah klrvdpvnfk llshcllvtl aahhpddfnp svhasldkfl
asvstvltsk yr
anvstvltsk yr
anvstvltsk yr
L’omologia delle sequenze suggerisce
relazioni funzionali ed evolutive tra le
proteine