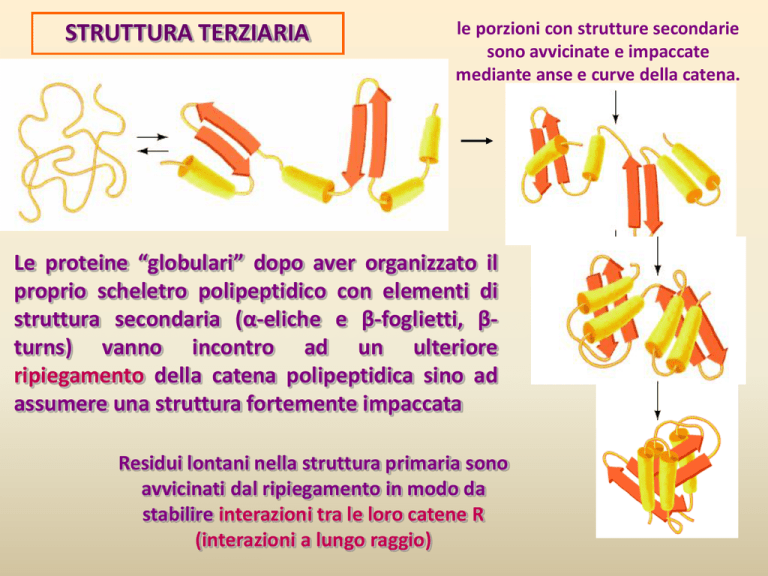
STRUTTURA TERZIARIA
le porzioni con strutture secondarie
sono avvicinate e impaccate
mediante anse e curve della catena.
Le proteine “globulari” dopo aver organizzato il
proprio scheletro polipeptidico con elementi di
struttura secondaria (α-eliche e β-foglietti, βturns) vanno incontro ad un ulteriore
ripiegamento della catena polipeptidica sino ad
assumere una struttura fortemente impaccata
Residui lontani nella struttura primaria sono
avvicinati dal ripiegamento in modo da
stabilire interazioni tra le loro catene R
(interazioni a lungo raggio)
Il ripiegamento (folding) di una proteina non è mai un processo casuale
Ma sempre dettato dalla sequenza amminoacidica della proteina.
Infatti, Il modo in cui una catena proteica si avvolge dipende dal tipo di
interazioni che si stabiliscono fra le catene laterali dei suoi residui amminoacidici.
Una proteina, fra tutti i possibili ripiegamenti che può assumere, addotta quello
in cui le interazioni fra le catene R dei suoi residui sono OTTIMALI e consentono di
raggiungere la maggiore stabilità (e quindi uno stato di minima energia)
La proteina addotta la sua conformazione nativa.
Il ripiegamento inizia durante la sintesi proteica ed è assistito da
“chaperoni molecolari”
Processo cooperativo: le prime interazioni che si formano tra le
catene laterali dei residui amminoacidici innescano le successive.
Processo rapido
E’ un processo che è innescato dall’effetto idrofobico
La catena polipeptidica si ripiega in modo da costringere le catene non
polari ad associarsi tra loro in un CORE core idrofobico da cui sono
escluse le molecole d’H2O.
H2O
Durante l’avvolgimento
le catene laterali polari
tendono ad essere
spinte sulla superficie
esterna della proteina, a
contatto con le molecole
d’H2O della sfera di
solvatazione.
Sotto la spinta dell’effetto idrofobico la proteina collassa su stessa e assume una struttura più
ordinata, quindi diminuisce l’entropia del polipeptide e aumenta quella del solvente (dal
core della proteina vengono espulse molecole d’H2O). II processo risulta energeticamente
favorito dalla formazione di numerosissime interazioni tra le catene laterali degli
amminoacidi che vengono avvicinati.
La struttura terziaria è stabilizzata dalle seguenti interazioni tra
le catene laterali R dei residui amminoacidici:
Ser
Non covalenti
Interazioni idrofobiche
Interazioni di van der
Waals
Legami idrogeno
Interazioni ioniche
Covalenti
Ponti disolfuro
Tyr
2 Cys
Leu
Lys
Phe
Asp
Durante il processo di avvolgimento della catena polipeptidica più
strutture secondarie riconoscibili si combinano a formare i MOTIVI O
STRUTTURE SUPERSECONDARIE
ansa β-α-β
Elica-ansa-elica
es: proteine
leganti calcio
Barilotto β
Motivo complesso caratteristico
dell’ α-emolisina di Staphylococcus
aureus (tossina che uccide le cellule
formando pori nella membrana)
DOMINI
Più motivi si possono associare a
formare dei DOMINI: Unità
distinte di una stessa proteina che
si ripiegano e si compattano
indipendentemente (da 30 a 300
residui amminoacidici).
Hanno una struttura riconoscibile e
sono in genere associati a
particolari funzioni.
Sono connessi da anse e legati
tra loro da interazioni deboli.
Non tutte le proteine globulari hanno
un’organizzazione a domini.
piruvato chinasi: 1 catena
polipeptidica organizzata in 3
domini
STRUTTURA QUATERNARIA
Presente in proteine costituite da 2 o più subunità, quindi da 2 o + catene
polipetidiche. Le subunità (o monomeri) possono essere identiche o diverse
I monomeri si associano nelle superfici di contatto per mezzo di
Interazioni idrofobiche, Forze elettrostatiche, legami idrogeno, ponti
disolfuro
Chaperonina
Emoglobina
DENATURAZIONE DI UNA PROTEINA
è la perdita della struttura tridimensionale
che si accompagna anche alla perdita della
funzione della proteina.
Può essere reversibile.
AGENTI DENATURANTI:
-CALORE (agisce sui legami idrogeno)
-pH ESTREMI (agiscono sulle interazioni
ioniche e legami idrogeno)
-SOLVENTI o SOLUTI ORGANICI (es. alcooli,
acetone, urea, cloruro di guanidinia) (agiscono su interazioni idrofobiche)
-DETERGENTI (es. SDS) - (agiscono su
interazioni idrofobiche)
-AGENTI RIDUCENTI (β-mercaptoetanolo,
ditiotreitolo) – (agiscono sui ponti
disolfuro)
Ribonucleasi A nativa,
cataliticamente attiva
Aggiunta di urea e
mercaptoetanolo
Ribonucleasi A
denaturata e
cataliticamente
inattiva
Rimozione di urea e
mercaptoetanolo
Ribonucleasi A
rinaturata con i ponti
disolfuro riformati
correttamente,
cataliticamente attiva
Modificazioni delle proteine
POST-TRASLAZIONALI = avvengono dopo la sintesi proteica
COTRASLAZIONALI = avvengono durante la sintesi proteica
Le proteine nella loro forma matura o per essere attivate nella loro
funzionalità spesso subiscono delle modificazioni che comprendono
una serie di trasformazioni chimiche della catena polipeptidica e
sono mediate generalmente (ma non sempre!) da enzimi specifici.
Sono numerose e interessano determinati residui amminoacidici
che si trovano all’interno di specifiche sequenze consenso o in
particolari motivi strutturali.
Tali modificazioni possono essere di natura covalente (ponti
disolfuro, Acetilazione, Ossidazioni, Fosforilazioni, Tagli proteolitici,
Glicosilazioni…..) o non covalente (coniugazioni con metalli o gruppi
prostetici attraverso interazioni polari o idrofobiche)
fosforilazione su serina
Acetilazione sull’N-terminale
3
H
C
-C |
H
N |
O
CO
O-
Anche i residui di Thr e Tyr
possono essere fosforilati
A
B
Taglio del peptide segnale: si ottiene la Proinsulina in cui le due porzioni N-terminale
(sequenza B) e C-terminale (sequenza A)
sono legate da 2 ponti disolfuro
Taglio proteolitico che rimuove la
sequenza di connessione interna: si
ottengono due peptidi A e B tenuti
insieme dai 2 ponti disolfuro
>>> INSULINA MATURA
GLICOPROTEINE
La glicosilazione è la più comune forma di modificazione
subita dalle proteine e può essere co-traduzionale (N-glicoproteine) o post-traduzionale (O-glico-proteine).
Le glicoproteine svolgono importanti
funzioni biologiche in svariati processi
cellulari: riconoscimento recettoriale e
immunologico, trasporto transmembrana
di proteine, infiammazione, patogenicità,
metastasi…
1) L’attacco degli zuccheri avviene attraverso un legame N-glicosidico
oppure O-glicosidico.
Proteine O-glicosilate → gli zuccheri
2) Proteine N-glicosilate → gli zuccheri
sono legati ad un residuo di asparagina sono generalmente legati ad un
residuo di serina (Ser) o treonina
(Asn). Legame β-N-glicosidico
(Thr). Legame β-O-glicosidico
N
HH
N
O
Asn - X - Ser/Thr
3) Nelle proteine N-glicosilate il
primo zucchero è sempre
N-Acetilglucosamina
(NAcGlc)
Ser/Thr
Nelle proteine O-glicosilate il primo
zucchero è frequentemente
l’N-Acetilgalattosamina (NAcGal), ma
potrebbe essere anche
N-Acetilglucosamina, fucosio,
mannosio, galattosio o xilosio.
Tutti gli N-glicani hanno lo stesso core pentasaccaridico costituito da
2 N-acetilglucosammine e 3 mannosi, su 2 di questi mannosi sono caricati due
rami di composizione variabile.
ramo 1
Fuc
Fuc
ramo 2
Gal
Gal
NAcGlc
NAcGlc
Man
Man
Man
NAcGlc
NAcGlc
--- Asn---
Core pentasaccaridico
A seconda del numero di siti di glicosilazione e del tipo di glicano presente su ogni sito si
possono ottenere tante glicoforme diverse della stessa
Glycoprotein structure determination by mass spectrometry. Science
291, 2351 (2001); Dell A. and Morris H.R.












