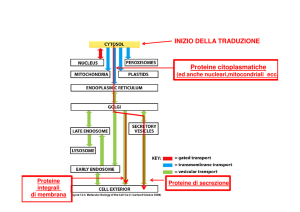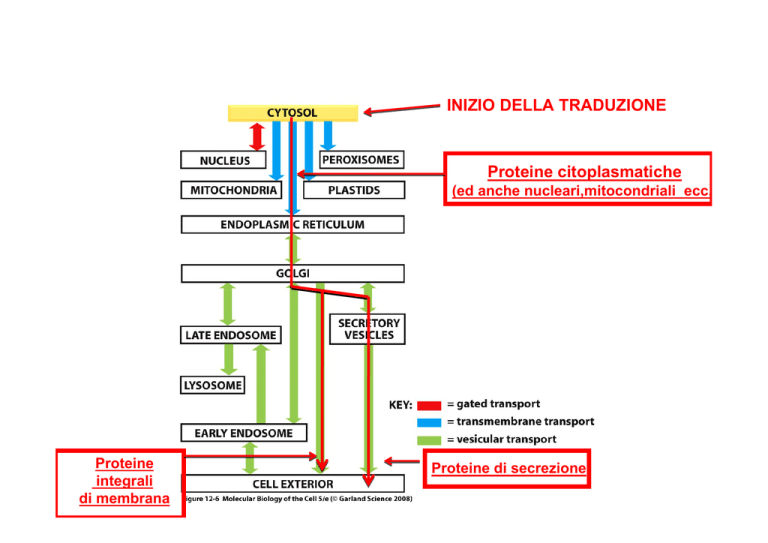
INIZIO DELLA TRADUZIONE
Proteine citoplasmatiche
(ed anche nucleari,mitocondriali ecc.
Proteine
integrali
di membrana
Proteine di secrezione
LA TRADUZIONE
INIZIA SEMPRE NEL CITOPLASMA
MEMBRANA
MEMBRANA
RER
proteine di secrezione
e di membrana
MITOCONDRI / CLOROPLASTI
PROTEINE DI SECREZIONE IN BATTERI
Proteine
citoplasmatiche
Proteine
di secrezione
o di membrana
PEPTIDE SEGNALE
eucariotico
AA
BASICI
AA
IDROFOBICI
Small Ribonuclear Particle
Un piccolo RNA (7SL) e 6 proteine
1
INIZIO DELLA
TRADUZIONE
2
4
SRP
RICONOSCE IL
PEPTIDE SEGNALE
(pausa nella traduzione)
3
RIPRESA
DELLA TRADUZIONE
ADESIONE DEL RIBOSOMA
ALLA MEMBRANA DI RER
6
LA PROTEINA
ENTRA NEL RER
VIA VIA CHE VIENE SINTETIZZATA
5
TAGLIO CON
ENDOPEPTIDASI
(Pept.segn.peptidasi)
Sequenza (o peptide) segnale
Sequenza di inizio della traslocazione al RER
•
Peptide segnale
•
che verrà
prontamente
digerito dalla
peptide-segnalepeptidasi
RIBOSOMA E SRP
“cerniera”
Tasca idrofobica
ricca di Met
7SL RNA
Sequenze Alu
Trasposoni SINE
Met
PROTEINA
DI
SECREZIONE
SECREZIONE
REGOLATA
COSTITUTIVA
LISOSOMI
PROTEINE
DI MEMBRANA
Citoplasma
Esterno
della cellula
MEMBRANA
CITOPLASM.
Tre modalità di inserimento
1
Sequenza segnale
iniziale
Alfa-elica di 20-22 AA
prevalentemente idrofobici
Sequenza di arresto
Rilascio dal poro
Taglio della
sequenza segnale
Aminoacidi
Idrofobici
dell’
alfa-elica
Alfa-eliche transmembrana
7 domini
un dominio
Tre modalità di inserimento
1
Sequenza segnale
iniziale
Alfa-elica di 20-22 AA
prevalentemente idrofobici
Sequenza di arresto
Rilascio dal poro
Taglio della
sequenza segnale
2
N
Sequenza segnale
INTERNA
Presenza di
AA carici
3
+
Inizio traslocazione
al RER
e successivo
intrappolamento
nella membrana.
Sequenza segnale: NON VIENE TAGLIATA
Seq. di arresto
Seq. Segnale INTERNA
Inizio traslocazione
Alfa-eliche transmembrana
7 domini
un dominio
Varie modalita’ di inserimento
• N-glicosilazione
Sequenza
Asn – X -- Ser/Thr
DOLICOLO
poli - isoprenoide
NEL RETICOLO ENDOPLASMATICO:
N – GLICOSILAZIONI
Formazione PONTI CISTINICI (disolfuro isomerasi)
Ripiegamento: proteina chaperone BiP
Gli enzimi “residenti” sono in buona parte SOLUBILI:
segnale di trattenimento nell’ ER: Lys-Asp-Glu-Leu-COOH
NEL GOLGI:
Processamento delle N-glicosilazioni operate nel RE
Aggiunta di O-Glicosilazioni
Enzimi spesso associati alla MEMBRANA
GLICOSILAZIONE
Reticolo endoplasmatico
Golgi
or Serine
LA TRADUZIONE
INIZIA SEMPRE NEL CITOPLASMA
MEMBRANA
MEMBRANA
RER
proteine di secrezione
e di membrana
MITOCONDRI / CLOROPLASTI
PROTEINE DI SECREZIONE IN BATTERI
PROTEINE DI SECREZIONE
IN BATTERI
Prima la
SINTESI
e poi la
SECREZIONE
Taglio del
peptide segnale
Trasporto ai mitocondri
Proteina già sintetizzata nel citoplasma,
con peptide segnale specifico all’ N-terminale.
Passaggio diretto nella matrice
Proteine della
membrana mitocondriale esterna
(meccanismi simili sono attivi nei batteri)
Sintesi seguita da ripiegamento e inserzione
Proteine della
membrana mitocondriale interna
o dello spazio intermembrana
Trasporto nel
cloroplasto
Mitocondri e cloroplasti
•
•
•
•
•
16500 bp
Contengono un genoma limitato ma funzionante,
e un completo apparato di sintesi proteica (somiglianza con i batteri).
Importano proteine codificate
da geni nucleari
Non esportano proteine
Genomi
(eccezione:’apoptosi)
ribosomiali
Si generano per fissione
Molto vari come dimensione e numero
Poche le proteine
comuni
a specie diverse
Genoma
mitocondriale
umano
Codifica per
13 proteine
22 tRNA
2 rRNA
Genoma in
“rapida” evoluzione
MITOCONDRI
•
Uso “rilassato” dell’accoppiamento codone/anticodone
(22 tRNA sono sufficienti).
•
Codice genetico leggermente diverso
Genoma di
cloroplasto
di pianta superiore
Forti similarità con
i cianobatteri
EREDITA’ NON MENDELIANA
(CITOPLASMATICA)
BIPARENTALE NEI LIEVITI
MATERNA IN ANIMALI SUPERIORI
Il fenotipo si
manifesta
casualmente
Colonie
piccole
Colonie
normali
o
am
l
iso
t
en
Colonie
piccole
Colonie NORMALI