La traduzione: dall’mRNA alle proteine
Le infezioni batteriche sono una grave causa di malattie e
morte in Europa e negli USA.
Le infezioni batteriche si curano con antibiotici che
colpiscono l’espressione genica .
Staphylococcus aureus è un batterio che può causare gravi
infezioni e inoltre può formare coperture mucillaginose
(biofilm) su superfici che rimangono costantemente umide. I
biofilm sono molto resistenti e difficili da eliminare.
Uno dei problemi più grandi nell’utilizzo dei cateteri è
proprio la possibilità di formazione di biofilm da cui poi si
distaccano batteri che creano stati infiammatori importanti
nei pazienti.
Le infezioni da S. aureus si curano con meticillina che si lega a
proteine batteriche necessarie per costruire nuove pareti
cellulari.
Il batterio ha però sviluppato resistenza alla meticillina (MRSA – Methycillin-resistant Staphylococcus
aureus) – molto comune in ospedali e case di riposo dove causa decine di migliaia di morti/anno
È stato sviluppato l’antibiotico tetraciclina che ha effetto sull’espressione genica del batterio, e non su
quella umana, perché lega una proteina specifica del ribosoma batterico, impedendo l’attacco del tRNA e
quindi la sintesi proteica.
… purtroppo negli ultimi anni MRSA ha sviluppato forme di resistenza anche alla tetraciclina!
Genotipo/fenotipo
• Il genotipo si esprime nel fenotipo e le proteine sono intermedi tra geni e
fenotipo.
• Ipotesi “un gene - un enzima”: la funzione di un gene sarebbe quella di
controllare la produzione di un singolo specifico enzima.
• Non tutte le proteine però sono enzimi quindi meglio “un gene- una
proteina”.
• Molte proteine sono costituite da più di una catena polipeptidica e ogni
catena è codificata dal proprio specifico gene. L’ipotesi più corretta è quindi
“un gene - una catena polipeptidica”.
• … ma ricordiamoci che molti geni trascrivono RNA che non diventano
polipeptidi, ma hanno funzioni di regolazione
Rapporto DNA/proteine
• Poiché le proteine sono i prodotti dei geni e i geni sono fatti di DNA,
l’informazione codificata dal DNA deve controllare il tipo e la quantità di
proteine presenti nella cellula.
• Come fanno i geni, costituiti da DNA, a controllare la produzione di
proteine?
• Le proteine sono molecole lineari formate dalle possibili combinazioni dei
20 aminoacidi.
• Se il DNA è formato dalla combinazione di 4 nucleotidi (A T G C) e ci sono
20 aminoacidi che formano le proteine, come viene tradotta l’informazione
genetica a partire da 4 nucleotidi?
Un codice a triplette soddisfa le esigenze di traduzione del codice genetico in
aminoacidi
- Se 1 nucleotide codifica per un singolo aminoacido, si potrebbero avere solo 4
aminoacidi
- Se fosse invece una sequenza di 2 nucleotidi a codificare per 1 aminoacido si
avrebbero 16 diversi aminoacidi
- Una sequenza di 3 nucleotidi permette 64 combinazioni
(44 combinazioni in più rispetto alle 20 necessarie per la sintesi degli aminoacidi esistenti)
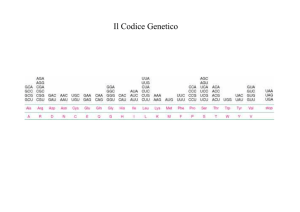
Codice a triplette
Il codice genetico
- Una sequenza di 3 nucleotidi (codone) codifica per 1 aminoacido
- Più codoni possono codificare per lo stesso aminoacido – il codice genetico è degenerato
- Alcuni codoni non codificano per nessun aminoacido
3 sono codoni di STOP (UAA, UAG, UGA)
1 è codone di inizio (AUG = metionina)
Il codice genetico è universale… ma con delle eccezioni
In tutti gli organismi gli stessi codoni codificano per gli stessi aminoacidi
(Supporta la teoria dell’evoluzione della vita sulla Terra da un progenitore comune)
Esistono tuttavia delle eccezioni:
-alcune specie presentano alcuni codoni che specificano per aminoacidi diversi
- Il codice genetico mitocondriale è parzialmente diverso da quello nucleare
Il codice genetico è degenerato… ma non è ambiguo
Ad eccezione dei codoni di inizio (1= AUG) e di stop (3= UAA, UAG, UGA),
rimangono 60 codoni disponibili, molti di più di quelli necessari per codificare i 19
aminoacidi rimanenti. A quasi tutti gli aa, corrispondono infatti più codoni
Il codice genetico è lineare e non sovrapposto
NO!
SI!
Il codice genetico è «senza virgole»
Se si toglie un nucleotide, la sequenza cambia da quel punto in avanti
Se si sostituisce un nucleotide, il messaggio, e quindi l’aminoacido,
può cambiare
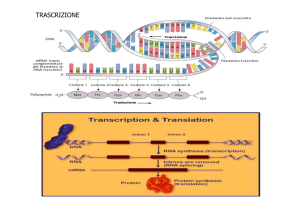
La sintesi proteica
Gli mRNA procariotici sono tradotti appena si staccano dal filamento stampo del DNA e la loro vita è in
genere di pochi minuti
Negli eucarioti l’mRNA viene modificato (maturazione), esce dal nucleo e viene tradotto nel citoplasma; la
vita media dell’mRNA è da molti minuti a ore
In entrambi i casi la traduzione è molto rapida (la sintesi di un polipeptide di centinaia di aminoacidi avviene in pochi minuti).
Cosa serve per fare la sintesi proteica
- Un mRNA (maturo)
- Gli aminoacidi
- I tRNA (adattatori che traducono i codoni dell’mRNA nella sequenza polipeptidica)
- I ribosomi
- Energia (sotto forma di ATP)
- Fattori proteici per l’inizio, l’allungamento e il rilascio della catena polipeptidica
mRNA
Struttura di un aminoacido
Gli aminoacidi
La struttura delle proteine
I 20 aminoacidi
Gli RNA transfer (tRNA)
DNA contiene geni che, trascritti, danno origine a RNA transfer
tRNA sono specifici per ciascun aminoacido
Struttura di 70-80 nucleotidi caratteristica per:
- 1 anticodone (sequenza di basi complementare al codone sull’mRNA)
- essere riconosciuta da specifici aminoacil-tRNA sintetasi
- 1 regione di attacco per l’aminoacido specificato dal codone complementare all’anticodone
- Essere riconosciuta dai ribosomi
La peculiare forma dei tRNA è fondamentale
per un preciso posizionamento degli aa
durante la traduzione
Uno specifico aminoacido si lega a un tRNA grazie all’enzima aminoacil-tRNA sintetasi
e consumo di ATP
Gruppo amminico libero per la
formazione di legami peptidici
Codone per Fenilalanina è UUC
Il caricamento della molecola di tRNA avviene per opera dell’aminoacil-tRNA sintetasi
che attiva uno specifico aminoacido e lo lega a uno specifico tRNA, utilizzando ATP
I ribosomi
Composti da proteine e RNA ribosomale (rRNA)
A differenza di mRNA e tRNA, l’rRNA non trasferisce informazioni, ma ha funzioni catalitiche
Costituiti da subunità minore e subunità maggiore.
Nel solco tra le due subunità «passa» l’mRNA
Hanno 3 siti di legame per i tRNA (A, P, E)
Sito P (o peptidilico), occupato dal tRNA con
attaccata catena polipeptidica in crescita
Sito E (o exit, di uscita), dove i tRNA che
hanno ceduto gli aminoacidi escono dal
ribosoma
Sito A (o aminoacilico), dove si lega
l’aminoacil-tRNA che porta l’aminoacido
successivo da inserire sulla catena
La traduzione è il processo mediante il quale l’informazione
dell’mRNA (derivata dal DNA) è usata per specificare una sequenza
di aminoacidi (polipeptide)
Come la trascrizione, avviene in tre fasi
- Inizio
- Allungamento
- Terminazione
L’inizio della traduzione
Formazione di un complesso di inizio:
tRNA carico, subunità minore del
ribosoma, mRNA
- Negli eucarioti subunità minore si
lega al 5’-cap dell’mRNA e scorre fino
al codone d’inizio (AUG)
-tRNA con anticodone complementare
(UAC) e carico di metionina lega il
codone e completa il complesso di
inizio
-Subunità maggiore si lega e il tRNAmet si trova così nel sito P. Sito A,
allineato con il secondo codone
Componenti assemblati da proteine
specifiche – fattori di inizio
L’allungamento
Un nuovo tRNA con anticodone complementare al
secondo codone si lega a sito A
Attività peptidil-transferasica della subunità maggiore
rompe legame tRNA-aa sul sito P e catalizza
formazione legame peptidico tra questo aa e quello
legato a sito A
(metionina diventa estremita N-term della nuova
proteina)
Quando ribosoma si muove di un codone lungo
mRNA in direz 5’-3’
- tRNA che ha rilasciato met si sposta verso sito E,
dove viene rilasciato nel citoplasma
-Il tRNA che ora porta un dipeptide slitta nel sito P
Il processo si ripete e la catena polipeptidica si
allunga
Fattori proteici di allungamento assistono queste fasi
La terminazione
La trascrizione termina quando un codone di
stop (UAA, UAG, UGA) entra nel sito A
Questi codoni non corrispondono a nessun aa
– non sono riconosciuti da alcun tRNA
Riconosciuti da un fattore di rilascio proteico
che idrolizza legame tra tRNA nel sito P e
ultimo aa del polipeptide
Il complesso tRNA, mRNA, subunità
ribosomali si separa e prodotti tornano
disponibili per un nuovo ciclo di traduzione
Riassunto segnali di inizio e terminazione per la
trascrizione e la traduzione
La velocità della sintesi proteica può essere
aumentata con la formazione di polisomi
Le cellule che sintetizzano grandi quantità
di proteine contengono moltissimi
polisomi e pochi ribosomi liberi
Il destino delle proteine e le
modifiche post-traduzionali
Quando un polipeptide
emerge dal ribosoma (nel
citosol) può acquisire la sua
forma definitiva e svolgere
subito il suo ruolo
Oppure polipeptide può
contenere una sequenza
segnale che lo dirige verso i
diversi comparti cellulari
Sequenza segnale lega uno
specifico recettore proteico su
superficie dell’organulo,
creando un poro che consente
l’ingresso della proteina
La maggior parte delle proteine mature non sono uguali al polipeptide tradotto –
modificazioni post-traduzionali
Proteolisi, catalizzata da proteasi. Es. rimozione della sequenza segnale del polipeptide nel RE (la
proteina potrebbe fuoriuscire dal RE); proteina virale HIV appena tradotta è troppo grande per poter
assumere conformazione attiva, viene quindi tagliata – alcuni farmaci anti-HIV agiscono inibendo
questa proteasi
Glicosilazione – aggiunta di carboidrati a formare le glicoproteine. Importanti per
direzionare proteine ai lisosomi o altri organuli e per il riconoscimento a livello di membrana
cellulare
Fosforilazione – aggiunta di gruppi fosfato a opera di protein-chinasi. Cambiamento di
conformazione della proteina che espone il sito attivo di un enzima o il sito di legame per un’altra
proteina