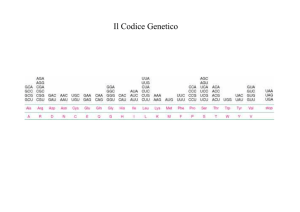
Il Codice Genetico
La decodifica della sequenza nucleotidica in
sequenza aminoacidica
•
La sequenza del mRNA viene letta a gruppi di 3 nucleotidi, senza interruzioni
e senza sovrapposizioni;
•
43 = 64 ---------64 triplette
•
61 triplette o codoni codificheranno per i 20 aminoacidi;
•
3 triplette UAA, UAG, UGA rappresentano i segnali d’arresto;
•
La lettura del messaggio inizia sempre con il codone AUG (Met);
•
Codoni multipli possono codificare lo stesso aminoacido (Ridondanza)
•
Un codone codificherà sempre per lo stesso aminoacido (non ambiguo)
•
In tutti gli organismi viventi (tranne alcune eccezioni) i codoni hanno lo
stesso significato (universale)
Il Codice Genetico
Ogni sequenza si presta ad essere tradotta in tre griglie di lettura
differenti, a seconda del punto in cui inizio il processo di
decodificazione sulla molecola.
In tutti i casi sarà soltanto una delle 3 griglie a dar vita ad una
proteina funzionale.
tRNA
Assumono una conformazione secondo
il modello a trifoglio
Methionine
Le differenze nelle sequenze nucleotidiche
determinano la capacità di legare un
amminoacido specifico
L’ansa II contiene la sequenza di 3 nucleotidi
detta ANTICODONE che si appaia, 1716 Pu A
9
17:1
13 12 Py 10
durante la traduzione al codone
G
22 23 Pu 25
G
dell’mRNA
A
26
2020:120:2
1
2
3
4
5
6
U* 7
A
C
C
73
72
71
70
69
68
67
Py 59A*
66
65 6463 62 C
49 50 51 52 G T C
y
Py
*
Questo appaiamento è fondamentale
per l’inserimento dell’amminoacido
corretto,come specificato dalla m-RNA,
nella catena polipeptidica in crescita.
Anticodon
27
1
28
29
30
31
Py*
Pu
47:16
47:15
4344
42 45
41 46
47
40
47:1
39
38
Pu*
U
34
U 35
C
A 36
20 aminoacidi
48 diversi anticodoni
molecole di tRNA)
61 codoni
(nei batteri 31 diverse
Wobble (Vacillamento)
Appaiamento sbagliato nella
terza posizione del codone
Codon-anticodon interactions
• codon-anticodon base-pairing is antiparallel
• the third position in the codon is frequently degenerate
• one tRNA can interact with more than one codon
• wobble rules
5’ tRNAmet
3’
UAC
AUG
5’
mRNA
3’
3’
• one tRNAleu can read two
of the leucine codons
5’
mRNA
5’
GAU
CUA
G
tRNAleu
wobble base
3’
Attivazione dell’aminoacido
(fase ATP-dipendente)
Legame dell’aminoacido al tRNA
(estremità COOH dell’aminoacido e il 3’OH del tRNA)
L’enzima che catalizza questa reazione è
l’aminoacil-tRNAsintetasi
1) Reazione altamente specifica
2) Fornisce l’energia che verrà utilizzata per la formazione del
legame peptidico durante la sintesi proteica
Reazione in 2 passaggi con utilizzo di ATP
1. ATP + Aminoacido = Aminoacido adenilato + 2P
2. Aminoacido adenilato + tRNA = AminoaciltRNA + AMP
Fase GTP-dipendente
Fase d’inizio
Il tRNA iniziatore portante la metionina si lega
alla subunità minore del ribosoma assieme a
dei fattori d’inizio.
Si lega l’mRNA, la subunità minore del
ribosoma scorre sull’mRNA fino a quando non
incontra il codone d’inizio AUG.
Si instaura il legame tra l’AUG e l’anticodone
del tRNA iniziatore.
Si associa la subunità maggiore.
Il tRNA iniziatore occupa il sito P, al sito A è
presente il secondo codone dell’ mRNA
Fase d’allungamento
Al sito A si lega il tRNA acilato (portante il
secondo
aminoacido
della
catena
polipeptidica).
Si forma il legame peptidico tra l’estremità
COOH terminale della Metionina e l’estremità
NH2 terminale del secondo aminoacido ad
opera dell’attività enzimatica della subunità
maggiore del ribosoma.
L’energia necessaria è data dall’attivazione
aminoacidica.
Formazione del legame peptidico
Sito P
Sito A
Polimerizzazione di testa
Il legame ad alta energia è fornito dal monomero già
inserito (proteine)
Polimerizzazione di coda
Il legame ad alta energia è fornito dal monomero
entrante (DNA – RNA)
TRASLOCAZIONE
Il tRNA scarico che occupava il sito P occuperà il
sito E
il tRNA portante il dipeptide che occupava il sito A
occuperà il sito P
al sito A sarà presente il terzo codone dell’mRNA
pronto ad ospitare un nuovo tRNA acilato
Terminazione
Quando al sito A sarà presente uno
dei 3 segnali di arresto, si legheranno
dei fattori di terminazione e la sintesi
sarà bloccata
Traduzione - Inizio
fMet
Large
subunit
E
P
A
UAC
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
Small mRNA
subunit
3’
Traduzione - Allungamento
Polypeptide
Arg
Met
Phe
Leu
Ser
Aminoacyl tRNA
Gly
Ribosome
E
P
A
CCA
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
mRNA
3’
Traduzione - Allungamento
Polypeptide
Met
Phe
Leu
Ser
Gly
Arg
Aminoacyl tRNA
Ribosome
E
P
A
CCA UCU
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
mRNA
3’
Traduzione - Allungamento
Polypeptide
Met
Phe
Leu
Ser
Gly
Arg
Ribosome
E
P
A
CCA UCU
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
mRNA
3’
Traduzione - Allungamento
Polypeptide
Met
Phe
Leu
Ala
Ser
Gly
Aminoacyl tRNA
Arg
Ribosome
E
P
A
CCA
UCU
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
mRNA
3’
Traduzione - Allungamento
Polypeptide
Met
Phe
Leu
Ser
Gly
Arg
Ribosome
E
Ala
P
A
UCU CGA
5’GAG...CU-AUG--UUC--CUU--AGU--GGU--AGA--GCU--GUA--UGA-AT GCA...TAAAAAA
mRNA
3’
Ciascuna proteina si ripiega normalmente in
un’ unica conformazione stabile che in genere
è quella in cui è minima l’energia libera.
Tuttavia la conformazione può cambiare
leggermente quando la proteina interagisce
con altre molecole della cellula.
Questo cambiamento di forma è spesso
cruciale per l’attività della proteina
Modificazioni post-traduzionali
• Glicosilazione
• Acetilazione
• Fosforilazione
10_01.jpg
Regolazione dell’attività di una proteina
Livello trascrizionale
Modificazioni chimiche di una proteina
Regolazione positiva
Regolazione negativa
01_23.jpg
Il Genoma cellulare specifica inoltre:
• La struttura primaria delle proteine
• Presenza o assenza della proteina in un
determinato tipo cellulare o in un
determinato momento della vita cellulare
• La struttura primaria di RNA non
tradotti
• Destinazione delle proteine all’interno
della cellula
La variazione di un singolo aminoacido può provocare
gravi effetti sul fenotipo dell’organismo