CORSO INTEGRATO di
Genetica e Biologia Molecolare
GENETICA
a.a. 2015-2016
03/11/2015
Lezioni 23 e 24
Analisi di linkage
Elisabetta Trabetti
LINKAGE
Geni in loci vicini sullo stesso cromosoma – concatenati
(loci sintenici)
Tendenza di loci (geni e/o marcatori)
vicini su un singolo cromosoma ad essere
trasmessi assieme, come una unità,
attraverso la meiosi
[Genetica - B.A. Pierce. Zanichelli 2005]
Gameti parentali e ricombinanti
Analisi di Linkage
Analisi di associazione o di concatenazione
Co-segregazione di alleli a 2 o più siti polimorfici
all’interno di una famiglia
Probabilità dei marcatori di essere associati
rispetto alla probabilità di essere indipendenti
2 loci polimorfici in chr diversi: 50% probabilità un
allele al primo locus segreghi con un allele al secondo
locus
A1A2 B1B2
A1B1 50%
2 loci sintenici: probabilità di co-segregazione correla
con la distanza tra i loci
Probabilità che avvenga un evento di ricombinazione tra
due loci
FREQUENZA o FRAZIONE DI
RICOMBINAZIONE: θ
Frazione di ricombinazione, θ
Probabilità che avvenga un evento di ricombinazione
tra due loci
θ=
q= 0,5
q=0
n° meiosi ricombinanti
n° tot meiosi
n° soggetti ricombinanti
n°tot figli
loci molto lontani o su cromosomi diversi Assort Indipend
loci molto vicini Associati
q = misura della distanza genetica
U di misura: centimorgan cM
1 cM= lunghezza genetica in cui 1% di ricombinazione (q =0,01)
NB: distanza genetica non è strettamente proporzionale alla distanza
fisica (lunghezza in nucleotidi)
MISURA DELLA DISTANZA GENETICA
q = n. figli ricombinanti/n. tot figli
= 1/8 = 0,125 = 12,5%
Distanza A-B = 12,5 cM
“Vero linkage o dovuto al caso?”
Poniamo q = 0.5 il valore che ci attendiamo per 2 loci
indipendenti
Lo spostamento del valore da 0.5 è significativo da un
punto di vista statistico?
Analisi di linkage nell'uomo utilizzano i metodi basati
sul calcolo della verosimiglianza o likelihood
Analisi di linkage nell'uomo utilizzano i metodi basati
sul calcolo della verosimiglianza o likelihood
Probabilità di un valore q in relazione al numero
di ricombinazioni osservate tra i loci
Probabilità che i loci siano associati a un dato q
LOD =Z(q) =
Probabilità che i loci non siano in linkage (q=0,5)
(q =0-0.5) Valore q con LOD score > è la frequenza di ricombinazione più
probabile tra i dati osservati nelle famiglie
Due loci sono in linkage per un q se LOD score ≥+3 (1000:1)
No linkage se LOD score ≤-2 (1:100)
COSTRUZIONE DI MAPPE GENETICHE
Frazione di ricombinazione tra i loci:
A e B = 0,06 (6cM)
B e C = 0,07
A e C = 0,10
Frazione di ricombinazione tra i loci:
A e D = 0,04
D e B = 0,08
0.08 < somma 0.04 + 0.06
Ordine 3 loci sul chr
RICOMBINAZIONE MASSIMA: 50% !
Ricombinazione semplice
Ricombinazione doppia
0.06
A
0.07
B
0.10
C
MAPPA GENETICA DEL CROMOSOMA 22
Distanza genetica e distanza fisica
Non c’è correlazione costante
< ricombinazioni maschio
mappa genetica più corta
Approssimazione: 1 cM = 1 Mb
ANALISI di LINKAGE:
applicazioni
● Mappaggio di geni malattia e di nuovi
marcatori
● Diagnosi indiretta di malattia - Deve
essere
nota
la
localizzazione
cromosomica del gene
SIMBOLI USATI PER LA COSTRUZIONE
DEI PEDIGREE
Il probando è l’individuo attraverso cui si è arrivati allo
studio di quel pedigree, viene indicato con una freccia
Ogni individuo è identificato in modo non ambiguo da una
coppia di numeri (un numero romano per la generazione
ed uno arabo per i soggetti all’interno della stessa
generazione – ordine di nascita)
Meiosi informative e non
AD
Locus marcatore A
Alleli 1 e 2
Meiosi informative = individuare i gameti
ricombinanti e non
Analisi di
segregazione degli
alleli locus
marcatore rispetto
al fenotipo
Loci associati sono
trasmessi assieme
Determinazione
della fase
Associazione degli alleli
al locus marcatore con
il locus malattia
AD
LINKAGE - ricombinazione
2 loci
Locus marcatore (1 o 2)
Locus malattia (- o +)
Segregazione di alleli al locus
marcatore e al locus malattia
P
M
1
2
F1
1
2
F2
2
1
2
F3
2
2
2
LINKAGE - ricombinazione
2 loci
Locus marcatore (1 o 2)
Locus malattia (- o +)
P
M
1
2
F1
1
2
F2
2
1
1
2
1
2
2
F3
2
2
2
Determinazione della fase
Associazione degli alleli al locus marcatore con il locus malattia
Ipotesi: allele 2 associato malattia
50% ricombinazione
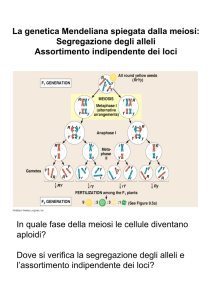
Assortimento indipendente:
gene malattia NON è in linkage con il marcatore
REQUISITI
Famiglie (numerose)
Affetti
Marcatori genetici
altamente polimorfi
uniformemente distribuiti
facile individuazione e basso costo
(A) Distinguere i due cromosomi omologhi dei genitori – marcatore linked
(B) Determinare la fase = cromosoma con allele patologico
(C) Quale cromosoma e’ stato trasmesso al probando
ANALISI di LINKAGE:
applicazioni
● Mappaggio di geni malattia e di nuovi
marcatori
● Diagnosi indiretta di malattia - Deve
essere
nota
la
localizzazione
cromosomica del gene
Informatività diagnostica di un marcatore
nella analisi di linkage - AR
1/1
-/+
2/2
-/+
Famiglia non informativa
1/2
-/-
1/2
-/+
1/1
-/1/2
-/+
1/2
-/-
1/2
-/+
Famiglia pienamente informativa
1/2
-/+
Famiglia parzialmente informativa
Diagnosi prenatale CF mediante analisi
di Linkage
MetH: extragenico, ~1Mb
1-2
-/+
1-1
-/+
1-2
?/?
1-1
-/-
MWM
P
M
CF
allele 2 p
allele 1 m:?
V
C+
1-2 1-1 1-1 1-2 2-2
1-2
?/+
?
• portatore (CF carrier)
o
• sano
Diagnosi prenatale CF
Xv-2c: extragenico, ~200 kb
1-2
Figlio affetto:
1-2
1-1
1-2
1*p e 2*m
allele 1 p
allele 1 m
?
oppure
1*m e 2*p
Feto:
MWM
P
M
CF
V
C+
1*p e 1 m
oppure
1*m e 1p
1-2 1-2 1-2 1-1 2-2
in entrambi i casi è
PORTATORE
Diagnosi prenatale CF
1*-2
1*-1*
1-2*
1*-2*
1-1*
1*-2
• sano o portatore
1*-2
1-1*
MetH: sano o carrier
Xv-2c: carrier
• portatore
Feto: carrier m causa CF
Fattori che determinano
l’accuratezza diagnostica nella analisi
di linkage
Vicinanza del marcatore al gene – frequenza di
ricombinazione
FC: KM19 <<0.01, MetH e J3.11 =0.01
DMD marcatori intragenici: 0.05
Errori da non paternità biologica
Errori di laboratorio
Nuove mutazioni
Eterogeneità genetica
Analisi di linkage e studio degli aplotipi
AD
Locus malattia DISTALE rispetto al primo marcatore
Il gamete con la combinazione 1-4-1 trasmesso assieme all’allele malattia
da I-1 a II-1 = aplotipo associato alla malattia controllo in generaz III
Aplotipo = serie di alleli in loci associati su un cromosoma (pat o mat)
LINKAGE disequilibrium
Specifica combinazione di alleli in fase a due o piu’
loci linked che si verifica più spesso di quanto
accadrebbe per puro caso
Associazione a livello di popolazione tra un particolare
allele marcatore e una malattia
In una popolazione molte persone non imparentate
hanno ereditato un cromosoma con una patologia da
un antenato comune
LINKAGE
DISEQUILIBRIUM
LINKAGE
M
1
M
3
M
1
Locus “A”
alleli 1 e 2
M
3
M
1
M
1
M
2
M
3
M
3
M
3
M
2
M
2
M
2
M
3
M
3
M
3
Locus A
Locus B
25%
40%
50%
25%
10%
0%
25%
10%
0%
25%
40%
50%
Linkage
equilibrium
Linkage
disequilibrium
parziale
Linkage
disequilibrium
completo
Origine del LD
Mutazioni
ricombinazioni
La determinazione del LD richiede lo studio di nuclei familiari
stabilire gli aplotipi
Possibilità di determinare elevato LD in soggetti non
familiari
(popolazioni isolate – pochi antenati pochi aplotipi originali)
AD: locus a 0.1 cM locus marcatore A
Malati (Mm)
Controlli (mm)
Individui con
allele A1
67
32
Totale individui
analizzati
84
275
Allele A1 >>> frequente negli affetti
Mut responsabile AD è avvenuta sul chr A1
LD responsabile delle differenze osservate
Grande numero di SNP vicini ha evidenziato che il
genoma umano è costituito da una serie di blocchi
di LD
Frequenze aplotipi nei blocchi diverse rispetto
all’equilibrio
Utili per la determinazione dei geni responsabili di
malattie complesse
Progetto HapMap
studio collaborativo internazionale - 2002
obiettivo: individuazione degli aplotipi comuni del genoma
umano per utilizzo in studi di linkage e GWAS (mappaggio geni
malattia)
- popolazioni con antenati Europei, Asiatici e Africani
- ciascun aplotipo contrassegnato da “tagging SNP”
- 500.000 – 600.000 tag SNP si può definire con buona
approssimazione i genotipi di 107 SNP comuni
CEU: Utah residents with Northern and Western European ancestry from the CEPH
collection, CHB: Han Chinese in Beijing, China, JPT: Japanese in Tokyo, Japan, YRI: Yoruba in
Ibadan, Nigeria
ASW: African ancestry in Southwest USA
CHD: Chinese in Metropolitan Denver, Colorado
GIH: Gujarati Indians in Houston, Texas
LWK: Luhya in Webuye, Kenya
MEX: Mexican ancestry in Los Angeles, California
MKK: Maasai in Kinyawa, Kenya
TSI: Toscans in Italy
HapMap: Ciscun aplotipo può essere identificato in
maniera univoca tramite una delle SNP che lo compone
(tag-SNP)
Costruzione aplotipi
(a) Single nucleotide polymorphisms(SNPs) are identified in DNA samples from multiple
individuals.
(b) Adjacent SNPs that are inherited together are compiled into "haplotypes."
(c) "Tag" SNPs within haplotypes are identified that uniquely identify those haplotypes.
By genotyping the three tag SNPs shown in this figure, researchers can identify
which of the four haplotypes shown here are present in each individual.
Identificazione di aplotipi
Utili in studi di associazione
Confronto aplotipi di “casi” con aplotipi di “controlli”
Aplotipo più frequente in casi
Gene causale interno o vicino aplotipo
Linkage disequilibrium e FC
Esercizi
(1) Dato il genotipo AaBb: quali sono i geni e quali gli alleli?
Disegnate schematicamente questo genotipo con i geni
concatenati in repulsione, posizionando gli alleli sui cromosomi
omologhi.
(2) Disegnate schematicamente un genotipo quadruplo
eterozigote con i geni A e C concatenati e i geni B e D
indipendenti, posizionando gli alleli sui cromosomi omologhi.
(3) Disegnare schematicamente due loci indipendenti. Potrebbero
essere posizionati sullo stesso cromosoma?
(4) Quali gameti produce prevalentemente un triplo eterozigote
AaBbCc considerando i 3 casi:
gli alleli A e B concatenati in repulsione e anche B e C
gli alleli A e B concatenati in cis e C in repulsione
gli alleli A e B concatenati in cis e il locus C non
concatenato
(5) Dato il seguente incrocio AaBbDD x AABbdd in cui tutti i
geni segregano indipendentemente
a) Quanti gameti geneticamente differenti sarà
possibile ottenere da ciascun genitore?
b) Quale sarà la proporzione di individui eterozigoti
a tutti i loci nella prole?
(6)
Quali fenotipi produce il seguente incrocio e con quali
frequenze
AaBbCc X AABBCc
(A, B, C, alleli dominanti)
Cambiano le frequenze se il primo individuo è omozigote per
l’allele recessivo al primo locus?
(8) Dal genotipo AaBb vengono prodotti i seguenti
gameti:
AB = 28 gameti
Ab = 472 gameti
aB = 469 gameti
ab = 31 gameti
I loci A e B sono associati o indipendenti?
E’ possibile determinare la distanza genetica tra i
due loci?