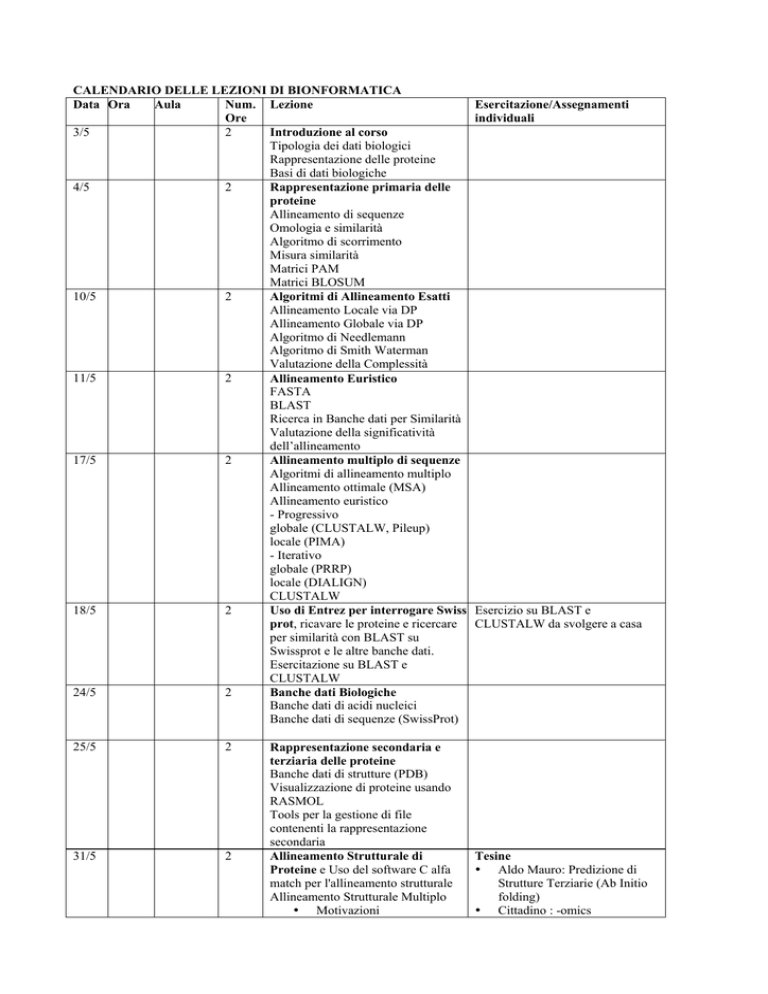
CALENDARIO DELLE LEZIONI DI BIONFORMATICA
Data Ora
Aula
Num. Lezione
Ore
3/5
2
Introduzione al corso
Tipologia dei dati biologici
Rappresentazione delle proteine
Basi di dati biologiche
4/5
2
Rappresentazione primaria delle
proteine
Allineamento di sequenze
Omologia e similarità
Algoritmo di scorrimento
Misura similarità
Matrici PAM
Matrici BLOSUM
10/5
2
Algoritmi di Allineamento Esatti
Allineamento Locale via DP
Allineamento Globale via DP
Algoritmo di Needlemann
Algoritmo di Smith Waterman
Valutazione della Complessità
11/5
2
Allineamento Euristico
FASTA
BLAST
Ricerca in Banche dati per Similarità
Valutazione della significatività
dell’allineamento
17/5
2
Allineamento multiplo di sequenze
Algoritmi di allineamento multiplo
Allineamento ottimale (MSA)
Allineamento euristico
- Progressivo
globale (CLUSTALW, Pileup)
locale (PIMA)
- Iterativo
globale (PRRP)
locale (DIALIGN)
CLUSTALW
18/5
2
Uso di Entrez per interrogare Swiss
prot, ricavare le proteine e ricercare
per similarità con BLAST su
Swissprot e le altre banche dati.
Esercitazione su BLAST e
CLUSTALW
24/5
2
Banche dati Biologiche
Banche dati di acidi nucleici
Banche dati di sequenze (SwissProt)
25/5
2
31/5
2
Rappresentazione secondaria e
terziaria delle proteine
Banche dati di strutture (PDB)
Visualizzazione di proteine usando
RASMOL
Tools per la gestione di file
contenenti la rappresentazione
secondaria
Allineamento Strutturale di
Proteine e Uso del software C alfa
match per l'allineamento strutturale
Allineamento Strutturale Multiplo
y Motivazioni
Esercitazione/Assegnamenti
individuali
Esercizio su BLAST e
CLUSTALW da svolgere a casa
Tesine
y Aldo Mauro: Predizione di
Strutture Terziarie (Ab Initio
folding)
y Cittadino : -omics
y
y
1/6
2
7/6
2
8/6
2
14/6
15/6
Tecniche
Clustering Gerarchico
Database di Famiglie di strutture
proteiche
y FSSP
y DALI
y SCOP
Mass Spectrometry-based
Proteomics
y Principi di funzionamento dello
spettrometro di massa (MS)
y Tipi di MS: MALDI MS e LC
MS
y Worflow di un esperimento
2D-GEL Proteomics
I dati generati da MS MALDI, LC,
2D-GEL
Preprocessing di dati MALDI
y Eliminazione rumore
y Riduzione numerosità dati
y Identificazione picchi
y Allineamento di picchi
Analisi di dati provenienti da MS
y Identificazione peptidi/proteine
y Estrazione di patterns
y Data Mining
Predizione di Strutture Secondarie
Predizione di Strutture Secondarie di
Proteine
Metodi Basati su Reti Neurali
Metodi Basati si Consensus
Cap /
Predizione di Strutture Terziarie
Homology Modeling
Threading
Ab Initio Folding
Esempi: Metodo di Heisemberg
Metodo di Rosetta
Server per la Predizione di Strutture
Swiss Model
Swiss PDB Viewer
y
Macri: Allineamento
Strutturale di Proteine
Progetti
y Argirò: Framework per
l'analisi di sequenze basato su
biojava
y Oliverio: Implementazione
Multithread di Alg.
Smith&Waterman
y Palmieri: Ricerca della
struttura secondaria
I ESERCIZIO
(i) Allineare Utilizzando il C alfa
Prothttp://bioinfo3d.cs.tau.ac.il/c_a
lpha_match/due proteine di PDB a
vostra scelta
(ii) visualizzare il risultato in
RASMOL ed inviare il PDB.
II ESERCIZIO
Allineare Utilizzando
http://bioinfo3d.cs.tau.ac.il/MultiPr
ot/quattro proteine di PDB a vostra
scelte
(ii) visualizzare il risultato in
RASMOL ed inviare il PDB.
Tesine
y Scarfone: Banche Dati
Biologiche
21/6
Sorgenti Dati in Genomica
Proteomica e Trascrittomica
Analisi di dati Proteomica
Caso di Studio
Analisi di dati Maldi TOF
22/6
28/6
Trascrittomica e Micro Array
Sequenziamento DNA
Visita al laboratorio di
sequenziamento DNA di Germaneto
29/6
Tesine:
y Aldo Mauro: Databases di Interazioni Proteina-Proteina
y Gianluca Scarfone: Databases di Sequenze biologiche
y A. Cittadino: Omics
y E. Macri: Allineamento Strutturale di Proteine
Progetti:
A. Palmieri: Sistema di retrieval della struttura secondaria memorizzata in odb
B. Oliverio: Smith Waterman Parallelo/Multithread
C. Argiro: Framework di analisi di sequenze in Biojava












