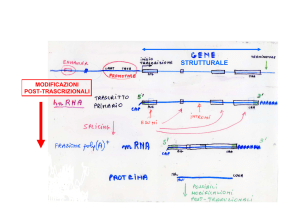
il flusso delle informazioni che parte dal DNA nel nucleo cellulare
e si concretizza nella sintesi proteica all’interno del citoplasma,
passando per l’intermediario fondamentale mRNA
RNA
produzione di fiori di petunia
con una colorazione più
vivace e intensa.
Le due piante in alto sono petunie wild-type; mentre le due in
basso contengono transgeni che inducono soppressione sia
del gene endogeno che del transgene, generando zone
bianche prive di pigmento nel fiore.
la down regulation è dovuta ad un’inibizione
post-trascrizionale dell’espressione genica
tramite un aumento della velocità di
degradazione dell’mRNA
Per
raggiungere
questo
scopo, introdussero in petunie
rosa alcune copie aggiuntive
del gene decodificante la
chalcone
synthase,
un
enzima
chiave
per
la
pigmentazione dei fiori nelle
petunie normalmente colorate
in rosa o viola.
molte piantine transgeniche
non avevano gli intensi colori
attesi ma presentavano fiori
con petali parzialmente o
completamente bianchi
Iniettarono
dsRNA
all’interno
di
Caenorhabditis elegans, un verme
nematode, individuando un potente
effetto di silenziamento.
Il termine RNA interference fu coniato
in questa occasione. Il C. elegans era un
sistema sperimentale utile poiché
l’origine evolutiva di tutte le cellule di
questo organismo è nota ed è possibile
iniettare RNA in embrioni ai primi stadi
ed osservare i cambiamenti rispetto al
modello durante lo sviluppo.
Effetto fenotipico dopo l’iniezione di RNA unc-22 a singolo o doppio filamento nella
gonade di C. elegans. Il gene unc-22 codifica per una proteina contenuta nelle
miofibrille muscolari. È noto che una diminuzione dell’attività di unc-22 provochi una
forte contrazione nei movimenti. L’iniezione di dsRNA, ma non di ssRNA, ha indotto le
contrazioni nella progenie
I principali risultati di tali esperimenti possono essere così
riassunti:
a)il silenziamento genico è indotto efficacemente mediante iniezione di dsRNA, invece
solo debolmente o per nulla in seguito all’iniezione di ssRNA senso o antisenso;
b) il silenziamento è specifico per l’mRNA omologo al dsRNA iniettato, altri mRNA non
sono coinvolti;
c)il dsRNA deve corrispondere alla sequenza dell’mRNA maturo, né la sequenza del
promoter né quelle relative agli introni inducono una risposta. Ciò indica
presumibilmente un meccanismo citosolico post-trascrizionale;
c)l’mRNA selezionato, ovvero omologo al dsRNA esogeno, scompare suggerendo una
sua degradazione;
c)soltanto poche molecole di dsRNA per cellula sono sufficienti a realizzare un
silenziamento totale. Ciò indica che il dsRNA è amplificato e/o agisce cataliticamente
piuttosto che in modo stechiometrico;
c)gli effetti del dsRNA possono diffondersi tra i tessuti e persino alla progenie,
suggerendo una trasmissione dell’effetto tra le cellule.
L’interferenza dell’RNA o RNAi
Processo mediante il quale RNAds silenzia, in maniera sequenza-specifica, l’espressione di
geni omologhi attraverso l’appaiamento con mRNA bersaglio seguito dalla sua
degradazione: silenziamento post-trascrizionale
Meccanismo biochimico conservato (dalle piante ai funghi, agli animali)
Probabilmente coinvolge più di 10 geni (analisi genetica) e gruppi di proteine correlate
Funzioni biologiche:
-Resistenza ai virus
-Silenziamento trasposoni
-Regolazione dell’espressione genica
La sua funzione antivirale è dimostrata chiaramente in piante (e insetti) dalla presenza nei
virus di geni che codificano soppressori dell’RNAi, importanti per la virulenza, mentre non è
ancora provata nei vertebrati
La sua funzione di silenziamento dei trasposoni è dimostrata dall’osservazione che
l’inattivazione di geni coinvolti nell’RNAi nei nematodi causa l’attivazione di molti
trasposoni della linea germinale
Cosa sono i miRNA
• Sono piccoli RNA non codificanti costituiti da circa 22
nucleotidi
• La loro scoperta risale al 1993
• Il primo mi-RNA fu identificato nel nematode
C.elegans
• Successivamente furono ritrovati sia nelle piante che
negli animali
• Oggi si ritiene che il numero complessivo di geni
codificanti per mi-RNA rappresenti circa lo 0.5-1%
del numero di geni codificanti per proteine
Funzione
• Agiscono a livello post-trascrizionale
• Inibiscono la traduzione dei propri
bersagli
Biogenesi
• Generalmente i geni codificanti per miRNA
sono trascritti nel nucleo dalla RNApolimerasi II che dà vita a lunghi trascritti,i
pri-miRNA,che presentano il cap e la
poliadenilazione
• Questi ultimi vengono processati da un’RNasi
III,Drosha,e dal suo cofattore,Pasha
• Si ottengono così trascritti di circa 70
nucleotidi,detti pre-miRNA
• Successivamente le proteine RAN-GTP ed
exportin 5 trasportano i pre-miRNA dal nucleo
al citoplasma,dove un’altra RNasi III,Dicer,li
processa per generare molecole di RNA
duplex di circa 22 nucleotidi,dette
miRNA:miRNA*
• Queste molecole vengono,poi,caricate nel
complesso miRISC (miRNA-associated
multiprotein RNA-induced silencing complex)
• I miRNA maturi a singolo filamento vengono
poi mantenuti all’interno del complesso
Meccanismo d’azione
• I miRNA possono avere due modi diversi
d’agire a seconda della
complementarietà che vi è tra il miRNA
e il suo target
1.Complementarità imperfetta
• I miRNA bloccano l’espressione dei loro
geni target a livello post-trascrizionale
• Per inibire la traduzione legano
solitamente le regioni non tradotte al
3’(3’ UTR), per le quali presentano
omologia
2.Complementarità perfetta
• I miRNA che legano i loro RNA bersaglio con
complementarietà perfetta inducono il taglio
del bersaglio che,quindi,non può più essere
tradotto
• In questo caso i miRNA trovano la loro regione
di omologia o nell’ORF(open reading frame) o
nella sequenza codificante
siRNA e miRNA
• GLI siRNA rappresentano un’altra classe
di piccoli RNA non codificanti
• Gli siRNA e i miRNA,pur presentando
numerose analogie in termini di
struttura e biogenesi,svolgono funzioni
essenzialmente diverse
• Gli siRNA agiscono principalmente
determinando la degradazione del mRNA
bersaglio attraverso un meccanismo definito
RNA interference(RNAi)
• Molecole di siRNA si producono a partire da
lunghe molecole di RNA bicatenario prodotte
da elementi genetici normalmente silenti o
estranei alla cellula,quali trasposoni,virus o
transgeni.
• RNAi perciò rappresenta un sistema di difesa
contro l’invasione di elementi genetici estranei
e di conservazione della stabilità del genoma
• siRNA agiscono solo per complementarietà
perfetta per cui ogni siRNA può avere un
unico mRNA bersaglio
• Diversamente,i miRNA costituiscono una
numerosa classe di geni endogeni
filogeneticamente conservati,la cui funzione è
di inibire l’espressione genica principalmente
attraverso l’inibizione della traduzione
• Ogni miRNA,per il suo caratteristico
meccanismo d’azione può avere più di un mRNA
bersaglio
• L’espressione temporale e tessutospecifica di miRNA suggerisce che i
miRNA svolgono un importante ruolo in
svariati processi biologici,quali sviluppo e
differenziamento cellulari.
• Inoltre alcuni possono avere una funzione
oncogena ed altri oncosoppressoria
DICER
Le ribonucleasi III sono enzimi
della classe delle idrolasi, che
catalizzano il taglio
endonucleolitico di un 5'-fosfomonoestere
DICER è coinvolta nella degradazione di lunghe molecole di
RNA a doppio filamento (dsRNAs) e di pre-microRNA (miRNA)
in filamenti di 21 nucleotidi (gli siRNA) durante il processo
della RNA interference
Ognuno presenta almeno cinque regioni specifiche:
una regione amminoterminale dipendente da ATP;
una regione a piattaforma carica positivamente (rosso);
una regione PAZ (Giallo), che si associa alla proteina Argonaute2;
due o più domini RNAsi III (verde);
una regione carbossiterminale in grado di legare molecole di dsRNA.
DICER
Si ritiene che la distanza tra la regione PAZ ed i domini RNAsi III sia di 65 angstrom, quanto
basta per alloggiare circa 25 nucleotidi.
Tale spazio sarebbe costituito proprio dalla regione piattaforma che, carica positivamente,
è in grado di instaurare legami elettrostatici con l'RNA. Secondo tale analisi, dunque, la
struttura di dicer potrebbe essere quella di una specie di righello molecolare, che
permetterebbe all'enzima di produrre frammenti di siRNA di dimensione estremamente
regolare.
Ago2
AGO2-contained within the RISC complex-contains two domains:
PIWI: The PIWI domain is a conserved domain within the Argonaute
family. The C-Terminus of this domain is required for endonuclease
activity.
PAZ: The PAZ domain is also a conserved domain within the Argonaute
family. It is located near the center of the AGO2 protein and provides
grooves substrate binding
RNA Interference (RNAi) is initiated by DICER an
RNase III
Enzyme, which uses an RNA
endonuclease activity to cleave double
stranded RNA into 20-23 nt fragments known
as miRNA. This cleavage is important because it
provides 3’overhangs on each strand of the
miRNA. miRNA binds to a heterodimer of R2D2
and Dcr-2 proteins. Once bound the R2D2/Dcr2 initiator complex is formed. The 5’end of
the RNA interacts with Dcr-2 forming the preRISC complex, which is the inactive form. TRBP
is an integral component of a Dicer-containing
complex. TRBP is required to recruit Ago2
protein to interact with the bound
miRNA. Ago2 protein actives the pre-RISC
complex, converting it into holo-RISC by
binding to the double stranded miRNA, which
is double stranded. Once Ago2 is bound, it
degrades one strand from the RNA (the
passenger strand), while selectively retaining
the guide strand. The holo-RISC complex
recognizes
mRNA
with
sequences
complementary to the guide strand and binds
to them. Once mRNA is bound, it is degraded.
Complesso RISC.
Il disegno che mostra l'RNA antisenso (il filo giallo nel
diagramma) legato al dominio PAZ, mentre in arancione si ha
l'mRNA ad esso complementare
microRNA as regulators of signal
transduction
miRNA regulation has been described in various physiological and
pathological processes, in particular developmental processes and
cancer
miRNA are important regulators of all hallmarks of cancer,
including cell growth and cell cycle control, evasion of apoptosis,
tissue invasion and metastasis, angiogenesis
miRNA are key players in signal transduction and partecipate in
various stages of the signal transduction process.
Signal transduction mediators respond to signals in a dosedependent manner. miRNA can serve as mediators of signaling
robustness either by amplifying a signal or by buffering and
balancing the signal response.
Analisi in silico mediante
algoritmo TargetScan 5.1
Akt signaling in prostate cancer
Akt,
also known as Protein Kinase B
(PKB), is a serine/threonine protein
kinase that plays a key role in multiple
cellular processes such as glucose
metabolism,
cell
proliferation,
apoptosis, transcription and cell
migration.
BAD: pro-apoptotica
CDKN1A: cyclin-dependent kinase
inhibitor 1A
mTOR: serine/treonine kinase
PTEN: phosphatase
VHL signaling in renal cell carcinoma
EP300: E1A binding protein p300
CBP: CREB binding protein
HIF1: Hypoxia inducible factor 1
VEGF signaling in renal cell carcinoma
Applicazioni:
siRNA nella ricerca di base
RNA a doppio filamento viene sintetizzato con una sequenza complementare
al gene d’interesse e introdotto in una cellula o organismo, dove è riconosciuto
come materiale genetico esogeno e attiva il processo della RNAi.
Effetto: drastico calo nell’espressione del gene target e studiando gli effetti di
questa diminuzione si può ricavare il ruolo fisiologico del gene prodotto.
Ovviamente questa tecnica non è applicabile al silenziamento di geni
essenziali la cui inattivazione comprometterebbe la vitalità della cellula. Dal
momento che RNAi non abolisce totalmente l’espressione del gene viene
chiamata tecnica knockdown.
siRNA in biotecnologia
sviluppo di piante che producano bassi livelli di tossine
naturali così che possano essere introdotte
nell’alimentazione.
1) i semi di cotone sono ricchi delle proteine che devono essere assunte con la dieta,
ma contengono un prodotto tossico, il gossipolo, rendendoli inutilizzabili per il
consumo umano. RNAi è stata utilizzata per produrre stock di cotone i cui semi
contengono ridotti livelli di delta-cadinene synthase, un enzima chiave nella
produzione di gossipolo, senza influenzare la produzione dell’enzima in altre parti
della pianta dove il gossipolo è importante nella prevenzione di danni da parassiti
delle piante .
2) riduzione dei livelli di allergeni nelle piante di pomodoro e la riduzione dei
precursori di carcinogeni nelle piante di tabacco.
3) studio recente di un nuovo un anti-zanzare.
Si utilizzano nanoparticelle per somministrare RNA a doppio filamento alle larve
di zanzara nel momento in cui si cibano. l’interferenza RNA è stata utilizzata per
silenziare
il gene responsabile della produzione della chitina, la principale
componente dell’esoscheletro negli insetti, nei crostacei e negli aracnidi.. Poiché il
dsRNA si diluisce
velocemente, non può in teoria essere somministrato
direttamente e per questo si ricorre all’utilizzo delle nanoparticelle. Il gruppo ha
siRNA nella ricerca medica
l’interferenza dell’RNA non si verifica direttamente sul DNA, ma
solo sulla sua copia (mRNA): potrebbero essere scavalcati tutti i
problemi legati all’etica
l’intervento non comporterebbe la sostituzione in blocco del gene
malato, ma la distruzione del suo intermedio di RNA, bloccando la
produzione della proteina responsabile della disfunzione.
Interrompendo il flusso dell’informazione “malata” si interverrebbe
pertanto in modo etico e queste terapie farmacologiche sarebbero
considerate alla stregua dei medicinali attualmente in commercio.
Attualmente si stanno sviluppando diverse terapie basate su RNAi
tanto che recentemente sono stati riportati i successi di numerosi
esperimenti pilota. Questi sviluppi recenti nelle applicazioni
terapeutiche di RNAi sono senza dubbio imputabili al
superamento di problematiche correlate con la veicolazione in
vivo dei siRNA.
Problematiche riguardanti la veicolazione in vivo
Un’iniezione di nanoparticelle deve:
evitare la filtrazione, fagocitosi e
degradazione nel flusso sanguigno (a);
essere trasportato attraverso l’endotelio
del vaso sanguigno (b);
diffondere nella matrice extracellulare ©;
essere internalizzato dentro la cellula (d);
evitare l’endosoma (e);
e rilasciare i siRNA (f) [71].
Barriere fisiologiche alla distribuzione
sistemica di nanoparticelle di siRNA.
Varie strategie di veicolazione in vivo di farmaci basati su siRNA sia veicolazione
specifica che non specifica.
modificazioni chimiche a livello della posizione 2’ del
ribosio, per esempio mediante metilazione o fluorurazione,
in quanto portano ad un aumento della stabilità dei siRNA
iniezione intravenosa di siRNA chimicamente modificati o
coniugati con il colesterolo o immagazzinati all’interno di
particelle liposomiali protettive:
uptake cellulare del siRNA attraverso il processo di
endocitosi mediata recettorialmente
incorporare l’acido nucleico in una carrier:
Attenzione alle caratteristiche di superficie!
L’aggiunta di PEG può migliorare le interazioni di carica e le
dimensioni per prevenire le aggregazioni.
accoppiamento dei siRNA terapeutici con frammenti di
anticorpi e aptameri o impacchettati in nanoparticelle
rivestite con ligandi che hanno come bersaglio i recettori:
incorporazione dei siRNA tramite l’endocitosi.
È possibile progettare ligandi strutturati di RNA noti come
aptameri che possono legarsi con specifici recettori della
superficie cellulare e coniugarli covalentemente a siRNA per
il rilascio cellulo-specifico in vivo. Le molecole di siRNA
vengono rilasciate dall’aptamero durante il processo di
ingresso all’interno della cellula.
Vectors for shRNA expression
• Plasmid vector
• Lentiviral vector
-genomic insertion
-permanent
The long terminal repeat (LTR) is the control center for gene expression.
• Adenoviral vector
- transient expression
- no genomic insertion