DNA non codificante
ncDNA
Teorie sul ruolo genetico
RNAi e miRNA
Liberamente tratto dalla tesina del Dr. Emiliano Mancini
ncDNA
•
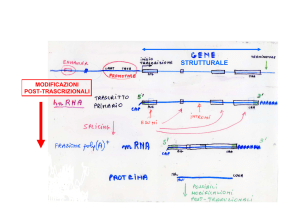
Per ncDNA si intende il DNA intronico, intergenico e altre zone non
codificanti del genoma.
•
ncDNA è caratteristico degli eucarioti:
– Sequenze codificanti → 1.5% del genoma umano
– Introni → in media 95-97% del gene codificante
– Almeno un terzo del genoma umano viene trascritto
•
L’evoluzione successiva dello spliceosoma ha facilitato la diffusione
degli introni negli eucarioti più complessi.
•
Nei procarioti si ha una piccolissima parte di ncDNA perché i processi
di trascrizione e traduzione sono quasi simultanei
•
ncDNA nei procarioti è meno dell’1%
Attività genetica:
tradizionalmente si riteneva...
Procarioti
Eucarioti
ncDNA
Ipotesi sul ruolo del ncDNA:
• facilita il processo di riassortimento
• è presente per motivi strutturali
• è una traccia dell’assemblamento casuale prebiotico
Il ncDNA dopo l’escissione viene semplicemente degradato e riciclato
Alcune regioni del genoma codificano per l’rRNA e il tRNA necessari
alla sintesi proteica
I genomi sequenziati di batteri e archeobatteri sono costituiti principalmente da
sequenze codificanti affiancate da regioni di controllo dell’espressione. .
...attualmente si ritiene
Eucarioti
RNA intronici ed esonici interagendo
con altre molecole possono dirigersi
selettivamente verso bersagli posti su
altre molecole di DNA ed RNA
Sono state identificate migliaia di
sequenze di RNA che vengono
trascritte e non tradotte in proteine.
Inutile spreco energetico?
RNAi
Il meccanismo dell’RNA interference, scoperto nel 1998 da studi sul C.Elegans1,
segue la scoperta delle capacità di gene silencing dell’RNA antisenso:
una molecola artificiale di RNA (single strand) che si lega all’mRNA e ne
impedisce la traduzione in proteina.
RNAi è in grado di combattere infezioni di RNA virus per cui si pensa che si sia
evoluto per proteggere le cellule eucariotiche contro forme invasive di acidi
nucleici
Caratteristiche importanti dell’RNAi:
• RNAi si diffonde nell’individuo e può essere trasmesso alla progenie
• Solo poche molecole di dsRNA (double-strand) sono sufficienti ad
innescare il meccanismo di RNAi presenza di componenti
catalitiche di amplificazione
• RNAi agisce a livello post-trascrizionale poiché dsRNA
corrispondenti a sequenze introniche non attivano l’RNAi
• RNAi è altamente specifico: l’iniezione di dsRNA omologo a
sequenze esoniche specifiche di un gene eliminano o riducono solo
l’mRNA corrispondete a quel gene particolare.
1Caenorhabditis
elegans è un verme lungo circa 1 mm, che vive nel suolo, in regioni temperate.
Micro-RNA
miRNA sono una classe di piccoli RNA non codificanti che si trovano nei genomi
degli eucarioti.
Nel genoma si trovano negli introni o in regioni non codificanti come singoli geni
o in cluster di vari miRNA diversi entro il raggio di alcune kilobasi
miRNA sembrano coinvolti nella
regolazione dei geni attraverso
vari meccanismi simili all’RNA
antisenso e all’iRNA che portano
al blocco della traduzione o alla
degradazione del mRNA.
miRNA trovati negli
invertebrati si trovano anche
nei vertebrati ma non
viceversa.
Micro-RNA
miRNA vengono trascritti dal DNA come
lunghi precursori primari (pri-miRNA), nel
caso di ammassi di miRNA come
polycistronic RNA con una distinta struttura
secondaria contenente diversi stem.loops
imperfetti
La maturazione dei miRNA richiede
almeno altri due passi di processazione:
• La ribonucleasi III Drosha taglia gli
stem-loops dal pri-miRNA dando luogo a
miRNA precursori (pre-miRNA o stRNA)
lunghi circa 70-80 nucleotidi
• Nel citoplasma il Dicer un’endonucleasi
di tipo III escinde il miRNA maturo (circa
22 nucleotidi) dal stRNA
RNAi
4 stadi:
1. Dicer taglia il dsRNA in
frammenti a doppia elica
lunghi 21-25 nucleotidi
(siRNA)
2. Gli siRNA vengono
incorporati in un
complesso detto RISC
(RNA-induced silencing
complex)
3. Attivazione del RISC
mediante la separazione
delle due catene
4. Degradazione di mRNA
complementare allo
strand di guida del
siRNA presente nel
RISC
5. Si ha un ulteriore step che varia a seconda degli organismi.
Questi siRNA secondari vengono generati durante un’amplificazione
ciclica nella quale l’RdRp (RNA-dependent RNA polimerase)
viene direzionata sul mRNA bersaglio dai siRNA esistenti
RNAi
4 stadi:
1. Dicer taglia il dsRNA in
frammenti a doppia elica
lunghi 21-25 nucleotidi
(siRNA)
2. Gli siRNA vengono
incorporati in un
complesso detto RISC
(RNA-induced silencing
complex)
3. Attivazione del RISC
mediante la separazione
delle due catene
4. Degradazione di mRNA
complementare allo
strand di guida del
siRNA presente nel
RISC
5. Si ha un ulteriore step che varia a seconda degli organismi.
Questi siRNA secondari vengono generati durante un’amplificazione
ciclica nella quale l’RdRp (RNA-dependent RNA polimerase)
viene direzionata sul mRNA bersaglio dai siRNA esistenti
Conseguenze nuova genetica
• Malattie come l’epilessia e l’autismo potrebbero essere legate
ad errori nelle zone di ncDNA
• Capire come funzionano i miRNA potrebbe dare indicazioni
importanti sul meccanismo di differenziazione cellulare
•
•
•
Zofia Szweykowska-Kuliñska, Artur Jarmowski and Marek Figlerowicz, RNA
interference and its role in the regulation of eucaryotic gene expression, Acta
Biochimica Polonica, Vol. 50 No. 1/2003, p 217–229
Andrea Tanzer. Jorg Lehmann, Peter F. Stadler, STATISTICAL EVIDENCE FOR
SPECIFIC EXPANSION OF THE miRNA REPERTOIRE IN VERTEBRATES
John S.Mattick , RNA regulation: a new genetics?, NATURE REVIEWS |
GENETICS, VOLUME 5 | APRIL 2004,p316-323