© Pietro Palatini
Dipartimento di Farmacologia ed Anestesiologia
Università degli Studi di Padova
A.A.2009/2010
RNA-interferenza
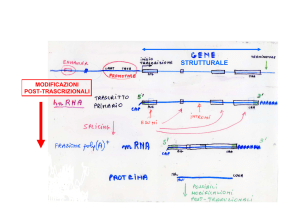
Definizione: inibizione dell’espressione genica a livello post-trascrizionale da parte di
brevi RNA a doppio filamento (gene silencing). Questa repressione genica viene attuata
mediante scissione dell’mRNA o impedendo il processo di traduzione nei ribosomi.
Solo una piccola parte del genoma trascrive RNA codificanti (RNA messaggero, RNA
transfer e RNA ribosomiale, che concorrono al processo di traduzione nei ribosomi), il
resto trascrive una grande varietà di RNA non codificanti. Tre di questi, che sono brevi
RNA a doppio filamento, sono responsabili del fenomeno della RNA-interferenza:
1) siRNA (small interfering RNA; brevi RNA interferenti): sono generati all’enzima Dicer
(ribonucleasi), che forma doppi filamenti di 21-23 nucleotidi a partire da lunghe
catene a doppio filamento, perfettamente appaiate, di RNA esogeno (virus) o
endogeno (codificato dai trasposoni) (Fig. 1-3). Si ritiene che gli siRNA si siano evoluti
sia come meccanismo di difesa contro le infezioni da virus a RNA, sia per controllare
l’attività dei trasposoni.
2) miRNA (microRNA): sono codificati dal genoma tramite l’RNA polimerasi II, la quale
trascrive pri-miRNA (microRNA primario), che ha forma di forcina (hairpin), da cui il
nome sh-RNA (short hairpin RNA), e non è perfettamente appaiato. Viene scisso da
una RNAasi II, detta Drosha a pre-miRNA (microRNA precursore), lungo circa 70 basi.
Il pre-miRNA viene trasferito nel citoplasma da una proteina detta esportina e scisso
dal Dicer a mi-RNA (di circa 22 basi), contenente un mismatch (spaiamento). La
cellula contiene circa 1000 miRNA, che hanno la funzione di regolare l’espressione
genica (la soppressione di questa funzione regolatrice non è compatibile con la vita)
(Fig. 3).
Una volta formati, siRNa e miRNA vengono incorporati nel complesso RISC (RNAinduced silencing complex, Fig. 4) costituito da proteine chiamate Argonauti.
L’appaiamento perfetto dell’siRNa con l’mRNA produce scissione dell’mRNA da parte di
una proteina del RISC, detta slicer. Il miRNA produce scissione dell’mRNA o previene il
processo di traduzione a seconda del livello di complementarietà. A causa
dell’appaiamento imperfetto, un singolo miRNA può ibridizzarsi con circa 100 mRNA
diversi. Questa mancanza di specificità impedisce, per ora, di sfruttarli come farmaci.
3) piRNA (RNA associati a proteine piwi). Sono brevi RNA, lunghi 24-30
nucleotidi,
generati da precursori a singolo, lungo filamento, mediante un processo
indipendente da Drosha e Dicer. Funzionano in associazione alla sottofamiglia Piwi
delle proteine Argonauti, espresse solo nelle cellule germinali, e hanno come
bersaglio l’RNA a doppio filamento codificato dai trasposoni. Questa attività di
controllo della codificazione mediata dai trasposoni è essenziale per lo sviluppo delle
cellule germinali.
Utilizzazione degli siRNA
A scopo terapeutico, non si possono introdurre nelle cellule di mammiferi doppi
filamenti di RNA lunghi (più di 30 basi), perché attivano il complemento, che causa
inibizione non specifica di trascrizione e traduzione. Si utilizzano quindi siRNA di sintesi.
Per introdurli nella cellula si usano vettori vari (liposomi, molecole lipofile, come
colesterolo, che vengono coniugate con gli siRNA, ecc.). Gli siRNA sono da 100 a 1000
volte più potenti dei nucleotidi antisenso e quindi è sufficiente che una quantità molto
più piccola penetri nella cellula. Attualmente sono in sperimentazione clinica vari siRNA
per il trattamento della degenerazione maculare o dell’infezione da virus respiratorio
sinciziale.
L’inattivazione funzionale di un gene, mediante somministrazione di siRNA, costituisce
un metodo alternativo alla delezione genica (organismi knockout) per individuare la
funzione del gene.