F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
SCHEDA ATTIVITÁ
Durata: 36 mesi
DATI GENERALI
Titolo dell'attività
proposta
CoSMiC: Coupling Smart Molecules into Chips
µ-Biochips per l’analisi chimica e biologica
Workpackage
WP7 Substask 1.11 - Verifica
Dipartimento/Istituto
che svolge l'attività
Scienze del sistema terra e tecnologie per l'ambiente
IIA - Istituto sull’Inquinamento Atmosferico
Responsabile
scientifico
Roberto Pilloton - 3313291173 – [email protected]
Staff
IBBR - Dr.D.Pignone, (Ba) / Scienze bio-agroalimentari
IBBR - Dr.F.Mercati – (Pa) / Scienze bio-agroalimentari
IBBR - Dr.Roberto Defez (Na) / Scienze bio-agroalimentari
IC – Dr. G.Rea, (ARRM1) / Scienze chimiche e tecnologie dei materiali
IIA – Dr.Virginia Andreoli / Scienze del sistema terra e tecnologie per l'ambiente
ISA - Dr M.Staiano, (Av) / Scienze dell’alimentazione
ISM – Dr.V.Foglietti, (AdRM1) / Scienze fisiche e tecnologie della materia
IFN – Dr.A.Notargiacomo (Roma)/ Scienze fisiche e tecnologie della materia
Convenzione IIA- DIIN (UniSa) / Prof. M.Di Matteo, Dr. D. Albanese
di alimenti contenenti derivati di OGM basata
sull'uso di un micro-biochip muliti-analita
V.Andreoli
S.D’Auria
F.Carimi
R.Defez
A.Notargiacomo
V.Foglietti
F.Mercati
D.Pignone
N.Pirrone
M.L.Pea
L.Ragazzi
R.Pilloton
G.Rea
M.Staiano
M.Saviano
D.Albanese
Dipartimenti
coinvolti (5)
Istituti Coinvolti
(6)
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 1
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
Stato dell’arte
I biosensori sono già applicati nel settore. Un enzima, un anticorpo, un
oligonucleotide, un batterio o un organello, interagiscono con l'analita per fornire
un segnale elettrico misurabile da un trasduttore. Biosensori a colinesterasi o
fotosistema rivelano pesticidi ed erbicidi nei prodotti agricoli, carboidrati, alcoli,
amminoacidi, ammidi, ammine, composti eterociclici, acidi carbossilici, gas, ioni
inorganici, cofattori, alcoli e fenoli nei prodotti alimentari (vino, birra e yogurt).
I biosensori microbici sono selettivi nella rilevazione di ammoniaca, metano e
del BOD per la qualità delle acque reflue (lattiero-caseario). Gli immunosensori
rivelano vitamine idrosolubili, contaminanti chimici (BPA, PAA, PCB) e
microrganismi patogeni presenti nella carne, pollame, uova e pesce. Specifiche
sequenze oligonucleotidiche sono impiegate per analizzare alimenti soggetti a
frodi, per stabilire la tracciabilità di un alimento, per escludere l’impiego di
OGM.
Word Cloud
Criticità
Attività e obiettivi
Si tratta di una ricerca mutidisciplinare da condurre con diverse figure
specialistiche di chimica, fisica, ingegneria elettronica, biologa molecolare,
Competenze presenti in diversi istituti e dipartimenti del CNR. Se da un lato può
considerarsi una sfida dall’altro c’è il rischio di una Babele.
Il µ-biochip appare come uno strumento a lettura immediata, facilmente
personalizzabile e flessibile a diverse esigenze, semplicemente sostituendo il
chip di lettura. Questo è spesso quello che accade nei laboratori e che oggi trova
risposta nell’acquisto di molta e differente strumentazione, spesso di costo
elevato. Tale circostanza di fatto pone un forte limite alle potenzialità di sviluppo
di molti laboratori di analisi e di ricerca. Le barriere per questa tecnologia sono
rappresentate dalle dimensioni e dal numero degli elettrodi nell’array. Infatti
l’impiego delle misure amperometriche come metodo di rivelazione se da una
parte semplifica il sistema e lo rende economicamente vantaggioso, dall’altra
risente della superficie dell’elettrodo che non può essere ulteriormente
miniaturizzata in quanto la corrente misurata è direttamente proporzionale alla
superficie elettrodica. Per questo motivo sono state individuate applicazioni che
non richiedono un gran numero di elettrodi (32, 49, comunque non superiore a
256). E’ utile puntualizzare che la strumentazione che abbraccia questa fascia di
mercato è estremamente poco conveniente in termini di spese di esercizio in quei
laboratori di analisi o di ricerca dove le problematiche sono di norma molto
inferiori all’acquisizione di un intero genoma.
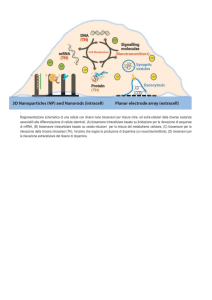
I nostri dispositivi bioelettronici usano materiali attivi dispersi in inchiostri per
ottenere strati di conduttori (argento, grafite, grafite drogata con platino, oro,
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 2
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
Impatto atteso
Potenziale
innovativo
nano tubi di carbonio o altri materiali nanostrutturati), isolanti, membrane e
molecole biologiche. Si ottengono biosensori del tipo “usa e getta” anche se la
durata media in condizioni di utilizzo è di circa un mese. I biosensori stampati
dotati di piste conduttive ed elettrodi su materiale plastico flessibile permettono
analisi quali-quantitative molto rapide sul campo con procedure drop-on e sono
adatti alle analisi di screening. Fungono da EWS (early warning system) per
segnalare situazioni di pericolo e avviare controlli analitici più dispendiosi
(tempo e danaro, apparecchiature, mano d’opera esperta, uso di solventi) solo su
un ridotto numero di campioni. Si vuole dunque sviluppare un dispositivo a µchip flessibile, versatile e utilizzabile in diversi settori. E’ stata già sviluppata
una procedura di immobilizzazione originale che consente di indirizzare su
ciascun elettrodo di un µ-array biomolecole differenti applicando un potenziale
elettrochimico. La reversibilità dell’immobilizzazione consente inoltre il rinnovo
dello strato biologico quando le biomolecole non sono più attive o se cambia il
target della determinazione analitica. Con biomolecole ingegnerizzate,
l’immobilizzazione può essere orientata in modo da esporre il sito attivo della
biomolecola alla soluzione contenente il campione, ottenendo una migliore
efficienza del monostrato biologico. L’uso di un braccio spaziatore per
l’immobilizzazione consente l’electron transfer senza la necessità di mediatori,
superando lo stadio diffusivo lento della reazione enzimatica e quindi passando
da 2-5 min a pochi secondi per la risposta. L’uso del braccio spaziatore sortisce
effetti positivi anche sul comportamento della biomolecola immobilizzata che
conserva le proprietà espresse in vivo senza risentire del microambiente
sottostante.
Con gli array di biosensori è possibile immobilizzare numerose molecole
biologiche differenti per un finger print analitico del campione. Un µ-biochip
multi-analito può anche realizzare analisi in serie. Nel monitoraggio della
vinificazione, ad esempio, l’array di sensori può ospitare gli enzimi per la
determinazione contemporanea di acido malico, glicerolo, glucosio, fruttosio,
etanolo, acido lattico, polifenoli e potere antiossidante, ciascuno con molteplicità
3 per acquisire la media e la deviazione standard. Un’altra applicazione riguarda
l’efficienza di enzimi sintetici, ottenuti mediante tecniche di ingegneria genetica,
rispetto ad enzimi wild type. E’ possibile studiare le cinetiche di enzimi
ricombinanti nelle stesse condizioni sperimentali, senza ricorrere ai saggi
tradizionali che, per piccoli quantitativi di enzima ricombinante, pongono grandi
difficoltà e limitazioni. Infine, l’applicazione più interessante del µ-biochip è la
verifica di alimenti contenenti derivati di OGM
La ricerca di OGM negli alimenti è condotta in laboratori appositamente
attrezzati. Il µ-biochip ha costi ridotti, è trasportabile, riutilizzabile e consente la
misura di diversi OGM contemporaneamente in pochi secondi, con una risposta
di tipo quantitativo e di facile lettura. Non necessita di un laboratorio
appositamente attrezzato e può essere configurato come uno strumento portatile.
Una volta addestrato, anche personale avulso dalle tecniche di biologia
molecolare può ottenere validi risultati. Occorrono piccoli quantitativi di DNA
estratti da piante, farine, matrici complesse o derivati alimentari, anche in
condizioni non ideali, come quelle nei magazzini o supermarkets, mediante
piccoli apparecchi e appositi kit già in commercio. Oggi per i tempi lunghi, i
costi e per il sovraccarico dei laboratori certificati, il numero di indagini
compiute è piuttosto limitato. Un laboratorio mobile attrezzato può effettuare
uno screening per escludere i campioni negativi dagli accertamenti certificati.
(PIANO NAZIONALE DI CONTROLLO UFFICIALE SULLA PRESENZA DI ORGANISMI
GENETICAMENTE MODIFICATI NEGLI ALIMENTI, 2013)
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 3
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
Progetto
Multidisciplinare
Progetto
Interregionale
CoSMiC - Distribuzione regionale degli Istituti coinvolti
Ripartizione di
genere
Al CNR il genere femminile si attesta al 44% del personale distribuito per il 39%
nella ricerca e per il 75% nell’amministrazione (2011). In questa proposta le
ricercatrici sono espresse al 50%.
:
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 4
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
Milestones &
Deliverables
Tempistica Milestone
6 Mesi
M6
-Selezione da banche dati di
aptameri , identificazione e
produzione di stabili sequenze
nucleotidiche di tipo PNA
specifiche per sequenze OGM
12 Mesi
-Design e produzione di elettrodi
stampati monouso
-250 elettrodi stampati
monouso
-Design della fluidica e del
microchip con 16 o 32
microelettrodi
M12
-Selezione di sonde fluorescenti o
proteine redox per la marcatura
delle sequenze identificate;
-1 Microchip con
microfluidica
-Scelta dei materiali elettrodici
nanostrutturati da depositare sugli
elettrodi stampati monouso e sul
microchip (nanoparticelle di oro,
TiO2 nanostrutturato).
24 Mesi
36 Mesi
Deliverable
D6
-Identificazione di almeno tre
sequenze di aptameri e
produzione di circa 10 mg di
PNA specifiche per
determinati target alimentari.
-Scelta di intercalanti elettroattivi.
M24
-Sviluppo di biosensori basati su
voltammetria ad impulso
differenziale, polarizzazione di
fluorescenza e/o risonanza di
plasmone superficiale
per la determinazione di sequenze
OGM.
M36
-Immobilizzazione, sviluppo e
validazione dei biosensori con
saggi in matrici reali
D12
-Circa 10 mg di PNA
fluorescenti e tre sequenze
specifiche di aptameri per la
rivelazione elettrochimica.
-150 elettrodi stampati nano
strutturati
-1 microchip multi elettrodo
nano strutturato con
microfluidica
D24
-Protocolli sperimentali per lo
sviluppo di biosensori con
sonde fluorescenti, proteine
redox e/o intercalanti
elettroattivi.
D36
-Protocolli sperimentali per
biosensori multi analita e con
rivelazione multipla
(fluorescenza ed
elettrochimica) in saggi con
matrici reali.
-Microchip elettrochimico
multianalita
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 5
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
References Albanese D., Di Matteo M. and Pilloton R. (2012) Quantitative screening and
resolution of carbamic and organophosphate pesticides mixture in extra virgin olive
oil by acetylcholinesterase -choline oxidase sensor - Journal of Environmental
Science and Engineering A 1 (2012) 68-77
Albanese D., Malvano F., Sannini A., Pilloton R. and Marisa Di Matteo; (2014) A
Doped Polyaniline Modified Electrode Amperometric Biosensor for Gluconic Acid
Determination in Grapes; Sensors 2014, 14, 11097-11109; doi:10.3390/s140611097
ISSN 1424-8220
Albanese D., Sannini A., Malvano F., Pilloton R., Marisa Di Matteo, (2014)
Optimisation of Glucose Biosensors Based on Sol–Gel Entrapment and Prussian
Blue-Modified Screen-Printed Electrodes for Real Food Analysis; Food Anal.
Methods (2014) 7:1002 – 1008
Andreoli V, De Marco EV, Trecroci F, Cittadella R, Di Palma G, Gambardella A. J
Neural Transm. 2014 Potential involvement of GRIN2B encoding the NMDA
receptor subunit NR2B in the spectrum of Alzheimer's disease. May; 121(5):53342.
Andreoli V, Trecroci F, La Russa A, Cittadella R, Liguori M, Spadafora P, Caracciolo
M, Di Palma G, Colica C, Gambardella A, Quattrone A. (2011) Presenilin enhancer-2
gene: identification of a novel promoter mutation in a patient with early-onset familial
Alzheimer's disease. (2011) Alzheimers Dement. Nov; 7(6):574-8.
Bianco C, Defez R (2010) Improvement of Phosphate Solubilization and Medicago
Plant Yield by an Indole-3-Acetic Acid-Overproducing Strain of Sinorhizobium
meliloti. Appl. Environ. Microbiol. 76: 4626-4632
Bianco C, Senatore B, Arbucci S, Pieraccini G, Defez R. (2014) Modulation of
Endogenous Indole-3-Acetic Acid Biosynthesis in Bacteroids within Medicago sativa
Nodules. Appl Environ Microbiol. 80: 4286-4293
Catalano D, Pignone D, Sonnante G, Finetti-Sialer MM (2012) In-silico and in-vivo
analyses of EST databases unveil conserved miRNAs from Carthamus tinctorius and
Cynara cardunculus. BMC Bioinformatics 13: S12
De Paola D, Cattonaro F, Pignone D, Sonnante G (2012) The miRNAome of globe
artichoke: conserved and novel micro RNAs and target analysis. BMC Genomics 13:
41
Defez R. (2014) Il caso OGM. Il dibattito sugli organismi geneticamente modificati.
Collana: Città della scienza, Carocci editore pp148
Husu I., Rodio G., Touloupakis E., Lambreva M.D., Buonasera K., Litescu S.C.,
Giardi M.T., Rea G. (2013) . Insights into photo-electrochemical sensing of
herbicides driven by Chlamydomonas reinhardtii cells. Sensors and Actuators B:
Chemical, 185:321–330, DOI
Ilie M., R Dejana R., Foglietti V., Renda R., Nardi L, Masci A., Lanza B., Montereali
MR, Della Seta L., Vastarella W, Pilloton R. (2010) Engineering a continuous flow
electrochemical micro-cell for biosensor application: new achievements- Intern. J.
Environ. Anal. Chem. Vol. 90, No. 1, 15 January 2010, 31–39
Lavecchia T., G. Rea, A. Antonacci, and M.T. Giardi. Healthy and Adverse Effects of
Plant-Derived Functional Metabolites- the Need of Revealing Their Content and
Bioactivity in a Complex Food Matrix (2013). Critical Review in Food Science and
Nutrition 53(2):198-213. doi: 10.1080/10408398.2010.520829.
Maly J., Ilie M., Foglietti V., Cianci E., Minotti A., Nardi L., Masci A., Vastarella W.
and Pilloton R. (2005) Continuous flow micro-cell for electrochemical addressing of
engineered bio-molecules - Sensors and Actuators B: Chemical (2005) 111, 317-322
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 6
F3 - PROGETTO PREMIALE INTERDIPARTIMENTALE “FOOD FOR FUTURE”
Maly J., Masojidek J, Masci A., Ilie M., Cianci E., Foglietti V., Vastarella W., Pilloton
R.(2005) Direct mediatorless electron transport between photosystem II and
poly(mercapto-p-benzoquinone) modified gold electrode: new design of biosensor for
herbicide detection, - Biosens & Bioelec (2005) 21 (6) 923-932
Mercati F, Riccardi P, Leebens-Mack J, Abenavoli MR, Falavigna A, Sunseri F
(2013) Single nucleotide polymorphism isolated from a novel EST dataset in garden
asparagus (Asparagus officinalis L.). Plant Science 203–204:115–123
Mercati F, Leone M, Lupini A., Sorgonà A, Bacchi M, Abenavoli MR, Sunseri F
(2013) Genetic diversity and population structure of a common bean (Phaseolus
vulgaris L.) collection from Calabria (Italy). Genet Resour Crop Evol, 60(3):839-852
Montereali MR., Della Seta L., Vastarella W., Pilloton R. (2010) A disposable
Laccase Tyrosinase based biosensor for amperometric detection of phenolic
compounds in wine Journal of Molecular Catalysis B: Enzymatic 64 (2010) 189–194
Pignone, D., & Hammer, K. (2013). Conservation, Evaluation, and Utilization of
Biodiversity. In Kole C.: Genomics and Breeding for Climate-Resilient Crops (pp. 926). Springer Berlin Heidelberg.
Scognamiglio V, Stano P, Polticelli F, Antonacci A, Lambreva MD, Pochetti G, Giardi
MT, Rea G. (2013) Design and biophysical characterization of atrazine sensing
peptides mimicking the Chlamydomonas reinhardtii plastoquinone binding niche.
Phys. Chem. Chem. Phys. 15, 13108-13115.
Sorgonà A, Lupini A, Mercati F, Di Dio L, Sunseri F, Abenavoli MR (2011) Nitrate
uptake along the maize primary root an integrated physiological and molecular
approach. Plant Cell Environ 34:1127-1140
Spadafora P, Liguori M, Andreoli V, Quattrone A, Gambardella A. (2012) CAV3
T78M mutation as polymorphic variant in South Italy. Neuromuscul Disord.
Jul;22(7):669-70.
Staiano M., Matveeva EG, Rossi M, Crescenzo R, Gryczynski Z, Gryczynski I,
Iozzino L, Akopova I, D’Auria S. (2009); Nanostructured silver-based surfaces: new
emergent methodologies for an easy detection of analytes. ACS Applied Materials &
Interfaces 1(12): 2909-2916
Staiano M., Scognamiglio V, Mamone G, Parracino A, Rossi M, D'Auria S. (2006);
Glutamine-binding protein from Escherichia coli specifically binds a wheat gliadin
Peptide. 2. Resonance energy transfer studies suggest a new sensing approach for an
easy detection of wheat gliadine. J Proteome Res. 5(9): 2083-6
Staiano M., Strianese M, Varriale A, Di Giovanni S, Scotto di Mase D, Dell'Angelo
V, Ruggiero G, Labella T, Pellecchia C, D'Auria S. (2012); D-Serine-Dehydratase
from Saccaromyces cerevisiae: A Pyridoxal 5 '-phosphate-Dependent Enzyme for
Advanced Biotech Applications. Protein Pept Lett 19(6): 592-5 2012
Vastarella W., Maly J., Ilie M., Pilloton R. (2009) INTEGRATION BETWEEN
TEMPLATE-BASED NANOSTRUCTURED SURFACES AND BIOSENSORS Biosensing Using Nanomaterials, (2009) 377-419
Vastarella W., Rosa V., Cremisini C., Della Seta L., Montereali MR., Pilloton R.
(2007) A preliminary study on electrochemical biosensors for the determination of
total cholinesterase inhibitors in strawberries - International Journal of Environmental
and Analytical Chemistry 87 (10-11) 2007, 689-699
CoSMiC: Coupling Smart Molecules into Chips - µ-Biochips per l’analisi chimica e biologica
Pagina 7