Relazione 08/10/02
Alcuni biosensori sono in grado di rispondere a reazioni enzimatiche in sequenza, e sono, perciò,
chiamati multienzimatici. Un particolare esempio applicativo è il biosensore a neuroaminidasi,
progettato per sviluppare nuovi farmaci antinfluenzali.
I virus dell’influenza di tipo A e B necessitano dell’acido sialico, quale componente fondamentale
del recettore cellulare, per l’attacco alla superficie della cellula ospite. Tale attacco viene mediato
da due glicoproteine di membrana: l’emagglutinina HA e la neuroaminidasi NA. In altre parole
queste due glicoproteine favoriscono l’inizio del processo infettivo.
L’emagglutinina HA ha affinità per i residui di acido sialico presenti sulla superficie delle cellule
bersaglio, consentendo al virus di legarsi ad esse.
La neuroaminidasi NA è un enzima che catalizza l’idrolisi dei legami glicosidici -(2,3) e -(2,6)
con cui l’acido sialico è legato alle catene poliglicosidiche sulle membrane cellulari. Tale attività
sembra favorire il rilascio del virus da parte della superficie cellulare del tratto respiratorio e il
movimento del virus all’interno dell’ospite, per cui è essenziale per l’intero processo infettivo.
Il funzionamento del biosensore a neuroaminidasi si basa sulla misurazione dell’attività enzimatica
di questo enzima.
È stato messo a punto un biosensore elettrochimico ad H2O2 per determinare l’attività di questo
enzima virale. Infatti, il lattosio che si forma in seguito all’azione idrolitica della NA sul sialil
lattosio, viene scisso dalla -galattosidasi in galattosio e glucosio. Quest’ultimo viene ossidato, in
presenza di O2, dalla glucosioossidasi in acido gluconico e H2O2 secondo la seguente reazione:
GOD
-D-glucosio + O2 acido gluconico + H2O2.
L’H2O2 che si forma viene a sua volta ossidata dall’elettrodo di Pt (anodo) polarizzato a 650 mV
rispetto al riferimento Ag/AgCl. L’intensità di corrente misurata è proporzionale alla
concentrazione di H2O2 liberata dalle reazioni enzimatiche sulla superficie anodica e, quindi, alla
concentrazione del lattosio.
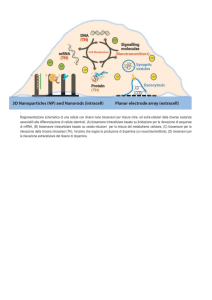
I biosensori non s’inquadrano specificamente in un singolo settore scientifico, ma hanno
applicazioni in svariati campi: tecnologia dell’informazione, biotecnologie e nanotecnologie; questi
settori si possono inter-relazionare e dar luogo a campi di applicazione maggiormente compositi.
Un esempio sono i biosensori con i MEMS (MicroElectroMechanical Systems): si applica la
tecnologia di fabbricazione dei circuiti integrati ai biosensori. Si tratta di lastrine – generalmente di
materiale plastico – in cui sono presenti delle scanalature adatte all’inserimento di diversi elementi.
Queste lastrine sono preparate depositando il materiale polimerico su matrici IDUA (Interdigitated
Ultramicroelectrode Array); si ottengono scanalature in verso opposto a quelle della matrice. Gli
IDUA si ottengono tramite tecniche fotolitografiche e di lift-off su SiO2 + Si3N4 depositati su vetro
o silicio.
Un particolare tipo di biosensori sono i DIPSTICK BIOSENSORS (biosensori a striscette), che
utilizzano dei liposomi per amplificare il segnale. Su una striscetta di materiale di microcellulosa
s’individuano differenti zone:
Su un’estremità si mette il campione, mescolato con antigene, in parte libero e in parte marcato
con un liposoma. Il liposoma contiene una sostanza elettroattiva – l’acido ascorbico – che,
arrivata sull’elettrodo, dà un segnale
C’è una zona di cattura, su cui si trovano gli anticorpi specifici per l’antigene, immobilizzati ed
orientati
Non tutto l’antigene mescolato al campione viene catturato: quello che supera la zona di cattura
arriva su una lastrina di detergente. Si rompe il liposoma e viene liberato l’acido ascorbico
C’è, infine, un elettrodo interdigitato che misura la sostanza elettroattiva. Il liposoma, dunque,
funziona come amplificatore di segnale.
Una disposizione di basi nucleotidiche immobilizzate su un chip può essere utilizzata per la
decodificazione del genoma umano. Tale dispositivo funziona basandosi sull’identificazione
reciproca tra le basi degli acidi nucleici. In particolare, un DNA-chip elettrochimico funziona in
questo modo. Un sensore elettrochimico d’oro è rivestito con streptavidina per realizzare un
accoppiamento con un filamento di DNA o RNA labellato con biotina. Questo filamento costituisce
una sonda di cattura che è complementare alla sequenza da determinare. Un’altra sequenza di DNA
è utilizzata come sonda di misura ed è funzionalizzata con un coniugato anticorpo-enzima. Quando
la sequenza riconosce il tratto complementare del filamento bersaglio, il coniugato anticorpoenzima libera il substrato, che viene rilevato dal sensore elettrochimico.
Ci sono diversi steps nel design e nell’implementazione di un esperimento con microarray di DNA:
Table 1. Steps in the design and implementation of a DNA microarray experiment
1) Probe
(cDNA/oligo
with known
identity)
Small oligos,
cDNAs,
chromosome,
...
(whole
organism on a
chip?)
2) Chip
fabrication
(Putting probes
on the chip)
Photolithography,
pipette, droptouch,
piezoelectric
(ink-jet), electric,
...
3) Target
(fluorecently
labeled
sample)
4) Assay
RNA,
(mRNA==>)
cDNA
Hybridization,
long, short,
ligase, base
addition,
electric, MS,
electrophoresis,
fluocytometry,
PCR-DIRECT,
TaqMan, ...
5) Readout
6) Informatics
Fluorescence,
probeless
(conductance,
MS,
electrophoresis),
electronic, ...
Robotics
control, Image
processing,
DBMS,
WWW,
bioinformatics,
data mining
and
visualization
Attività sperimentale
Abbiamo preparato una soluzione 0.1 M di KCl. Tramite questa soluzione elettrolitica e utilizzando
un elettrodo polarizzato a 1 mV, abbiamo clorurato la parte terminale delle strisce di Ag degli
elettrodi stampati di grafite.