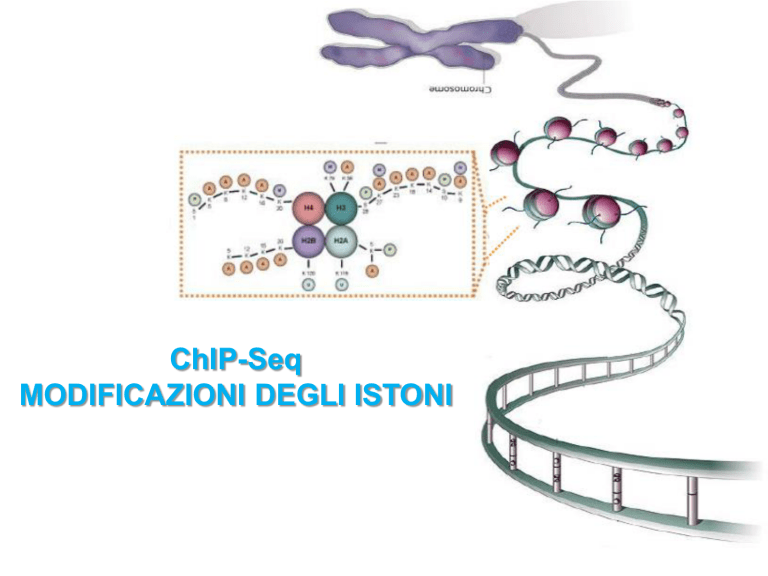
ChIP-Seq
MODIFICAZIONI DEGLI ISTONI
LE MODIFICAZIONI PT DEGLI ISTONI CORRELANO CON SPECIFICI
STATI DELLA CROMATINA NELLE REGIONI REGOLATORIE DEI GENI
Arricchimenti di specifiche modificazioni a livello di:
Promotori (attivi/non attivi)
Regioni a monte di geni trascritti/non trascritti
Regioni interne di geni trascritti/non trascritti
Enhancers, silencers
Esperimento
• Eseguire una ChIP-Seq per diverse
modificazioni istoniche, partendo da quelle
più “classiche”
• Verificare:
– Se ciascuna modifica ha una sua localizzazione
“preferenziale” sul genoma o rispetto ai geni (es.
nel promotore, nella regione trascritta, etc.)
– Se ciascuna modifica è “correlata” in qualche
modo alla trascrizione/espressione dei geni
Esperimento
• ChIP-Seq associata a una particolare
modificazione (es, H3K4me3)
• Domanda: la modificazione è “correlabile” alla
trascrizione dei geni?
• Ovvero, la modificazione “marca” particolari
nucleosomi rispetto all’inizio della trascrizione,
o alla regione trascritta?
• Esempio: potrebbero esserci modificazioni che:
– Marcano l’inizio della trascrizione
– Marcano tutta e solo la regione trascritta
– “Silenziano” particolari loci genici
impedendo la trascrizione
•
•
Barski et.al - Cell 129 823-837, 2007
20 histone lysine and arginine methylations in CD4+ T cells
–
–
–
–
–
–
–
–
•
H3K27
H3K9
H3K36
H3K79
H3R2
H4K20
H4R3
H2BK5
Plus:
– Pol II binding
– H2A.Z (replaces H2A in some nucleosomes)
– insulator-binding protein (CTCF)
Per correlare le modificazioni con la trascrizione hanno analizzato
12,720 geni umani I cui livelli di espressione nei linfociti T CD4+ T
erano noti
I geni sono stati separati in 12 gruppi di 1000 secondo il loro livello
di espressione (alto-medio-basso-silente)
Ciascun gruppo di geni è stato allineato rispetto al TSS, ed è stato
riportato il livello di modificazioni trovate per ciascun gruppo
H3K4me3 si ritrova immediatamente a monte e a valle del TSS dei geni trascritti
H3K4me2 (not me3!) si trova immediatamente prima e dopo del TSS dei geni trascritti,
ma più lontano di H3K4me3
H3K4me1 si trova prima e dopo il TSS ei geni trascritti ma più distante di H3K4me3
e H3K4me2
H3K27me3 l’intera regione genomica dei geni silenti non trascritti
H3K27me1 è associata alle regioni a monte e a valle del TSS dei geni trascritti
H3K36me3 è ritrovato all’interno dei geni trascritti- un po’ più a valle del TSS
H3K9me1 ha un profilo simile a H3K4me3
LE MODIFICAZIONI DEGLII ISTONI DEMARCANO GLI ELEMENTI FUNZIONALI NEI GENOMI
DEI MAMMIFERI
I promotori attivi sono marcati con H3K4me2, H3K4me3, acetilazione (ac) e H2A.Z.
Le regioni trascritte sono arricchite per H3K36me3 e H3K79me2.
I geni repressi possono essere localizzati in grossi domini di H3K9me2 e/o H3K9me3 o
H3K27me3.
Gli Enhancers sono arricchiti per H3K4me1, H3K4me2, H3K27ac .
GENE TARGETING
Ricombinazione omologa in embrionic stem (ES) cells
•SCOPO: Perdita o modifica di funzione
•CARATTERISTICA: inserzione mirata
La procedura per ottenere il gene targeting si basa su 4
fasi distinte
La procedura per ottenere il gene targeting si basa su 4
fasi distinte
1. Preparazione
delle ES cells
pluripotenti e
trasfezione
2. Selezione
delle ES
Preparazione delle ES
pluripotenti dalla blastocisti
3. Iniezione
delle ES
trasfettate in
una nuova
blastocisti
4. Screening
della
progenie
Preparazione del transgene
Gene non attivo
Tk - thymidine
kinase
Sequenze di
ricombinazione
Omologa
Neor – resistenza
alla Neomicina
Elettroporazione
La procedura per ottenere il gene
targeting si basa su 4 fasi distinte
1. Preparazione
delle ES cells
pluripotenti e
trasfezione
2. Selezione
delle ES
3. Iniezione
delle ES
trasfettate in
una nuova
blastocisti
4. Screening
della
progenie
Trattamento con neomicina e
ganciclovir
No integrazione
Gene non attivo
Tk - thymidine
kinase
Sequenze di
ricombinazione
Omologa
Neor – resistenza
alla Neomicina
X
Integrazione sito-specifica (1/1000)
Integrazione random
X
La procedura per ottenere il gene
targeting si basa su 4 fasi distinte
1. Preparazione
delle ES cells
pluripotenti e
trasfezione
2. Selezione
delle ES
3. Iniezione
delle ES
trasfettate in
una nuova
blastocisti
4. Screening
della
progenie
ES inserite in una
blastocisti di topo nero
La procedura per ottenere il gene
targeting si basa su 4 fasi distinte
1. Preparazione
delle ES cells
pluripotenti e
trasfezione
2. Selezione
delle ES
Topo “chimera”
3. Iniezione
delle ES
trasfettate in
una nuova
blastocisti
4. Screening
della
progenie












