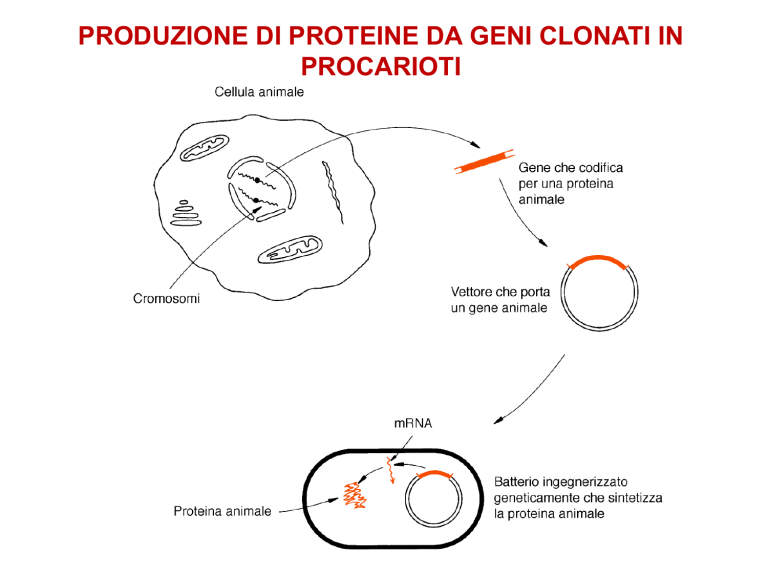
PRODUZIONE DI PROTEINE DA GENI CLONATI IN
PROCARIOTI
fermentatore
PRODUZIONE DI PROTEINE DA GENI CLONATI IN
PROCARIOTI
Vettori a cassetta
PRODUZIONE DI PROTEINE A PARTIRE DA GENI CLONATI
Espressione di geni esogeni in procarioti
Riconosciuto dalla
subunità sigma
RNA pol E. coli
Sequenze promotore in procarioti e eucarioti
riconosciute da RNA polimerasi specifiche
Monitoraggio della
proteina esogena con
possibili effetti dannosi
sul procariote
promotore tac: ibrido tra trp e lac
Ceppo speciale di E. coli lisogeno per
T7 (fago T7 integrato e alterato: gene
per RNA pol T7 a valle del promotore
lac, indotto dall’IPTG )
SVANTAGGI PER LA PRODUZIONE DI PROTEINE
ETEROLOGHE IN BATTERI
- Non vengono rimossi eventuali introni
- Possibile formazione di strutture secondarie della porzione iniziale trascritto (fig)
- Possibile presenza di segnali di terminazione per il batterio (fig)
- Propensione per alcuni codoni (fig)
- Mancanza di modificazioni post-traduzionali (glicosilazioni, fosforilazioni, etc.)
- Limite della lunghezza amminoacidica
- Mancato ripiegamento della struttura terziaria e sovraespressione determinano
i corpi di inclusione
Formazione di strutture secondarie all’inizio del trascritto quali
possibili conseguenze per inserimento sequenza esogena
Vettori a cassetta per la fusione di geni in E. coli
Fusione con parte iniziale di gene batterico
Aumento resa e solubilità proteina eterologa
Vantaggi di una proteina di
fusione in frame:
- presenza seq. nucleotidica di E. coli per
legame ribosoma
- peptide batterico iniziale stabilizzante
- peptide batterico iniziale da utilizzare
come segnale localizzazione cellulare
(esportata nel mezzo di coltura o tra
membrana esterna ed interna
Fusione con la proteina di E. coli Glutatione-S-transferasi (GST) e
purificazione con cromatografia per affinità al substrato (glutatione)
Vettore:
componente N-terminale della GST,
componente C-terminale proteina
da purificare.
Promotore Lac inducibile
libero
Glutatione: tripeptide (cisteina,
glicina, ac. glutammico)
Rimozione della proteina di fusione
es: con bromuro di cianogeno (se alla giunzione c’è metionina),
con trombina (taglia adiacente a residui di arginina).
Non deve essere tagliata all’interno la proteina di interesse!
Proteina priva di struttura terziaria precipita e forma corpi di inclusione.
Aggregati recuperati in forti condizioni denaturanti che non permettono
corretto ripiegamento in vitro della proteina che quindi sarà inattiva!
Screening cloni batterici trasformati con vettori di espressione
Screening cloni batterici trasformati con vettori di espressione
Produzione di proteina
- studi biochimici e funzionali, cristallografici
- produzione anticorpi specifici
SVANTAGGI PER LA PRODUZIONE DI PROTEINE
ETEROLOGHE IN BATTERI
- Non vengono rimossi eventuali introni
- Possibile formazione di strutture secondarie della porzione iniziale del trascritto
- Possibile presenza di segnali di terminazione per il batterio
- Propensione per alcuni codoni
Mancanza di modificazioni post-traduzionali (ponti disolfuro, glicosilazioni,
fosforilazioni, acetilazione, carbossilazione, etc.)
- Limite della lunghezza amminoacidica
- Mancato ripiegamento della struttura terziaria e sovraespressione determinano
i corpi di inclusione
PRODUZIONE INDUSTRIALE DI PROTEINE DA COLTURE
DI CELLULE EUCARIOTICHE (LIEVITI E FUNGHI)
COMPOSTO
Antibiotici
Penicilline
Cefalosporine
Cloramfenicolo, streptomicina
MICRORGANISMO
Penicillium spp.
Cephalosporium spp
Streptomyces spp.
Enzimi
Proteasi, amilasi
Bacillus ssp. (batterio), Aspergillus spp.
Alcool
Aceto
Acetone
S. cerevisiae
S. cerevisiae
Clostridium ssp.
ac. Acetico
BIOTECNOLOGIE: USO DEI PROCESSI BIOLOGICI NELL’INDUSTRIA E
NELLA TECNOLOGIA
VETTORI DI CLONAGGIO PER GLI EUCARIOTI
Lieviti e funghi filamentosi: produzione di proteine eterologhe anche a scopi
biotecnologici con sintesi di prodotti medicinali o alimentari
Saccharomyces cerevisiae
Fungo unicellulare
Ruolo fondamentale per produzione di birra e pane
Vantaggi: conoscenza genetica e biochimica, facilità di coltura, alta resa
Possibile utilizzo per clonaggio del plasmide naturale 2 µm
Svantaggi: iperglicosilazione, difficoltà di secrezione delle proteine prodotte
nel terreno di coltura, propensione per un codone
PROMOTORI UTILIZZATI IN VETTORI DI ESPRESSIONE PER
EUCARIOTI MICROBICI
Galattosio epimerasi
Esempio di glicosilazione dell’asparagina.
L’iperglicosilazione di alcuni funghi può provocare reazione antigenica se la proteina
è iniettata
Saccharomyces cerevisiae
2 µm:
plasmide naturale 6,3 kb,
tra 70-200 copie
risiede nel nucleo,
si replica autonomamente,
segrega sia in meiosi
che in mitosi
REP1 e REP2 (per replicazione)
FLP (proteina che riconosce siti specifici nel DNA del lievito: integrazione
per ricombinazione )
Ori
D = funzione ignota
Saccharomyces cerevisiae
Vettori basati sul plasmide 2 µm: plasmidi episomici di lievito = YEp
Procedura
- clonaggio in seq. di pBR322
-selezione dei ricombinanti in E. coli
(resistenze antibiotici)
- inserimento in lievito e selezione (LEU2)
Saccharomyces cerevisiae
Vettori basati sul plasmide 2 µm: plasmidi episomici di lievito = YEp
Saccharomyces cerevisiae
Vettori basati sul plasmide 2 µm: plasmidi episomici di lievito = YEp
Solo occasionalmente si integra
Ricombinazione omologa tra geni LEU 2
Solo una copia di LEU2
funziona
Saccharomyces cerevisiae
Plasmide integrativo di lievito: YIp = plasmidi batterici (pBR322) con
resistenza antibiotici per selezione in batteri + URA3 (gene di lievito per
la selezione)
Non contiene i geni di 2 µm (REP e ori) e non si può replicare se non si integra nel
DNA dei cromosomi di lievito, stesso meccanismo di YEp
Saccharomyces cerevisiae
Criteri di scelta: efficienza di trasformazione, numero di copie, stabilità
YIp: ideale per stabilità in coltura a lungo termine
Saccharomyces cerevisiae
Caratteristiche del clonaggio in YEp:
- Frequenza di trasformazione molto alta: da 10000 a 100000 cellule trasformate
per microgrammo di DNA plasmidico
- Alto numero di copie: tra 20 e 50 per cellula
- Svantaggi: nel processo di gemmazione spesso copie YEp non segregano
nella cellula figlia
QUINDI ALTA PRODUZIONE PROTEINA DEL GENE CLONATO MA
INSTABILITA’ DELLA TRASFORMAZIONE
Caratteristiche del clonaggio in YIp:
-Frequenza di trasformazione bassa: 1000 cellule trasformate per microgrammo
di DNA plasmidico per bassa efficienza di integrazione cromosomica
- E’ presente in una sola copia per cellula
QUINDI BASSA PRODUZIONE PROTEINA DEL GENE CLONATO MA
GRANDE STABILITA’ DELLA TRASFORMAZIONE PER LUNGHI PERIODI
DI COLTURA












