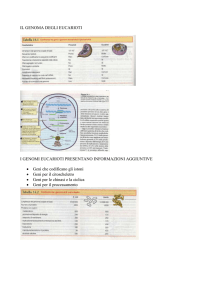
Concetti addizionali: come controllare gli attivatori?
Espressione tessuto-specifica o inducibile di fattori di trascrizione costitutivamente
attivi
Attivazione post-traduzionale di fattori inattivi (es. tramite fosforilazione)
Regolazione della localizzazione cellulare (e.s sequestramento nel citoplasma)
Attivazione mediata da ligando (es. ormoni steroidei)
Attivazione/repressione trascrizionale a lungo raggio:
Le regioni di controllo di un locus (Locus Control Regions – LCR)
Le regioni di attacco alla matrice nucleare (MAR)
Gli isolatori
Attivazione/repressione trascrizionale a lungo raggio:
Regioni cromosomali trascrizionalmente attive (eucromatina)
Regioni cromosomali trascrizionalmente inattive (eterocromatina)
Fenomeno del silenziamento posizionale o eterocromatizzazione
Lo sviluppo di neoplasie comporta sempre anomalie nell’espressione genica:
Fattori di trascrizione come oncogeni.
Esempio: attivazione inserzionale
c) Attivazione tramite traslocazione nel gene della catena pesante delle Ig – t(8;14)
-> LINFOMA DI BURKITT
Fattori trascrizionali come soppressori di tumore.
Esempio: p53
• La stabilizzazione di p53 causata da danno al DNA induce l’espressione di
p21, un inibitore delle cinasi ciclina-dipendenti
• l’ inibizione dell’attivita’ cinasica delle cicline Cdk1, 2, 4 e 6 provoca arresto del
ciclo cellulare
L’importanza delle acetilasi degli istoni: CBP/p300
• L’assenza di CBP nel topo causa lo sviluppo di tumori ematologici
(simile alla sindrome Rubinstein-Taybi, associata all’inattivazione dell’attivita’
enzimatica di CBP)
• Possibile meccanismo: ridotta attivita’ di p53
•Gli oncogeni virali SV40 large T e E1A dell’adenovirus bloccano CBP e
necessitano di questa attivita’ per trasformare le cellule: coinvolgimento di p53?
• Il virus oncogeno HTLV-1 necessita di CBP per attivare la
trascrizione del suo genoma:
Acetilazione aberrante nel cancro:coinvolgimento
sia di HAT che di HDAC
HAT:
Geni codificanti per diverse HAT si trovano traslocati, amplificati,
sovraespressi e/o mutati in diversi tipi di tumori, sia ematologici che epiteliali
Mutazioni missenso o associate a p300 tronca NON FUNZIONALE si trovano
associate a tumori del colon/retto e gastrici (secondo allele deletato)
Gli individui con sindrome di Rubinstein-Taybi portano una mutazione che
inattiva l’attività HAT di CBP -> aumentato rischio di tumori
L’80% dei glioblastomi mostra associazione con perdita di eterozigosi per
p300
perdita di eterozigosi attorno al locus di CBP è associata a carcinomi
epatocellulari.
Traslocazioni di p300 e CBP risultanti in fusioni in frame con diversi geni ->
associate a diversi tumori ematologici
HDAC e cancro
HDAC:
Leucemia acuta promielocitica (APL): PML-RARa, l’oncoproteina generata
da una fusione per traslocazione cromosomica, reprime i geni normalmente
attivati da RARa mediante reclutamento di un co-repressore contenente
attività HDAC
Linfoma non-Hodgkin: il repressore trascrizionale LAZ3 è anormalmente
sovraespresso e causa repressione trascrizionale aberrante (HDAC dipendente)
con conseguente trasformazione tumorale
La leucemia acuta mieloide M2 è associata con t(8;21) che genera una
proteina di fusione AML1/ETO che agisce come un potente repressore
trascrizionale dominante tramite reclutamento di HDAC
Uno squilibrio nell’acetilazione degli istoni può quindi causare
alterazioni della struttura della cromatina e regolazione aberrante di
geni coinvolti nel controllo del ciclo cellulare, del differenziamento e/o
dell’apoptosi
Inibitori delle HDAC: una nuova terapia anti-cancro
Diverse sostanze che causano differenziamento, arresto della crescita
e/o apoptosi di cellule trasformate -> inibitori di HDAC
(es. DMSO, Tricostatina A (TSA), butirrato)
Questi inibitori causano arresto del ciclo cellulare in G1 o G2,
differenziamento e/o apoptosi in vitro, documentati in tutti i tipi
cellulari trasformati incluse linee cellulari derivate sia da tumori
ematologici (leucemie, linfomi, mielomi) che epiteliali (tumore dei
polmoni, delle ovaie, del seno, della prostata)
Le concentrazioni biologicamente attive correlano con quelle richieste
per causare accumulazione di istoni acetilati
SI IGNORA quali siano gli HDAC la cui attività deve essere bloccata
Gli inibitori causano aumento dell’ acetilazione sugli istoni (H2A,
H2B, H3 e H4) anche in cellule normali, ma l’attività di soppressione
della crescita sembra essere limitata alle cellule trasformate.
Geni bersaglio:
CDKN1A (codificante per l’inibitore dell’attività cinasica ciclinadipendente p21/WAF1) (tramite un sito di SP1 sul promotore)
CDKN2A (p16)
Ciclina E
Altro meccanismo per reprimere la trascrizione: la
metilazione del DNA
La metilazione nei vertebrati avviene esclusivamente sui residui di C
nel dinucleotide CpG
Le sequenze CpG sono sotto-rappresentate nel genoma (probabilmente
per la tendenza della 5-metilcitosina a venire deaminata e mutata in T)
-> ma abbondanti nelle regioni promotrici dei geni
In cellule non embrionali, 80% dei CpG sono metilati, ad eccezione
delle isole CpG dei promotori
Le DNA metil-transferasi (Dnmt) hanno particolare affinità per le
sequenze semi-metilate: tendono quindi a metilare il nuovo filamento
che si è formato su uno stampo metilato-> mantenimento del pattern di
metilazione
La metilazione è regolata durante lo sviluppo:
Il DNA viene ipermetilato nei gameti
Demetilato durante le prime divisioni cellulari
Successivamente rimetilato in modo specifico per i diversi tipi cellulari
(modificazione epigenetica)
Funzioni della metilazione:
Difesa contro i trasposoni
Regolazione genica
Metilazione e regolazione genica:
Dnmt metilano il DNA
Il DNA metilato è legato da proteine che legano il metile
(MeCP2, MBD1-4)
Queste a loro volta sono in grado di reclutare diversi HDAC ->
repressione della trascrizione
Il pattern di metilazione dei geni è alterato nelle cellule tumorali, e
metilazione alle isole CpG di certi geni è associata al loro
silenziamento specifico (es. PML-RAR lega il promotore del gene
RARb e causa il reclutamento di DNMT -> metilazione -> MBD ->
HDAC)
La metilazione è il meccanismo su cui si basa l’imprinting
Modificazioni epigenetiche, trascrizione e ambiente
In quanto reversibili, i cambiamenti epigenetici possono essere
modificati dall’ambiente
Sia ipo- che iper-metilazione sono stati associati all’invecchiamento
(perdita generica di metilazione, ipermetilazione di geni specifici:
silenziamento di soppressori tumorali?)
Vitamine quali l’acido folico, che sono importanti per l’attività degli
enzimi che forniscono gruppi metilici, hanno effetti notevoli
sull’incidenza di tumori quali in cancro al colon
Dieta scarsa in folati -> instabilità genomica e ipometilazione
Dieta scarsa di gruppi metilici -> cancro del fegato, e
ipometilazione+sovraespressione di oncogeni (c-ras, c-myc, c-fos)
Cooperatività tra inibitori di HDAC e di DNMT
5-aza-2’-desossicitidina (inibitore di DNMT1) -> ipometilazione e
riattivazione di soppressori di tumori silenziati
Può cooperare con inibitori dele HDAC quali TSA
Con potenziale riduzione delle dosi richieste e della tossicità, e minori
possibilità di insorgenza di meccanismi di resistenza