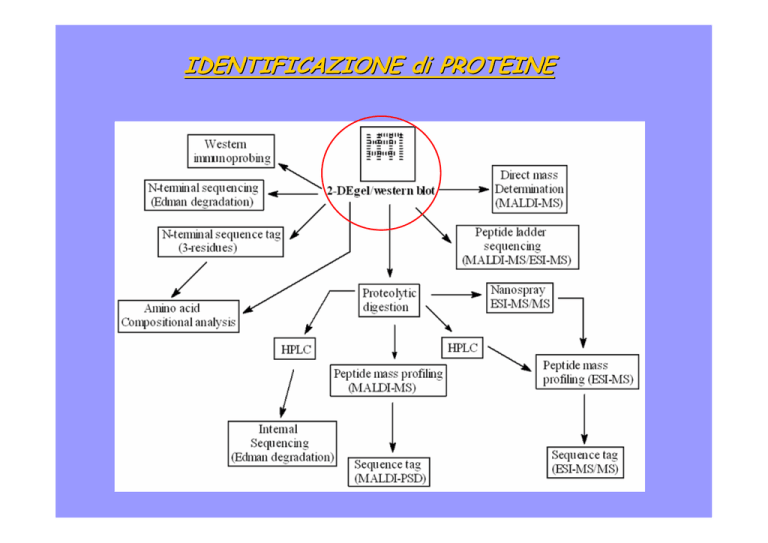
IDENTIFICAZIONE di PROTEINE
APPROCCIO GENERALE ALLO STUDIO PROTEOMICO
1a Tappa
9 Preparazione del campione
9 Produzione di gel 2D
2a Tappa
9 Analisi delle immagini
Visualizzazione
–
Manipolazione
dell’immagine - Identificazione e
quantificazione
degli
spot
Comparazione delle immagini - Analisi
dei dati - Collegamento a banche dati
3a Tappa
9
9
9
9
9
9
9
Estrazione degli spot di interesse
Eluizione della proteina (raramente)
Digestione proteolitica in gel della proteina
Estrazione dei peptidi
Analisi MS
Comparazione dei risultati con databases
Identificazione della proteina
4a Tappa
Studi sulla struttura e sulle modificazioni della proteina
5a Tappa
Analisi della funzione ………..
Identificazione di una proteina con
Peptide Mass Fingerprinting (PMF)
Siti di taglio della tripsina: Arg and Lys
Proteina
Digestione
K
K
R
R
.....
m3
Peptide mass
fingerprint con
MALDI-TOF MS
m1
m2
m4
m/z Da
Ricerca in Database
Identificazione
della proteina
m1
m2
m3
......
mr exper.
mr theor.
902.546
971.477
1234.690
........
902.535 Da
971.447 Da
1234.690 Da
...........
Diff.
Residues
0.012
0.030
0.000
........
124-131
188-195
136-146
........
Peptide sequence from
the database
GLFIIDPK
EYFEAANK
HITINDLPVGR
DIGESTIONE ENZIMATICA
Enzimi proteolitici più usati:
ENZIMA
Tripsina
Chimotripsina
Glu-C (SP V8 proteasi)*
SPECIFICITA’ DI
TAGLIO
/K-, /R-, \P
/W-, /Y-, /F-, \P
/E-, /D-, \P
Lys-C
/K-, \P
Asp-N
/D-
* Il legame in cui è coinvolto l’Asp viene tagliato solo in tampone fosfato
Può essere fatta anche una proteolisi chimica.
Es: BROMURO DI CIANOGENO (BrCN). Sito di taglio: /M-
Per aumentare l’efficienza del taglio proteolitico è importante
ridurre ed alchilare i ponti disolfuro
s
s
R
s s
riduzione
s
s
s
s
s
s
s
s
R
R
R
alchilazione
s
R
R
s
s
Digestione
enzimatica
R
R
s
DIGESTIONE “IN GEL”
9 ISOLARE LO/GLI SPOT
9 ELIMINARE COLORANTE, SDS E ALTRI CONTAMINANTI
presenti nel gel, che potrebbero interferire con il processo di
digestione:
• Lavaggio in acetonitrile 50%
• Essiccamento
• Reidratazione ed equilibratura nel tampone di digestione
9 RECUPERARE I PEPTIDI:
• Diffusione passiva
• Estrazione in ambiente acido (TFA 1%) con ultrasuoni
n.b. Difficile il recupero di peptidi con p.m.>5 kDa!
ANALISI MEDIANTE SPETTROMETRIA DI MASSA
Spettro di massa di una miscela peptidica ottenuta
per digestione proteolitica di una proteina:
ANALISI DEI PESI MOLECOLARI DEI PEPTIDI OTTENUTI
PER DIGESTIONE SPECIFICA DI UNA PROTEINA
L’identificazione di una proteina dipende dai seguenti parametri:
9Accuratezza nella determinazione della massa dei frammenti
9Numero delle masse sottomesse per l’interrogazione del database
9Distribuzione delle masse
9Numero delle masse che sono in accordo fra quelle sperimentali e
quelle teoriche per la proteina del database
9Dimensione del database di sequenze proteiche
9Numero delle modificazioni della proteina considerate
SPETTROMETRIA DI MASSA
Tecnica utilizzata per la determinazione del peso molecolare delle molecole
generazione di ioni in fase
gassosa
separazione degli ioni in
funzione del rapporto m/z
rivelazione degli ioni separati
SPETTRO DI MASSA : mappa dell’ abbondanza relativa degli ioni
prodotti in funzione del rapporto massa/carica (m/z)
Requisiti essenziali per ottenere uno spettro di massa:
¾ Produrre ioni in fase gassosa
¾ Accelerarli ad una velocità specifica
¾ Proiettarli in un adatto analizzatore di massa
¾ Rivelare le molecole cariche che giungono al detector
Tecniche di ionizzazione
Analizzatore
• Impatto Elettronico (EI)
• Settore
• Ionizzazione Chimica (CI)
• Quadrupolo
• Bombardamento con atomi
• Trappola ionica
“veloci” (FAB)
• Tempo di volo (TOF)
• Ionizzazione/Desorbimento di
• “Trasformata di Fourier”
Campo (FI/FD)
(FTMS)
• Ionizzazione per Desorbimento
Laser Assistito da Matrice
(MALDI)
• Ionizzazione Electrospray (ESI)
Metodi di ionizzazione comunemente
usati per l’analisi di proteine:
EI: Electron impact Ionisation
È la modalità più classica,
tuttora molto utilizzata.
È una ionizzazione ‘hard’.
Il campione deve essere allo
stato gassoso.
Adatto per composti piccoli
(< 800 Da), volatili,
termicamente stabili.
Interfaccia con GC, anche
HPLC.
Sorgente di ionizzazione ad
Impatto Elettronico
Filamento di rutenio
o tungsteno
ricoperto di ossido
di torio
Molecole neutre
ed in movimento
casuale
ee-
Entrata
campione
ee-
+
+
+ + +
eee-
Analizzatore
ee-
Lenti
focalizzatrici
Repulsore
+20 eV
+ + +
Trappola +70 eV
-4kV
La ionizzazione generalmente avviene:
per espulsione di un elettrone
(generazione di ioni positivi)
e((
+
))
ione molecolare
o per cattura di un elettrone
(generazione di ioni negativi)
e((
))
ione molecolare
Se la quantità di energia assorbita dalla
molecola è elevata si avrà una decomposizione
della molecola in frammenti di massa inferiore
e( (( (
+
+
) ) ))
ione molecolare
+
+
Ioni
frammenti
+
IONIZZAZIONE CHIMICA
9 Si basa essenzialmente su una sorgente EI.
9 La sorgente viene riempita con metano (CH4) o
ammoniaca (NH3).
9 Si formano le specie CH5+ e NH4+, potenti
donatori di protoni allo stato di vapore.
9 Ionizzazione dell’analita per protonazione (dopo
interruzione del fascio di elettroni).
9 Utilizzato nello studio di farmaci e dei loro
metaboliti secondari.
Lo
ione
molecolare
del
metano,
generato
per
impatto
elettronico, puo' reagire con l'eccesso di metano:
.
CH + + 2e-
CH4 + eCH4 + CH4
4
.+
CH3 + CH5+
Il catione CH5+ è una specie instabile e si comporta come un
acido forte. Può quindi protonare con una reazione acidobase praticamente qualsiasi molecola organica.
Questa tecnica di ionizzazione genera uno ione molecolare
MH+ con un bassissimo eccesso di energia vibrazionale, e le
reazioni di frammentazione sono quindi poco importanti.
FAB
“Fast Atom Bombardment”
Il campione viene prima dissolto
in una matrice liquida (es.
glicerolo).
La superficie liquida viene
bombardata da un fascio di
atomi ad alta energia cinetica
(xenon o argon).
Le molecole di campione vengono
ionizzate, per protonazione o
deprotonazione, ed accelerate
nell’analizzatore in fase gassosa.
Adatto ad es. per peptidi, piccole proteine, altri biopolimeri
(fino a p.m. ≈ 5000).
Ionizzazione “soft’”.
Quantita’ minima: ≈ 20 picomoli
Solitamente abbinato ad ANALIZZATORI A SETTORE
Ionizzazione FAB:
Si adatta molto bene all’analisi della maggior parte
delle sostanze biologiche (peptidi, piccole proteine,
altri biopolimeri).
Le molecole vengono
mescolate ad una matrice.
introdotte
in
soluzione
Le molecole vengono bombardate da atomi che
possiedono una elevata energia cinetica.
Si possono ottenere sia ioni positivi che ioni negativi.
L’introduzione di questa tecnica nei primi anni ‘80 ha
rivoluzionato la spettrometria di massa, aprendola alla
biologia e alla medicina.












