”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
CORSO DIPi GENETICA
o
n
i
o rb
t
r
U
e
i
b
d
o
LA
GENETICA
DI
R tà
i
s
BATTERI
E VIRUS
r
e
v
i
n
U
La coltivazione dei batteri
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
Una colonia è formata da
r
e
circa 10 cellule.
v
i
n
Clone = discendenti di
U
un’unica cellula.
7
Scambio di materiale genetico
”
o
i
B
l
i
t rlo
n
Meccanismi mediante i qualieavviene a
scambio
g
C
r
“
di materiale genetico
tra
batteri:
e o
i
P
n
i
¸ trasformazione
o rb
t
r
U
e
¸coniugazione
i
b
d
o
R ¸tàsesduzione
i
s ¸trasduzione
r
e
v
i
n
U
La trasformazione
”
o
La trasformazione è il trasferimento di materale genetico da un
batterio all’altro mediato da frammenti di DNA extracellulare.
i
B
l
i
t rlo
n
pneumoniae)
è
La trasformazione (in Streptococcus
e
a
g Griffith
C
osservata per la prima volta da Frederick
nel 1928.
r
“
e o
i
n
Nel 1944 Avery, MacLeod P
e
i
o
b
t
McCarty dimostrarono rche il r
U
e
principio trasformante
era
il
i
b
d
o
DNA.
R tà
i
Nella trasformazione s
frammenti
r
e
isolati di DNA vengono
assorbiti
v
i
dall’esterno
all’interno
della
n
U
cellula.
stata
La mappa per
trasformazione
”
o
i
B
l
i
t rlo
n
e Ca
Con la
g
r
“
e
trasformazione
è
i
o
P
possibile mappare iin
o
t
geni: r
quanto piùrb
U
dueemarcatori
sono
i
b
d
o
vicini, tanto
più
R probabile
à
è la loro
t
i
s
cotrasformazione.
r
e
v
i
n
U
p ed o non stanno mai insieme ->
sono i più distanti -> q è al centro!
La trasformazione a livello
molecolare
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
La scoperta
della
coniugazione - 1
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i no
Nel 1946 Joshua Lederberg e P
i
o
Edward Tatum dimostrarono
b
t
r
r
che due ceppi di eE.coli
U
b bio di
auxotrofi (E. coli A o
met
à
thr leu thi ed E.R
coli B met
t
i
bio thr leu thi ), posti
a
s
r
contatto, possono e
scambiarsi
v
i
materiale genetico
e creare
n
U
dei batteri prototrofi.
-
+
+
+
-
+
+
-
-
Colonie prototrofe: 1x10-7
”
o
La scoperta della coniugazione - 2
i
B
l
i
Alla fine degli anni
t rlo
n
‘40 Bernard Davis
e Ca
g
scopre che se i
r
“
e
due ceppi sono Pi
o
n
i
separati da un o
b
t
r
r
filtro attraverso
U
e
i
cui possono
b
d
o
passare
sostanze
R tà
i
(ma non batteri) non si hasla
r il
formazione di prototrofi:
e
vdue ceppi è
i
contatto fisico tra
i
n
indispensabile! U
”
o
Il fattore di fertilità (F)
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Nel 1953 William Hayes scoprì che il trasferimento
genico avviene in una sola direzione: da un donatore
(maschio) ad un ricevente (femmina). Hayes trovò
un donatore “sterile” (cioè incapace di trasferire
l’informazione) che si era trasformato in un
ricevente, pertanto ipotizzò che la capacità di
fungere da donatore fosse una condizione ereditaria
determinata da una fattore di fertilità F. I ceppi che portano F sono
donatori (F+), quelli senza F sono riceventi (F-). Inoltre osservò che il
trasferimente genico era un evento raro, ma il fattore F veniva
trasferito facilmente. Successivamente fu dimostrato che il fattore
F è un plasmide, cioè un anello di DNA indipendente dal cromosoma
batterico e non facente parte del genoma batterico, che si replica
nel citoplasma batterico in maniera autonoma.
La scoperta della
coniugazione - 3
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
I ceppi Hfr
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
Quando si mescolano
cellule
F+ con cellule F- il fattore F si
d
o
R
à
integra nel cromosoma tbatterico
con una bassa frequenza. Ciò
i
s di geni batterici ma con bassa
determina trasferimento
r
e
frequenza. Nell’incrocio
Hfr x F- invece, dal momento che tutti i
v
i hanno F integrato, la frequenza di ricombinanti
batteri donatori
n
U
è alta.
Luca Cavalli Sforza scoprì un nuovo ceppo
derivato da un F+ chiamato high frequency of
recombination (Hfr). L’incrocio Hfr x F- dava
1000 volte più ricombinanti dell’incrocio F+ x
F-, ma nessuna cellula F- diventava F+. Si
scoprì poi che il ceppo Hfr si forma in seguito
all’integrazione di F nel cromosoma batterico.
L’integrazione
”
o
di
F
i
B
il
t rlo
n
e Ca tra due anelli di
La ricombinazione
g
r
DNA,
in “seguito ad un singolo
e
i
o
over,
porta
alla
P crossing
n
i
o
formazione
di un anello unico che
b
t
r
r
è la somma dei due precedenti. In
U
e
i
b
questo modo un ceppo F+ (con F
d
o
R tà
plasmide libero) può trasformarsi
i
in un ceppo Hfr. Una volta
s
r
e
inserito, il plasmide F mantiene la
v
i
sua capacità di riconoscere un
n
U
batterio F- e, in questo caso,
tenta di iniziare la coniugazione.
Il trasferimento
di Hfr - 1
i
B
l
i
t rlo
Il trasferimento di F integrato (Hfr)n
e Ca
comincia sempre a partire dall’origineg di
r
“
e
replicazione O. Ma quando è integrato
O
i
o
P
n
non si trova all’estremità di F: il ricevente,
i
o
b
t
per poter diventare F+, dovrebbe
ricevere
r
r
U
tutto
il
cromosoma ebatterico
del
i
b
donatore. Poiché il piloosessualedè instabile,
R
à
anche a causa del motoitbrowniano del
s
liquido di coltura, il trasferimento
non è
r
e
(quasi) mai completo:
il ricevente resta Fv
i
n
ma ottiene una parte
di DNA batterico dal
U
donatore che può eventualmente essere
integrata nel cromosoma.
”
o
Il trasferimento di Hfr - 2
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
A questo punto, il ricevente non ha ricevuto un DNA circolare, ma
lineare! Questo potrà eventualmente essere integrato nel genoma
del ricevente per ricombinazione sfruttando l’omologia del DNA
batterico, e solo in seguito ad un numero pari di crossing over.
La coniugazione interrotta
Elie Wollman e François Jacob, 1957
i
B
l
i
o
t
l
n
Si può sfruttare la labilità del
r
e
a
g “C
pilo e l’inserzione di F per
r
e o
mappare i geni di un cromosoma
i
P in
batterico.
Infatti,
leo
b
t
r
integrazioni di F sono casuali
r
U
e
sia come punto di inserzione,
i
b
d
o
sia come orientamento
del
R tà
plasmide, quindi ogni ceppo
i
s geni
r
batterico Hfr trasferirà
e
v
nel ricevente con uni ordine che
n
riflette la disposizione
U dei geni
lungo il cromosoma.
”
o
La procedura
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
I due tipi di cellule vengono
mescolate in gran quantità in
un terreno liquido a 37°C. A
vari tempi vengono prelevati
dei campioni della coltura,
vengono
agitati
(per
interrompere artificialmente
la coniugazione) e piastrati su
terreni selettivi (nell’esempio
illustrato a lato, contenente
streptomicina) per uccidere le
cellule Hfr. Si ottiene in
questo modo una mappa a
tempo
del
cromosoma
batterico donatore.
”
o
thr+leu+aziRtonR
Esempio: donatore HfrH
lac+gal+strS
ricevente: F- thr leu aziStonS lac gal strR
Le mappe a tempo o”
B
li
i lo
t
n
r
e
a
¸ ogni allele del donatore appariva nel g
ricevente
F- dopo un intervallo
C
r
“
e
di tempo ben preciso dall’inizio della
coniugazione;
i
P ino
¸ gli alleli del donatore sito
presentavano
sempre in una specifica
b
r
r
sequenza;
U
e
i
b
o à dpiù tardi comparivano in un numero
¸ i marcatori cheRentravano
t
minore di cellule.
i
s
r
Da queste osservazioni
dedussero che il trasferimento avviene a
e
v
i ben preciso sul cromosoma del donatore chiamato
partire da un punto
n
U secondo una modalità lineare.
origine O e prosegue
Wollman e Jacob arrivarono a queste conclusioni perché:
Costruzione della
mappa ”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
Solo dopo circa 2 ore i riceventi diventano
Come unità di misura si usa il tempo in
minuti impiegato dagli alleli del donatore
ad entrare nel ricevente.
F+ Ÿ il fattore F entra per ultimo.
L’integrazione di F
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R l’integrazione
à
Fu Campbell che scoprì che t
di F avviene tramite un singolo
i
sanelli di DNA, portando alla loro fusione e
crossing over tra i due
r
e
creando un unico anello
che è la somma dei due componenti. Poiché
v
i
n
esistono nel cromosoma
batterico vari siti con sequenza omologa alla
U
regione di appaiamento di F, si possono ottenere tanti ceppi Hfr, ognuno
dei quali trasferisce i geni in un ordine diverso.
Il cromosoma batterico
”
è circolare i
o
B
l
i
t rCampbell
Usando vari ceppi Hfr ed ottenendo mappe n
diverse,
lo ipotizzò
e Ca
che il cromosoma batterico fosse circolare.
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
Il saggio a tre punti nei batteri
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
La sesduzione,
ovvero F’
i
B
l
i
t rlo
Nel 1959 E. Adelber scoprì che in
n
e Ca
un ceppo Hfr il fattore F può
g
r
“
excidersi e generare un ceppo F+.ie
o
P
n
Se però excide in maniera errata,
i
o
b
t
può incorporare il gene batterico
r
r
U
e
accanto al quale era inserito.
In
i
b
d
o
questo caso prende il nome
di F’.
Il
R
à
plasmide F’ può coniugare
ed
t
i
s
inserire il gene che ha rincorporato
e
in
un
batterio
ricevente
v
i
(sesduzione) chen diventa F+ e
U
produce un diploide parziale
stabile, o merozigote.
”
o
La trasduzione
”
o
i
B
l
i
t rlo
n
e Ca
g
I fagi che
effettuano
la trasduzione
r
“
e veicolano
i
generalizzata
qualsiasi porzione
P ino
del ogenoma b
dell’ospite, mentre i fagi che
t
r
r
effettuano
la trasduzione specializzata
U
e
i
b
trasferiscono
solo porzioni specifiche.
d
o
R La
à
trasduzione generalizzata fu scoperta nel
t
i
s 1951 da Joshua Lederberg e Norton Zinder
r
e
che ottennero prototrofi anche con
v
i
n
l’esperimento del “tubo a U”. Il meccanismo
U
venne poi chiarito da Ikeda e Tomizawa con
È il trasferimento dei geni batterici mediato
da batteriofagi.
il fago P1 nel 1965.
Il ciclo vitale di un fago litico
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
La trasduzione generalizzata
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
NB: nella
testa del
fago non
restano
geni fagici!
La mappa per trasduzione
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb donatore leu+thr+azi x
t
r
U
ricevente leu-thr-azi
e
i
b
d
o
Dalla prima parte (leu ):
R tà
thr
leu azi
i
s
r
oppure
e
thr
azi leu
v
i
n
U parte si deduce che è la prima, la mappa giusta!
Ma dalla seconda
Con la trasduzione generalizzata si possono stabilire relazioni
di associazione tra i geni.
La cotrasduzione è il trasferimento di due geni batterici
(molto vicini) ad opera dello stesso fago. Ad esempio:
R
S
+
La frequenza di cotrasduzione
i
B
l
i
t rlo
n
e dei
numero
trasduttanti
a
frequenza di
g
C
r
trasduttanti
per entrambi
i marcatori
“
cotrasduzione
=
e
x 100
i
o
P numero
n
di trasduttanti totali
i
o rb
t
r
U
e
selezionando
per
a+
(a+b+)
i
b
x 100
freq. di cotrasduzione
=
d
o
R tà (a+b-)+(a+b+)
i
s
r
e
selezionando
per b+
(a+b+)
v
i
x 100%
freq. di cotrasduzione =
n
(a-b+)+(a+b+)
U
donatore a+b+ x
a+ba-b+
a+b+
”
o
ricevente a-b-
La trasduzione specializzata
”
o
i
B
l
i
t rlo
n
Alcuni batteriofagi, una volta entrati e
nell’ospite, possono seguire
a
g
C
due strade alternative. Come descritto,
possono
procedere con il
r
“
e entrare
i
classico ciclo litico. Oppure possono
in un ciclo detto
o
P
n
i
lisogenico (o lisogeno); in questo
caso
si
parla di fagi temperati o
o
b
t sonor dei fagi quiescenti che
profagi. I fagi temperati
r
U
e
i
sopravvivono nell’ospite
attraverso
le generazioni cellulari senza
b
d
o
lisarlo, e sfruttano
la
replicazione
del DNA batterico per la loro
R tà
i
stessa replicazione e sopravvivenza.
La quiescenza avviene grazie
s
r
ad un sistema di econtrollo
genetico che impedisce al fago di
v litico finché non si verificano determinate
innescare il ciclo
i
n
condizioni. In
U questo modo il fago può sopravvivere senza nuocere
all’ospite, in teoria indefinitamente.
Il ciclo lisogenico
del profago l
i
B
l
i
t rlo
n
e Ca
Il profago l si può integrare con un
g
r
“
di
singolo crossing over a livello del “sito
e
i
o
P
attacco di lambda” tra i geni gal e bio. in
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Meccanismo della
trasduzione specializzata
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
NB: nella testa del
fago restano
ANCHE i geni fagici!
Riassumendo…
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Gli eterozigoti
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
Un organismo eterozigote
doppio
rispetto
a
due
mutazioni può essere cis o
trans eterozigote rispetto ad
esse. Se le due mutazioni sono
sullo stesso cromosoma, allora
si dice che esse sono in cis.
Altrimenti sono in trans. In
generale, tutti gli elementi
genetici che si trovano sulla
stessa molecola di DNA sono
in cis tra loro.
”
o
Le
mutazioni
in cis
Le due
mutazioni
possono
essere
intrageniche
o
intergeniche!
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Le mutazioni in
trans
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
Le mutazioni in
U
e
i
b
trans possono
d
o
R tà
NON dare
i
fenotipo
s
r
selvatico!
e
v
i
n
U
”
o
I fenotipi dei fagi
”
o
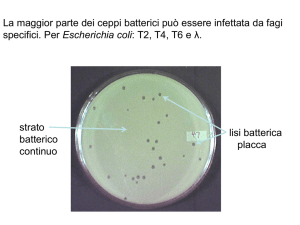
I fenotipi fagici possono essere riconosciuti per la forma e/o le
dimensioni delle placche di lisi, e per la specificità d’ospite (ceppo
batterico che un fago è in grado di lisare).
i
B
l
i
t rlo
n
e Ca
g
Marcatori del fago T2:
r
“
e
i
o
h+ lisa il ceppo B di E. coli ma non
P il ceppoB/2
n
i
o
b
t
h lisa sia il ceppo B che il B/2
r
r
U
e
i
b
r+ forma placche piccole
con
margini
indistinti
d
o
R
à
t
r forma placche grandi con
margini netti
i
s
r
su uno strato misto di cellule B e B/2
Quando i fagi h+ crescono
e
v
formano placche itorbide
perché lisano solo i batteri B, mentre i
n
batteri B/2 U
crescono nelle placche provocandone la torbidità. I
fagi h formano invece placche chiare.
I fenotipi dei fagi
Esistono alleli mutanti del gene
r detti rII.
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
L’incrocio
tra fagi
i
B
l
i
t rlo
n
e Ca
Per incrociare tra loro
g
r
dei fagi (organismi
“
e
i
aploidi,
riproduzione
P ino
o rb
non
sessuata)
di
t
genotipo diverso si può er
U
i
b
coinfettare un batterio
d
o
R tà
ospite usando opportune concentrazioni dei si
r
tre organismi. I DNA
e
v
i
fagici
nel
batterio
n
U
possono eventualmente
ricombinare tra loro.
”
o
Mappatura dei geni fagici
”
o
i
B
l
i
t rlo
n
Frequenza e
di
a
g
C
r
ricombinazione
“
e o
i
traPh ed rin
o rb
t
r
U
e
i
b
d
o
R tà
i
s
placcher (h+r+) + (h-r- )
x 100
e
v
i placche totali
n
U
La complementazione nei fagi
”
o
Esistono tanti ceppi mutanti di fagi di tipo rII; per vedere se questi
appartengono tutti allo stesso locus genetico si esegue un test di
complementazione.
i
B
l
i
t rlo
n
e conCmargini
a netti; sono in
g
I fagi mutanti rII producono placche grandi
r
“
e
grado di lisare il ceppo B di E. coli ma
non
il
ceppo
K(l).
i
o
P in
oplaccherbpiccole con margini irregolari;
I fagi selvatici rII+ producono
t
sono in grado di lisare siaeilrceppo BU
di E. coli che il ceppo K(l).
i
b
o àd
Per fare il test diRcomplementazione
tra due fagi mutanti rII (es.
t
rII e rII ) si fa una doppia
infezione (o infezione mista) su K(l) e si
i
soppure no.
r
verifica se avviene la lisi
e
v
i mutanti vengono piastrati ad alta molteplicità di
I fagi dei due ceppi
n
Uad un concentrazione elevata in modo che i batteri
infezione, cioè
1
2
vengano infettati allo stesso tempo da entrambi i tipi fagici mutanti.
I risultati di una doppia
infezione o”
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
Se si ha la lisi delle cellule K(l) vuol
dire che le mutazioni sono a carico di
geni diversi e pertanto complementano.
Se non si osserva lisi le mutazioni sono
a carico dello stesso gene e di
conseguenza non complementano.
Nella complementazione i genotipi della
progenie fagica rimangono mutanti come
quelli dei genitori. Avviene solo un
mescolamento
di
prodotti
genici.
Viceversa,
nella
ricombinazione
i
genotipi della progenie sono diversi da
quelli dei genitori.
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Analisi genetica della
progenie fagica o”
i
B
l
i
La progenie fagica si piastra
su
o
t
l
n
r
e
a
g “C
r
E.coli B
e E.colio K(l)
i
P in
o rb
t
Complementazione
r
U
e
i
b
d
o
R tà
i
s
Ricombinazione
r
e
v
i
n
U
La frequenza di ricombinazione è di solito molto
tutti i fagi
formano placche
(titolo elevato)
non si osserva
formazione di
placche
tutti i fagi
formano placche
(titolo molto
basso)
formano placche
la metà dei fagi
(i selvatici) che
lisano B. I doppi
mutanti non
formano placche
bassa e non interferisce con la complementazione.
Il cistrone
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Mediante il test di
trans
cis
complementazione
+
m1
m1
m2
è possibile definire
+
m2
+
+
il gene come unità
complementazione
di funzione. Un
complementazione
gene o cistrone è la
cis
regione
genetica
trans
m1 +
+
+
m1
m2
all’interno
della
+
+
+ m2
+
quale
non
c’è
+
complementazione
assenza di
complementazione
tra mutazioni. Il
complementazione
cistrone è l’unità
di funzione.
Il cistrone prende il nome dal test di complementazione che si
chiama test cis-trans.
La struttura fine del geneo” - 1
i
B lavorando
l
i
Negli anni ‘40 Edward
B.
Lewis,
t rlo
n
del acromosoma 2 di
sul locus Star
e
g controlla
C
Drosophila r(che
le dimensioni
“
e trovòoche su 57.000 individui
i
dell’occhio)
P
n S (Star, dominante) per
i
natiodall’incrocio
b
t
r
r
ast
(asteroid, recessivo, anch’esso con
U
e
occhioi ridotto) solo 16 avevano occhi
b
d
o
normali
R tà (quindi con genotipo +/+). La
i
spiegazione più semplice era che fosse
s
r avvenuta ricombinazione tra mutazioni in
e
v siti diversi dello stesso gene. S ed ast
i
n
furono chiamati pseudoalleli.
U
La struttura fine del gene - 2
”
o
i
B
l
i
o
t lozenge
l
Nel 1940 Oliver lavorava sul gene
(lz) sul
n
r
e lz/lz
a
hanno occhi
cromosoma X di Drosophila (gli individui
g
C
r
“
con superficie lucida e liscia).
Isolò
molti
alleli lz che
e
i
o
lz lz ) ed in eterozigosi
mappavano allo stesso locusP(lz lz in
oReincrociando
b
t
davano fenotipo mutante.
eterozigoti per due
r
r
U
e
diversi alleli lz si ottenevano
selvatici, a bassa frequenza.
i
b
d
o
Anche in questo
caso
si suppose che avvenisse
R tà del gene lozenge e gli alleli lz
ricombinazione all’interno
i
s
furono chiamati rpseudoalleli.
Usando la frequenza dei
e
ricombinanti selvatici
si poteva costruire una mappa delle
v
i
mutazioni lznall’interno del locus lozenge.
U
BS
K
l
g
Seymour Benzer
”
o
Benzer negli anni ‘50 utilizzò un sistema molto
sensibile
per
mettere
in
evidenza
la
ricombinazione intragenica nel fago T4 e costruì
una mappa genetica dettagliata di siti all’interno
del gene rII. La sensibilità del sistema adottato
era data dal fatto che i fagi producono progenie
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
omolto numerosa,
b
t
per cui era possibile
r
rII
rII
r
U
e
mettere
in
evidenza
eventi
di
i
b
assenza di
placche
d
o
ricombinazione
estremamente rari come
lisi
piccole
R
à
margini
t
quelli che si verificano all’interno di un
i
irregolaris
singolo gene. Egli isolò circa 3.000
r
placche
placche
e
mutanti rII e li saggiò a coppie, tramite
v
grandi
piccole
i
n
margini
margini
doppie infezioni, per vedere se erano
U
netti
irregolari
allelici o no.
+
K(l)
B
rIIA ed rIIB
”
o
i
B
l
i
t rlo
n
e Ca tra loro ma
g
I mutanti del cistrone A non complementavano
r
“
e
complementavano con tutti i mutanti
del
cistrone
B. I mutanti del
i
o
P
n
cistrone B non complementavano tra loro ima
complementavano con tutti
o
b
i mutanti del cistrone A. r
Iltnumero rdei mutanti assegnati a ciascun
U
e
cistrone risultò più o meno
lo
stesso
(circa 1.500).
i
b
d
o
fece doppie infezioni con
R tà Benzer
coppie di mutanti r I I (tutti
i
s
appartenenti allo stesso cistrone)
r
e
v
sul ceppo B ed il lisato fagico
i
n
ottenuto lo piastrò
U sul ceppo K(l) (sul quale potevano crescere solo i
Dai dati di complementazione Benzer capì che le mutazioni rII
mappavano in due unità di funzione, che chiamò cistrone A e cistrone B.
ricombinanti selvatici) ed ottenne ricombinanti selvatici che si erano
originati da ricombinazione intragenica.
La metodologia
I dati di Benzer
confermarono ciò
che avevano intuito
Lewis e Oliver, cioè
che la ricombinazione può avvenire
anche tra siti all’
interno del gene
stesso. Questi siti
furono
all’epoca
chiamati reconi.
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
frequenza di
ricombinanti rII+ x2
ricombinazione =
totale progenie
tra due alleli
(numero di placche
presenti sul ceppo B)
”
o
I risultati ottenuti
”
o
i
B piastrando
l
i
Per ciascun incrocio Benzer faceva un
controllo
o
t
l
n
singolarmente i fagi di partenza (mutanti)
sulr ceppo B e poi
e
a
g
trasferendo il lisato su K(l). In questo
modo“era
possibile calcolare
C
r
etutti iocasi si vide che essa era
i
la frequenza di retromutazione. In
P
n
i
trascurabile rispetto alla frequenza
di
ricombinazione.
o rb
t
Benzer non osservò mai frequenze
di ricombinazione inferiori allo
r
U
e i in grado di mettere in evidenza
0,01% anche se il suobsistema era
d
o
frequenze di ricombinazione
dello
R tà 0,0001%. Ciò suggeriva l’esistenza
di un limite fisico ali di sotto del quale non può avvenire
s
r
ricombinazione.
e
v
Successivamente isi capì che la più piccola unità di ricombinazione
n
(il recone) coincide
U con la singola coppia nucleotidica.
Risultati strani
”
o
i
B
l
i
o
t Benzer
notò
che
l
n
r coppie di
e
alcune
a
g “mutanti
C
r
rII analizzati
e
i
P ino non davano mai luogo
o rb né a reversione
t
r
(retromutazione) né a
U
e
i
b
ricombinazione. Quali
d
o
R tà
tipi
di
mutazioni
i
possono dare questi
s
r
risultati?
e
v
i
n
U
complementazione
ricombinazione
intragenica
retromutazione +
assenza di placche
Le delezioni
”
o
i
l
BenzerBipotizzò che
i
t sirpotesse
lo trattare
n
e Cadi delezioni. Queste
g
r
“
e
infatti non possono
i
o
P in ricombinare con muo rb
t
tazioni puntiformi
r
U
e
sovrapposte.
i
b
d
o
Benzer mappò le delezioni
R tà
incrociandole tra loro: se
i
s
r
dall’incrocio tra due diverse
e
v
delezioni non si ottengono
i
n
ricombinanti selvatici, allora
U
esse si sovrappongono.
Due delezioni NON sovrapposte
possono invece ricombinare
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
Le delezioni scoperte da Benzer
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
La mappa per delezione
Benzer mappò le
delezioni tra
loro e poi le
utilizzò per
mappare più
velocemente
tutte le
mutazioni
puntiformi della
regione rII
tramite test di
complementazione a due a
due.
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
La tecnica genetica
”
o
i
B per esempio
l
i
Come si mappa un mutante utilizzando le delezioni?
Siano
t rlo
n
Esse definiscono tre aree del
D1 e D2 due delezioni del gene rIIA. e
a
g
C
gene, che chiameremo i, ii, iii.
r
“
eSe unaomutazione dà ricombinanti
i
P in
o rselvatici
solo se incrociata con
b
t
r
D2, allora risulterà localizzata
U
e
i
b
nella regione i.
m
m o
md
R tà
i
Se una mutazione non dà
ricombinanti selvatici né con D1 né con D2,
s
r nella regione ii.
allora risulterà localizzata
e
v
i
Se una mutazione
dà ricombinanti selvatici solo se incrociata con D1,
n
Ulocalizzata nella regione iii.
allora risulterà
La mappa fine dei loci rII - 1
”
o
i
B
l
i
Benzer utilizzò deficienze sempre più piccoleted infine
incrociò in tutti
o
l
n
r segmento per
i modi possibili i mutanti puntiformi interni
ad unoastesso
e
g
C
r
costruire una mappa genetica dettagliata.
“
e o
i
P intra loro non davano ricomSe due mutazioni puntiformi incrociate
oesse erano
b
binanti selvatici significava che
a carico dello stesso sito.
t
r
r
U
e
Benzer notò che i siti non
erano itutti uguali rispetto alla suscettibilità
b
d
o
alla mutazione. In molti
casi
si
R tà aveva una sola mutazione per sito, ma in
alcuni casi se ne avevanoi molte. I siti maggiormente mutabili furono
s
r
caldi).
chiamati hot spots (punti
e
v
i
Gli hot spots si mettono
in evidenza anche trattando con mutageni.
n
U
La mappa fine dei loci rII - 2
i
B
l
i
t rlo
n
e Ca
g
r
“
e
i
P ino
o rb
t
r
U
e
i
b
d
o
R tà
i
s
r
e
v
i
n
U
”
o
Conclusioni
”
o
i
B
l
i
t rlo
n
e Ca
g
r
“
e
L’analisi della struttura finei del gene
ha dimostrato che
o
P
n
i
ciascun gene è costituito
da
una
serie lineare di subo
b
t
r
r
elementi o siti che possono
essere
alterati dalle mutazioni
U
e
i
b
e possono essere
separati
dalla ricombinazione.
d
o
R tà che ciascun sito consiste in una
Successivamente si vide
i
s della doppia elica del DNA.
singola coppia di basi
r
e
v
i
n
U