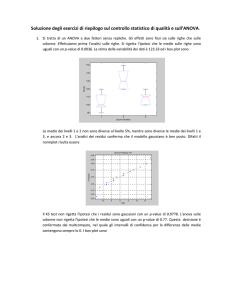
16/01/2014
Test di ipotesi
In una carta X -bar diremo che un processo è in controllo statistico se per ogni k , indice dei sottogruppi,
xk ∈ ( LCL,UCL).
Prendere questa decisione equivale ad effettuare un test di ipotesi.
Per effettuare un test di ipotesi è necessario:
• selezionare un campione casuale x1 , x2 ,… xn
• calcolare il valore di una statistica sul campione (nel CSQ è xk )
• caratterizzare un intervallo (nel CSQ è ( LCL, UCL)) che consenta di rigettare l'ipotesi iniziale quando la statistica test non
vi appartiene (nel CSQ equivale a dire che il processo non è
in controllo statistico)
1
16/01/2014
In generale, si tratta di decidere se il parametro di una certa popolazione assume un determinato valore.
Ad esempio, se la media della popolazione µ assume il valore µ0 .
Nel caso del CSQ, si tratta di decidere se la media della popolazione assume valore pari al target.
H 0 : µ = µ0 ( ipotesi nulla )
H1 : µ ≠ µ0 ( ipotesi alternativa )
Nell’esempio del contenuto dei flaconi, potremmo essere interessati
a decidere se la media della popolazione coincide con il valore target 95.
La media campionaria risulta essere 98.51. Il valore 98.51 è sufficientemente vicino al valore 95 per ritenere che la media della popolazione
coincide con 95?
Per rispondere a questo quesito, abbiamo bisogno di fissare una distanza del valore osservato per la media campionaria dal valore target 95:
se 98.51 dista da 95 meno di questa distanza prefissata, allora riterremo
95 un valore credibile per la media della popolazione.
In sostanza si tratta di fissare un intervallo, che contenga 95 (in genere
si assume sia centrato su 95) tale che se, 98.51 appartiene a tale intervallo, si ritiene plausibile, l’ipotesi nulla:
H 0 : µ = 95 ( ipotesi nulla )
98.51
?
95
REGIONE DI ACCETTAZIONE
2
16/01/2014
H1 : µ ≠ 95 ( ipotesi alternativa )
95
98.51
REGIONE CRITICA
?
Per evitare che questa scelta sia soggettiva, possiamo fissare a-priori la
probabilità di commettere un errore prendendo una decisione sulla
media della popolazione.
Che tipi di errori si possono commettere?
ERRORE DI I TIPO
α = P ( si rigetta H 0 - a posteriori - quando H 0 è vera - a priori )
ERRORE DI I TIPO
α ← livello di significatività del test
La probabilità dell’evento complementare si riferisce a una decisione
corretta:
1 − α = P ( si accetta H 0 - a posteriori - quando H 0 è vera - a priori )
3
16/01/2014
ERRORE DI II TIPO
β = P ( si rigetta H1 - a posteriori - quando H1 è vera - a priori )
1 − β ← potenza del test
ERRORE DI II TIPO
La probabilità dell’evento complementare si riferisce a una decisione
corretta:
1 − β = P ( si accetta H1 - a posteriori - quando H1 è vera - a priori )
1 − β = P ( si rigetta H 0 - a posteriori - quando H 0 è falsa - a priori )
7
1 − α = P ( si accetta H 0 - a posteriori - quando H 0 è vera - a priori )
si accetta H 0 quando µ =µ0 ⇒ x ∈ ( µ0 − δ , µ0 + δ )
Quando µ =µ0 , X ≈ N ( µ0 ,...) ⇒ ( µ0 − δ , µ0 + δ )
1 − α ⇒ δ =?
σ
δ = zα /2
−δ
µ0
n
σ
n
δ
Statistica osservata
4
16/01/2014
Regione di accettazione
REGIONE DI
ACCETTAZIONE
α ← livello di significatività del test
?
?
Quale si sceglie di fissare?
P ( STATISTICA ∈ regione di accettazione ) = 1 − α
α = 0.10, 0.05, 0.01
Come?
σ
1 − α = P µX ∈∈Xµ ± zα /2
n
σσ
1 − α = P X ∈ µ0±±zzαα/2/2 µµ==µµ00
nn
?
9
Si rigetta l’ipotesi nulla…
Pertanto fissare l’errore di I
tipo equivale a fissare la
regione di accettazione, e
viceversa.
µ0
Statistica osservata
10
5
16/01/2014
L’errore di II tipo
µ = µ0
µ = µ1
µ1
µ0
Statistica osservata
11
La potenza del test
µ = µ0
µ = µ1
E’ un valore che può
essere calcolato solo
se si conosce un valore
alternativo. Quale?
µ0
µ1
Statistica osservata
12
6
16/01/2014
Le statistiche test vanno scelte a secondo delle informazioni che si
hanno sul campione.
Ad esempio,
1) il campione proviene da una popolazione gaussiana (qqnorm )
2) la varianza è nota
σ
X ≈ N µ,
n
Ad esempio,
1) il campione proviene da una popolazione gaussiana (qqnorm )
2) la varianza non è nota
X −µ
≈ Tn −1
S
n
Ad esempio,
1) il campione non proviene da una popolazione gaussiana (qqnorm)
2) la varianza non è nota
S
3) la taglia supera 30
X ≈ N µ,
n
Torniamo all’esempio dei flaconi, e verifichiamo se il campione proviene
da una popolazione con media 95.
Non conoscendo la varianza teorica, la statistica da utilizzare ha legge
T-Student.
La function è t.test().
> t.test(data,mu=95)
One Sample t-test
data: data
t = 4.5358, df = 119, p-value = 1.379e-05
alternative hypothesis: true mean is not equal to 95
95 percent confidence interval:
96.98146 100.05187
sample estimates:
A quale conclusione giungiamo?
mean of x
98.51667
7
16/01/2014
Legge T-Student
con 119 gradi di libertà
Regione
critica?
Statistica test=4.53
CHE COSA
E’ IL P-VALUE?
Se p ≥ α ⇒ stat ∈ reg.accett. ⇒ H 0 non si rigetta
Se p < α ⇒ stat ∉ reg.accett. ⇒ H 0 si rigetta
L’area a destra
della
statistica test.
Se
p − value < α / 2
⇒ si rigetta H 0
Nell’esempio, il p-value
è molto inferiore a 0.025,
quindi l’ipotesi nulla
si rigetta.
Statistica
test=4.53
8
16/01/2014
Avendo stabilito che la media della popolazione non è 95, potremmo
essere interessati a sapere se la media è maggiore o minore di 95.
In quel caso si effettua un test a una coda.
A 1 coda
H 0 : µ = µ0
H1 : µ > µ 0
REGIONE DI
ACCETTAZIONE
> t.test(data,alternative=c("greater"),mu=95)
One Sample t-test
data: data
t = 4.5358, df = 119, p-value = 6.895e-06
REGIONE
CRITICA
Si rigetta l’ipotesi nulla
perché la media
campionaria ha un valore
che ricade nella regione
critica.
Si rigetta l’ipotesi nulla, in favore di
quella alternativa perché il p-value
é inferiore a 0.05.
Potremmo usare un valore del livello di significatività diverso da 0.05.
> t.test(data,alternative=c("greater"),mu=95,conf.level=0.99)
One Sample t-test
data: data
t = 4.5358, df = 119, p-value = 6.895e-06
alternative hypothesis: true mean is greater than 95
99 percent confidence interval:
96.68839 Inf
sample estimates: mean of x 98.51667
> t.test(data,alternative=c("greater"),mu=95)
One Sample t-test
data: data
t = 4.5358, df = 119, p-value = 6.895e-06
alternative hypothesis: true mean is greater than 95
Cambia solo
95 percent confidence interval:
l’intervallo di
97.23138 Inf
confidenza
sample estimates: mean of x 98.51667
9
16/01/2014
Per il caso
1) il campione non proviene da una popolazione gaussiana (qqnorm)
2) la varianza non è nota
S
3) la taglia supera 30
X ≈ N µ,
n
si continua ad utilizzare il t.test() poiché la v.a. T-Student è approssimata da
una v.a. gaussiana
Per il caso
1) il campione proviene da una popolazione gaussiana (qqnorm )
2) la varianza è nota
σ
X ≈ N µ,
n
In questo caso la libreria da utilizzare è BSDA (basic statistical data analysis) che a sua
volta necessita di e1071, class, lattice. La function è z.test(). La sintassi è:
> z.test(difetti, y = NULL, alternative = "two.sided", mu = 20, sigma.x = 7.8, sigma.y = NULL,
+ conf.level = 0.99)
Il risultato per il dataset difetti.txt è
> z.test(difetti, y = NULL, alternative = "two.sided", mu = 20, sigma.x = 7.8, sigma.y =
+ NULL,conf.level=0.99)
One-sample z-Test
data: difetti
z = -5.922, p-value = 3.181e-09
alternative hypothesis: true mean is not equal to 20
99 percent confidence interval:
7.898483 15.234850
sample estimates:
In tal caso il p-value è molto inferiore a 0.05.
mean of x
11.56667
Pertanto l’ipotesi nulla si rigetta.
>
10
16/01/2014
CURVA CARATTERISTICA OPERATIVA
Diremo che il processo è in controllo statistico se per ogni
i, indice dei sottogruppi, xi ∈ ( LimInf , LimSup).
Regione di accettazione
α = P (rigettare H 0 | µ = µ0 ) = P( xt ∉ ( LCL, UCL) | µ = µ0 )
β = P(rigettare H1 | µ ≠ µ0 ) = P( xt ∈ ( LCL, UCL) | µ ≠ µ0 )
FALSO ALLARME
MANCATO ALLARME
21
Non avendo ipotesi alternative certe, immaginiamo che µ = µ1 = µ0 + kσ
Se la popolazione è gaussiana, allora
β = P ( xt ∈ ( LCL, UCL) | µ = µ0 + kσ )
UCL − ( µ0 + kσ )
LCL − ( µ0 + kσ )
= Φ
−Φ
σ/ n
σ/ n
Il plot dei valori assunti da questo parametro per un opportuno valore
di k, si chiama curva caratteristica operativa.
In R l’istruzione per effettuare questo grafico è oc.curves()
> obj<-qcc(data,type='xbar')
> oc.curves(obj)
11
16/01/2014
vari valori di k
Specificando un valore pari a TRUE per la variabile identify, è possibile
identificare interattivamente dei punti sul grafico
Vari valori di beta possono essere ottenuti specificando un parametro
di output:
> beta<-oc.curves(obj)
Se la media del processo è
95+0.4*st.dev, per n=5, la
prob. di un mancato allarme è 0.98
Per valori di k inferiori, aumenta la
probabilità di un mancato allarme.
12
16/01/2014
ALTRO USO DELLA CURVA OPERATIVA
Nella progettazione delle carte di controllo è necessario specificare sia la dimensione
del campione che la frequenza di campionamento.
• Più grande è il campione più sensibile è il rilevamento di una variazione all’interno del
processo.
• La pratica corrente tende a diminuire la dimensione del campione e ad aumentare la
frequenza di campionamento.
Si fissa β , e si cerca quel valore di n tale che
UCL − ( µ0 + kσ )
LCL − ( µ0 + kσ )
−Φ
σ/ n
σ/ n
σ
σ
Se UCL = µ0 + 3
e LCL = µ0 − 3
, allora
β = Φ
n
(
n
) (
β = Φ 3 − k n − Φ −3 − k n
)
25
Si fissa β , e si cerca quel valore di zβ tale che Φ ( zβ ) − Φ ( − zβ ) = β
ossia, ricordando le proprietà della gaussiana...
β +1
2Φ ( zβ ) − 1 = β ⇒ zβ = Φ −1
⇒ zβ = 3 − k n
2
⇒ è possibile ricavare n
3 − zβ
⇒n=
k
2
Ad esempio per β = 0.3 > qnorm(1.3/2)
[1] 0.3853205
e per k = 1
> (3-qnorm(1.3/2))^2
[1] 6.836549
n=6
13
16/01/2014
IL CONFRONTO TRA DUE POPOLAZIONI
Cambiamento della macchina per imballaggio?
Sono statisticamente differenti le due produzioni?
27
CONFRONTO TRA DUE POPOLAZIONI
( x1 , x2 ,… , xn )
POPOLAZIONE 1
( y1 , y2 ,…, yn )
POPOLAZIONE 2
• I due campioni provengono dalla stessa popolazione?
• Le due popolazioni hanno la stessa media?
• Le due popolazioni hanno la stessa varianza?
• Se provengono da popolazioni distinte, queste sono indipendenti
oppure c’è una qualche forma di relazione tra loro?
14
16/01/2014
Le due popolazioni hanno la stessa varianza?
Supponiamo di aver monitorato la produzione di una sbarretta metallica
in una azienda in due periodi diversi dell’anno. Vogliamo verificare se la
produzione è rimasta costante nel tempo.
I dati sono in due files distinti: produzione1.txt e produzione2.txt
> prod1<-matrix(scan('C:/Programmi/R/R-3.0.2/produzione1.txt'),ncol=1, byrow=TRUE)
Read 20 items
> prod2<-matrix(scan('C:/Programmi/R/R-3.0.2/produzione2.txt'),ncol=1, byrow=TRUE)
Read 20 items
>var.test(prod1, prod2, ratio = 1, alternative =
+ "two.sided", conf.level = 0.95)
> var(prod1);var(prod2)
F test to compare two variances
[,1]
data: prod1 and prod2
[1,] 1.574374
F = 1.44, num df = 19, denom df = 19, p-value = 0.4341
[,1]
alternative
hypothesis: true ratio of variances is not equal
[1,] 1.093336
to 1
95 percent confidence interval: 0.5699588 3.6380212
Non si rigetta
sample estimates: ratio of variances 1.439973
29
F-TEST
DISTRIBUZIONE DI FISHER
Siano χ n21 e χ n22 due var. al. con legge chi-quadrato con gradi
(
) (
di libertà rispettivamente n1 e n2 . La var. al. χ n21 / n1 / χ n22 / n2
)
ha legge di Fisher di gradi di libertà ( n1 , n2 ) .
S12
≈ Fisher ( n1 − 1, n2 − 1)
S22
(n1 − 1)
(n2 − 1)
S12
σ
2
1
S
σ
2
2
2
2
≈ χ12
≈ χ 22
χ12
n1 − 1
χ 22
n2 − 1
15
16/01/2014
Le due popolazioni hanno la stessa media?
La function da usare è sempre la stessa, t.test()
> t.test(prod1, prod2, mu = 0, alternative ="two.sided",paired = FALSE, var.equal =
+ TRUE, conf.level = 0.95)
Two Sample t-test
Non si rigetta l’ipotesi nulla.
data: prod1 and prod2
t = 1.3622, df = 38, p-value = 0.1812
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval: -0.2418487 1.2368487
sample estimates: mean of x mean of y
100.2350 99.7375
Le procedure sono distinte a secondo che le varianze siano uguali oppure diverse.
PAIRED va posto uguale a TRUE quando i due campioni casuali si riferiscono alle stesse unità statistiche e quindi c’è una forma di dipendenza tra i due campioni. In tal caso
le taglie devono essere uguali.
Se PAIRED è posto uguale a FALSE, le taglie dei due campioni possono essere diverse.
I due campioni provengono dalla stessa popolazione?
Il test di Kolmogorov-Smirnov consente di stabilire se due campioni provengono da due popolazioni aventi la medesima legge di probabilità
L’idea è quella di
costruire le funzioni
di ripartizioni
empiriche per i due
campioni e poi
di valutare la distanza
massima tra queste
ultime
In R la function da usare è ks.text(). Nella variabile di input alternative è possibile specificare
se l’ipotesi alternativa prevede che una funzione di ripartizione sia maggiore dell’altra o viceversa.
16
16/01/2014
> ks.test(prod1,prod2)
Two-sample Kolmogorov-Smirnov test
data: prod1 and prod2
D = 0.25, p-value = 0.5596
alternative hypothesis: two-sided
Warning message:
In ks.test(prod1, prod2) : cannot compute exact p-value with ties
> plot(ecdf(prod1),xlim=range(c(prod1,prod2)),
+ main='Confronto f.r.empiriche')
> par(new=TRUE)
> plot(ecdf(prod2),add=TRUE,col='red')
> legend(100,0.3,c("prod1","prod2"),
+ lty=c(1,1),lwd=c(2.5,2.5),col=c("black","red"))
>
• Se provengono da popolazioni distinte, queste sono indipendenti
oppure c’è una qualche forma di relazione tra loro?
Per rispondere a questo quesito, è necessario prima calcolare il coefficiente di correlazione tra i due datasets:
> cor.test(prod1,prod2)
Pearson's product-moment correlation
data: prod1 and prod2
t = 0.9373, df = 18, p-value = 0.361
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.2507354 0.6008810
sample estimates:
cor
0.2157209
Non si rigetta
l’ipotesi che li
coefficiente di
correlazione è
nullo.
Se le popolazioni da cui provengono i due campioni hanno legge gaussiana, allora risultano indipendenti. Poiché abbiamo mostrato che le due
popolazioni hanno medesima distribuzione, basta provare per un solo
campione che la popolazione da cui proviene segue una legge gaussiana.
17
16/01/2014
In questo caso, il valore del secondo parametro deve essere una stringa numerica contenente il nome della funzione distribuzione del modello teorico che si vuole testare.
> ks.test(prod1,"pnorm",mean(prod1),sd(prod1))
One-sample Kolmogorov-Smirnov test
data: prod1
D = 0.1326, p-value = 0.8289
alternative hypothesis: two-sided
Siccome il p-value > 0.05
l’ipotesi nulla non si rigetta.
Trattandosi di campioni gaussiani, con un coefficiente di correlazione
quasi nullo, si può ritenere siano indipendenti.
Il ks-test può essere usato solo con modelli teorici continui. Nel caso
discreto, è possibile usare il test chi-quadrato.
Ad esempio, potremmo essere interessati a sapere se i dati del file
difetti.txt hanno effettivamente legge binomiale.
TEST CHI-QUADRATO
In questo test, vengono confrontati gli istogrammi costruiti sul campione con quelli che
si ottengono adoperando un modello teorico.
Il campione va ripartito in modalità e per ciascuna di queste va contato il numero di occorrenze
nel campione.
> setwd("C:/Programmi/R/R-3.0.2/")
> data<-read.table("difetti.txt",header=TRUE)
> attach(data)
> boundaries<-seq(0,max(difetti))
> tab.out<-table(cut(difetti,boundaries))
> appoggio<-cbind(tab.out)
>
La function cbind crea una matrice con il risultato della
function table. La function table crea una tavola di contingenza,
con il risultato della function cut, che calcola le frequenze
di occorrenza della modalità del campione. Il risultato è
> appoggio[1:24]
[1] 0 0 0 1 2 2 2 2 3 3 1 3 2 1 2 1 1 1 0 1 0 1 0 1
18
16/01/2014
Ad esempio, per il file difetti.txt, il modello teorico è quello di una binomiale di parametri n=50
e p pari alla linea centrale della carta di controllo p, ossia 0.23.
Per un modello teorico discreto, l’analogo dell’istogramma è il diagramma delle
frequenze assolute.
Il test chi-quadrato valuta le differenze tra queste due curve, normalizzate all’ordine di
grandezza delle frequenze teoriche.
La libreria di riferimento è la vcd.
> gf<-goodfit(difetti, type=“binomial", par = list(prob = 0.23, size = 50))
> summary(gf)
Goodness-of-fit test for binomial distribution
X^2 df P(> X^2)
Pearson
NaN 50
NaN
Likelihood Ratio 34.96249 17 0.006292134
Warning message:
In summary.goodfit(gf) : Chi-squared approximation
may be incorrect
Per ottenere il grafico , usare le variabili
gf$observed; gf$count, gf$fitted che si ottengono
usando str(gf).
Il motivo per cui il test non restituisce un valore finito per il p-value è la presenza di troppi
0 nelle frequenze osservate. Vale infatti la cosiddetta regola empirica del pollice che prevede
in ciascuna classe un numero di elementi maggiori di 5.
>
>
>
>
attach(data)
boundaries<-seq(0,max(difetti),3)
osservate<-table(cut(difetti,boundaries))
osservate
(0,3] (3,6] (6,9] (9,12] (12,15] (15,18] (18,21] (21,24]
0
5
7
7
5
3
1
2
> boundaries<-c(0,9,12,15,50)
> osservate<-table(cut(difetti,boundaries))
> osservate
Regola
del
pollice
(0,9] (9,12] (12,15] (15,50]
12
7
5
6
In tal caso la function da usare è chisq.test() che prende in input le
frequenze osservate ed il vettore della massa di probabilità. Quest’
ultimo viene trasformato nel vettore delle frequenze attese
all’interno della function.
19
16/01/2014
Il valore 0 non è presente nel campione casuale. Posso dunque lasciare inalterato l’intervallo
(0,9].
> osservate
(0,9] (9,12] (12,15] (15,50]
12
7
5
6
> attese<-pbinom(boundaries[2],50,0.23)
> attese[2:3]<-pbinom(boundaries[3:4],50,0.23)-pbinom(boundaries[2:3],50,0.23)
> attese[4]<-1-sum(attese[1:3])
> sum(attese)
[1] 1
> chisq.test(osservate, p=attese)
REGOLA EMPIRICA DEL
POLLICE
Chi-squared test for given probabilities
data: osservate
X-squared = 9.0582, df = 3, p-value = 0.02853
Warning message:
In chisq.test(osservate, p = attese) :
Chi-squared approximation may be incorrect
Supponiamo di voler verificare per questo vettore se segue una legge binomiale.
ESEMPIO:
14
15
13
14
14
12
12
12
14
13
10
13
13
14
6
12
11
12
11
11
> dati<-c(14,15,13,14,14,14,13,11,12,13,14,12,12,10,6,11,12,13,12,11)
I parametri n e p vengono stimati direttamente dalla function goodfit usando il metodo
di massima verosimiglianza se specifichiamo un certo parametro di input:
> gf<-goodfit(dati, type=“binomial", method='ML')
> summary(gf)
Goodness-of-fit test for binomial distribution
X^2 df P(> X^2)
Likelihood Ratio 10.99785 5 0.0514226
20
16/01/2014
> str(gf)
List of 7
$ observed: num [1:16] 0 0 0 0 0 0 1 0 0 0 ...
$ count : int [1:16] 0 1 2 3 4 5 6 7 8 9 ...
$ fitted : num [1:16] 1.35e-10 9.22e-09 …..
$ type : chr "binomial"
$ method : chr "ML"
$ df : num 5
$ par :List of 2
..$ prob: num 0.82
..$ size: int 15
- attr(*, "class")= chr "goodfit"
> plot(gf$count,gf$observed,type='s',col='red',main='Observed vs Theoretical')
> par(new=TRUE)
> plot(gf$count,gf$fitted,type='l',lwd=4,col='green',ylim=range(0,5),ylab=' ')
>plot(gf)
Serve a capire su quale
osservazione agire per
migliorare il fitting.
Esempio: supponiamo di aver lanciato
un dado e di voler verificare se è stato
truccato. Le frequenze di occorrenza
risultano:
> osservate<-c(18,24,15,25,17,23)
> attese<-rep(1/6,6)
> sum(attese)
[1] 1
> sum(osservate)
[1] 122
> chisq.test(osservate, p=attese)
Chi-squared test for given probabilities
data: osservate
X-squared = 4.2951, df = 5, p-value = 0.5078
21
16/01/2014
Esempio: supponiamo che il numero di prove necessario prima di avere un guasto in
un determinato sistema risulti essere:
> osservate<-c(24, 21, 15, 12, 10, 5, 4,2)
> boundaries<-c(1, 2, 3, 4, 5, 6, 8, 9)
Verificare se è possibile assumere valido un modello geometrico.
> media<-sum(osservate*boundaries)/sum(osservate)
> p<-1/media
> attese<-dgeom(boundaries[1:5],p)
> attese[6]<-dgeom(6,p)+dgeom(7,p)
> attese[7]<-dgeom(8,p)
> attese[8]<-1-sum(attese[1:7])
> sum(attese)
[1] 1
> chisq.test(osservate, p=attese)
Chi-squared test for given probabilities
data: osservate
X-squared = 54.7321, df = 7, p-value = 1.685e-09
Warning message:
In chisq.test(osservate, p = attese) :
Chi-squared approximation may be incorrect
Z-TEST PER DUE CAMPIONI
Assumiamo che nella produzione della sbarretta metallica dell’azienda
presa in considerazione siano note le varianze nei due diversi periodi
dell’anno. In tal caso, invece del test t conviene usare lo z-test.
Per il primo dataset immaginiamo che la deviazione standard sia 1.25, per il secondo
dataset immaginiamo che la deviazione standard sia 1.
> z.test(prod1, prod2, alternative = "two.sided", sigma.x = 1.25, sigma.y=1)
Two-sample z-Test
data: prod1 and prod2
z = 1.3899, p-value = 0.1646
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval: -0.2040603 1.1990603
sample estimates: mean of x ; mean of y ; 100.2350 99.7375
22
16/01/2014
T-PAIRED TEST
> t.test(prod1, prod2, mu = 0, alternative ="two.sided",paired = FALSE, var.equal =
+ TRUE, conf.level = 0.95)
Quando la variabile PAIRED è posta uguale a TRUE, le unità statistiche sulle quali viene
effettuato il test sono le stesse.
Esempio: Una scuola ha arruolato un nuovo istruttore e vuole verificare l’efficacia di un nuovo programma di training proposto, confrontando il tempo medio
di 10 atleti che corrono sui 100 metri.
> a<-c(12.9, 13.5, 12.8, 15.6, 17.2, 19.2, 12.6, 15.3, 14.4, 11.3)
> b<-c(12.7, 13.6, 12.0, 15.2, 16.8, 20.0, 12.0, 15.9, 16.0, 11.1)
> t.test(a,b,paired=TRUE)
Paired t-test
data: a and b
t = -0.2133, df = 9, p-value = 0.8358
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval: -0.5802549 0.4802549
sample estimates: mean of the differences -0.05
IL CONFRONTO TRA DUE POPOLAZIONI
Cambiamento della macchina per imballaggio?
Sono statisticamente differenti le due produzioni?
46
23
16/01/2014
TEST SULLE PROPORZIONI
H 0 : p1 = p2
H1 : p1 > p2
Per effettuare un test sulle proporzioni è necessario usare la function
prop.test() . Come parametro di input è necessario costruire una tabella
a doppia entrata contenente il numero di pezzi difettosi riscontrati nel
primo campione e il numero di pezzi difettosi riscontrati nel secondo
campione.
> sum(value[labels][1:28])
[1] 301
> sum(value[labels][29:52])
[1] 133
Yates’s correction
Difettosi
Totale
primo
301
1400=28*50
secondo
133
1200=24*50
Totale
480
2600
> prop.test(x=c(301,133),n=c(1400,1200),correct=FALSE)
2-sample test for equality of proportions without continuity
correction
data: c(301, 133) out of c(1400, 1200)
X-squared = 50.4187, df = 1, p-value = 1.242e-12
alternative hypothesis: two.sided
95 percent confidence interval: 0.07626364 0.13206970
sample estimates: prop 1
prop 2
0.2150000 0.1108333
ANALISI DEI RESIDUI
Nella costruzione di un modello teorico lineare per descrivere la relazione esistente tra due
campioni casuali, era stato tralasciato un punto che rientra nella validazione del modello: ossia
provare che i residui del modello di regressione seguono una legge gaussiana di media nulla
e deviazione standard pari all’errore standard residuo.
Si definiscono residui le distanze
tra i valori della variabile dipendente ottenuti mediante la retta
di regressione e quelli osservati.
ei = yi − yˆi
Perchè il modello sia valido
è necessario provare che
ε ∼ N(0,σ 2 )
Per la varianza usiamo il valore del residual
standard error che fornisce la function lm di R
24
16/01/2014
L’analisi della regressione è una tecnica statistica per modellare e
investigare le relazioni tra due (o più) variabili.
Nella tavola è riportata la % di purezza di ossigeno,
rilasciata in un processo di distillazione chimica, e il
livello di idrocarbonio, presente nel condensatore principale di unità di distillazione.
Dati
salvati
in
un file
Osservazioni Liv.Idrocarbonio Purezza
1
0,99
90,01
2
1,02
89,05
3
1,15
91,43
4
1,29
93,74
5
1,46
96,73
6
1,36
94,45
7
0,87
87,59
8
1,23
91,77
9
1,55
99,42
10
1,4
93,65
11
1,19
93,54
12
1,15
92,52
13
0,98
90,56
14
1,01
89,54
15
1,11
89,85
16
1,2
90,39
17
1,26
93,25
18
1,32
93,41
19
1,43
94,98
20
0,95
87,33
49
>result<-lm(datiy ~ datix)
>summary(result)
Residuals:
Min
1Q
Median
3Q Max
-1.83029 -0.73334 0.04497 0.69969 1.96809
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 74.283
1.593
46.62
< 2e-16 ***
datix
14.947
1.317
11.35 1. 1.23e-09 ***
--Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 1.087 on 18 degrees of freedom
F-statistic: 128.9 on 1 and 18 DF, p-value: 1.227e-09 (hp_0:m=0)
25
16/01/2014
> str(result)
…
$ residuals : Named num [1:20] 0.9287 -0.4797 -0.0429 0.1744 0.6234 ...
Dovendo dimostrare che i residui hanno legge gaussiana,
un test più robusto del KS-test è il test di Shapiro-Wilk
> shapiro.test(result$residuals)
Shapiro-Wilk normality test
data: result$residuals
W = 0.9796, p-value = 0.9293
> ks.test(result$residuals,"pnorm",0,1.087)
One-sample Kolmogorov-Smirnov test
data: result$residuals
D = 0.0904, p-value = 0.9916
alternative hypothesis: two-sided
Si basa sul confronto tra la varianza
campionaria e una varianza ottenuta
usando dei pesi particolari.
Il test di Shapiro-Wilks è molto usato per verificare se un campione casuale ha legge gaussiana.
Infatti quando sussiste tale ipotesi, vi sono vari metodi che consentono
di effettuare i test in condizioni di robustezza.
STATISTICA PARAMETRICA
Quando tale ipotesi non è verificata, i metodi cui si ricorre rientrano
nell’ambito della statistica non parametrica.
Ad esempio si consideri un insieme di dati aventi tendenza centrale (8),
ma con legge uniforme.
x<-4+8*runif(90,min=0,max=1)
Come valore di riferimento della tendenza centrale usiamo la mediana
H0 : M = M 0
H1 : M ≠ M 0
Ad esempio, vogliamo verificare se la mediana del
dataset così costruito è 6.
Se fosse 6, allora circa una metà dei valori di x dovrebbe essere inferiore a 6.
26
16/01/2014
Pertanto la probabilità che un valore di x sia
inferiore a 6 dovrebbe essere circa il 50%.
Ossia il numero di segni positivi e negativi
del campione casuale dovrebbe essere circa
lo stesso.
> install.packages('plyr')
> library('plyr')
> count(sign(x-6))
x freq
1 -1 17
2 1 73
>
Il test che verifica se il numero dei segni negativi nel campione casuale è
circa la metà di tutto il campione è il sign test (o test binomiale).
> binom.test(17,90)
Exact binomial test
data: 17 and 90
number of successes = 17, number of trials = 90, p-value = 1.948e-09
alternative hypothesis: true probability of success is not equal to 0.5
95 percent confidence interval: 0.1140597 0.2851131
sample estimates: probability of success 0.1888889
CONFRONTO TRA DUE CAMPIONI IN MANCANZA
DELL’IPOTESI DI POPOLAZIONE GAUSSIANA
Analogo T-test - PAIRED=FALSE
Mann-Whitney test
Analogo T-test - PAIRED=TRUE
Wilcoxon test
Ipotesi nulla:
Ipotesi nulla:
i due campioni provengono da una Differenze tra le osservazioni accopcomune distribuzione di probabilità piate provengono da una distribuzione di probabilità simmetrica attorno
allo 0.
Ipotesi alternativa:
c’è un ordinamento stocastico tra
le due distribuzioni di probabilità Ipotesi alternativa:
Differenze tra le osservazioni accopche generano i campioni
piate provengono da una distribuzione di probabilità asimmetrica.
27
16/01/2014
> wilcox.test(x,y,paired=…,alternative=‘two sided’)
Se paired=FALSE, allora si ritiene che i due campioni sono indipendenti
(Mann-Whitney).
Se paired =TRUE, allora si ritiene che i due campioni siano accoppiati
(Wilcoxon).
Esempio: Un’azienda che produce scarpe da trekking vuole lanciare una
nuova linea. Per questo motivo seleziona 13 scalatori, ad ognuno dei
quali fa indossare una scarpa da trekking della vecchia linea produttiva
ed una scarpa da trekking della nuova linea. Viene poi misurato il numero di chilometri percorsi prima che inizi il deterioramento della scarpa.
Dopo aver effettuato una analisi descrittiva per entrambi i campioni,
stabilire con un test di ipotesi se c’è differenza.
> old<-c(460,420,520,515,490,490,500,550,480,530,518,515,475)
> new<-c(530,525,500,505,520,450,495,575,474,515,490,480,493)
campione casuale old
campione casuale new
Visto che è necessario verificare se entrambi i campioni hanno legge gaussiana, conviene usare lo Shapiro-Wilk test.
> shapiro.test(old)
Shapiro-Wilk normality test
data: old
W = 0.9553, p-value = 0.6806
> shapiro.test(new)
Shapiro-Wilk normality test
data: new
W = 0.9634, p-value = 0.8051
28
16/01/2014
Poiché vengono usati sempre gli stessi scalatori per testare le scarpe, effettuiamo un T-paired test.
> t.test(old,new, mu = 0, alternative ="two.sided", paired = TRUE)
Paired t-test
data: old and new
t = -0.5824, df = 12, p-value = 0.5711
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-32.46024 18.76793
sample estimates: mean of the differences -6.846154
Se effettuando lo Shapiro-Wilk test uno dei due campioni fosse risultato
non normale, allora la sintassi da usare sarebbe stata:
> wilcox.test(old,new,paired=TRUE,alternative="two.sided")
Wilcoxon signed rank test
data: old and new
V = 45, p-value = 1
alternative hypothesis: true location shift is not equal to 0
29