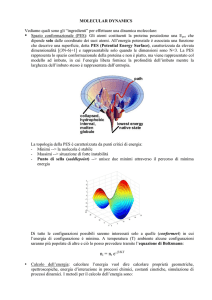
Molecole biologiche
Macromolecole:
Proteine e Acidi nucleici
catene polimeriche di
aminoacidi e nucleotidi
formate da 103 a 106 atomi, con una massa molecolare
compresa tra 103 a 1012 amu (1 amu è circa la massa di un
atomo di idrogeno).
Il loro comportamento è determinato da
Solvente (acqua, pH)
Flessibilità (perché sono catene lunghe)
Tempi caratteristici
Scale di tempi e di distanze
•
•
•
•
•
•
Movimenti locali:
fluttuazioni e vibrazioni atomiche
movimenti delle catene laterali
1 ÷ 500 pm
10-15 ÷ 10-1 s
Movimenti di corpo rigido:
movimento delle eliche
100 ÷ 1000 pm
10-9 ÷ 1 s
Movimenti su larga scala:
transizioni elica - coil
dissociazione/associazione
folding e unfolding
500 ÷ 50000 pm
10-7 ÷ 104 s!
I polipeptidi: catene molecolari
Un polipeptide (proteina) consiste in una lunga catena di
aminoacidi:
Proteine: proprietà strutturali
Molecole lineari formate da sequenze
di 20 diversi tipi di aminoacidi (Natomi =
40 ÷ 103 e più…).
Gli aminoacidi sono costituiti da un
gruppo peptidico e una catena laterale
(side chain)
I gradi di libertà rotazionali dei legami
peptidici rendono il backbone molto
flessibile
I legami peptidici formano il backbone
della proteina
Livelli di struttura
Primaria: la specifica
sequenza degli aminoacidi
Secondaria: motivi conformazionali locali:
α-eliche e β-sheets
Terziaria: la vera struttura 3D
Quaternaria: organizzazione
spaziale delle sub-unità molecolari
in un complesso proteico
Esempio: triose phosphate isomerase
Rappresentazione all-atom,
colorata a seconda del tipo di
atomo
Rappresentazione semplificata
che descrive il backbone colorato
per tipo di struttura secondaria
Superficie esterna accessibile al
solvente, colorata a seconda del
tipi (acido - rosso, basico - blu,
polare - verde, nonpolare –
bianco) di residuo
DNA (cenno)
Fino a circa 2 108 paia di nucleotidi (4+1 tipi),
ognuno di circa 20 atomi.
Un backbone abbastanza rigido formato da
pentosio e fosfato alternati.
Chiralità (⇒ attività ottica) dei legami della
catena, che si attorciglia spontaneamente.
Legami a idrogeno tra coppie di polimeri:
struttura a doppia elica.
Limiti delle tecniche sperimentali
risoluzione spaziale
misure ad alta risoluzione di strutture molecolari sono
possibili solo per sistemi relativamente rigidi
risoluzione energetica
misura delle energie di interazione e deformazione
molecolare difficoltosa (i moti lenti di grande ampiezza sono
spesso associati a energie piccolissime)
risoluzione temporale
i primissimi eventi dei processi biologici sono veloci, quindi
di difficile misurazione
Limiti delle tecniche computazionali
Sistemi biomolecolari troppo complessi per una descrizione che
contenga tutta la meccanica quantistica. Si usa invece la
meccanica classica con funzioni empiriche per descrivere le
interazioni tra gli atomi del un sistema molecolare
Simulazione del comportamento di un sistema molecolare su un
computer: anche con queste semplificazioni, solo un numero
limitato di atomi (di solito 103÷107 atomi, ≪NA≃6·1023), per un
limitato periodo di tempo (1÷10 ns) può essere simulato. Questo
permette di studiare solo sistemi piccoli, per fenomeni veloci
(tempi di rilassamento brevi)
Campionatura limitata dello spazio delle conformazioni (un
concetto statistico!) di una macromolecola: necessario l'utilizzo
dei dati sperimentali per restringersi alla regione interessante
Importanza della complementarietà dell'approccio teoricosperimentale!
Complementarità di tecniche
computazionali e sperimentali
L’utilizzo complementare di tecniche di tipo
sperimentale e di tipo computazionale è
attualmente l’approccio ottimale per lo studio della
dinamica delle macromolecole e di molti processi
biologici relativi
in particolare gli aspetti strutturali del problema,
ovvero lo studio della conformazione, o variazione di
conformazione, di una molecola biologica in
relazione alla sua attività
Alcune Applicazioni
Conoscere, comprendere, spiegare i meccanismi che portano alla
struttura nativa a risoluzione atomica della molecola per
comprendere, spiegare, ed eventualmente anche modificare, la
sua attività biologica.
Alcune applicazioni
Monitorare i cambiamenti strutturali indotti su peptidi o
proteine da parte di membrane biologiche
Alcune applicazioni
Effettuare mutazioni puntiformi, che possono fornire
indicazioni utili per il riconoscimento del sito attivo o di strutture
indispensabili all'attività della molecola o dirette ad una certa
funzione.
Alcune Applicazioni
Studiare le variazioni
conformazionali provocate
dall’interazione della proteina
con uno o più ligandi.
Attivazione o inattivazione di
sezione necessaria a
compiere la propria funzione
biologica
Alcune applicazioni
Applicazioni farmacologiche: viene
fornita un’indicazione specifica, o
quanto meno restrittiva, della
struttura opportuna in funzione del
bersaglio del farmaco.
In questo campo, la costruzione di
strutture calibrate permette di
ridurre la ricerca ad un ristretto
raggio d’azione.
Alcune applicazioni
Comprendere il processo di folding delle proteine, ovvero il
meccanismo di ripiegamento con cui esse raggiungono la
conformazione nativa biologicamente attiva.
Protein folding
A dispetto della loro complessità, la maggior parte delle
proteine reali si ripiegano (“foldano”) ad un unico stato nativo
mediante un “rapido” e “sicuro” processo di folding
A causa di ciò, in genere si dice che lo stato nativo di una
proteina è codificato nella sua sequenza primaria.
Capire come ciò sia possibile è un problema
multidisciplinare
(coinvolge dati biologici, biochimici, fisici, e richiede tecniche
informatiche, statistiche, biotecnologiche) e multiscala (il processo di
folding coinvolge movimenti strutturali che coprono scale temporali
molto diverse).
Lo stato nativo non è lo stato in cui la proteina nasce!
Obiettivi principali
1) L’analisi e la comprensione dei fenomeni fisici essenziali alla
base del folding:
questo potrebbe permettere, in principio, la costruzione, su base
chimico-fisica, di efficienti algoritmi per la predizione di strutture
2) La predizione dello stato nativo a partire dalla sequenza degli
aminoacidi di una proteina:
protein modeling
Meccanica molecolare (MM)
alias dinamica molecolare (MD)
Le simulazioni MD generano informazioni a livello
microscopico (posizioni, velocità degli atomi). Per
convertire queste informazioni in osservabili
macroscopiche (pressione, energia, capacità
termica, ecc.) c'è bisogno di
Termodinamica + Meccanica Statistica
Meccanica molecolare
o dinamica molecolare (MD)
Permette di simulare al computer le strutture ed alcune
proprietà dinamiche e termodinamiche dei materiali.
Applicabile solo a molecole/solidi/liquidi nello stato
elettronico fondamentale.
Descrive l’evoluzione temporale dei sistemi complessi
secondo la legge di Newton:
Fj = mj aj
Inizialmente la MM si applica in vacuo ma oggigiorno è
possibile trattare anche gli effetti del solvente.
20
Altri campi di applicazione della MD
Si può utilizzare per simulare al computer il moto degli
atomi e comprendere meccanismi intimi di
•piccole molecole, sia organiche che inorganiche
•reazioni chimiche
•transizioni di fase
•solidi ad altissime pressioni, fratture
•liquidi e miscugli di liquidi (es. liquidi ionici)
•liquidi in geometrie confinate
•lubrificazione
•....
Assunzioni fondamentali della MD
•
Nucleo ed elettroni sono «ammassati» in una sola particella, che
rappresenta l'atomo j-esimo nel punto rj.
•
L'energia si conserva a livello microscopico. Quindi le forze
Fj = –∇jV(r1, r2, r3, ...)
•
•
•
sono scrivibili come i gradienti di un'energia potenziale totale V
Le interazioni tra gli atomi che contribuiscono all'energia
potenziale sono parametrizzate da forme analitiche più o meno
semplici, come potenziali di molle o coulombiani o altri potenziali
classici (esempi più tardi). Questa è un'approssimazione poco
controllata!
L'equazione del moto si integra con algoritmi dove il tempo è
“discretizzato” a passettini.
Oppure le forze Fj si possono usare per cercare la
configurazione piu stabile (minimo dell'energia V)
Energia
Le interazioni determinano la distribuzione spaziale degli atomi e le
energie relative che rendono più o meno stabili determinate
configurazioni. Le forze sono appunto date da − i gradienti di V
Energia =
energia delle interazioni di legame
+
energia delle interazioni di non legame
Force field:
insieme delle equazioni (e
dei
parametri)
che
descrivono come varia
l'energia quando gli atomi
si spostano.
Energia di legame
Esempio di parametrizzazione
del campo di forze:
Energia di allungamento
(stretching) dei legami
+
Energia di piegamento
(bending) degli angoli tra
coppie di legami
+
Energia di torsione degli
angoli diedri
bending
stretching
torsion
Termini di legame: stretching
Energia di stretching: potenziale armonico (molle-legame)
V stretch = ∑
bonds
ks
2
r −r 0
ks = costante elastica,
r = |rj – rj'| = lunghezza istantanea del legame,
r0 = distanza di equilibrio
2
Termini di legame: bending
Energia di bending: potenziale armonico
V bend =
∑
angles
kθ
2
2
θ−θ 0
kθ = rigidità della molla-angolo,
θ = arccos((rj – rj')·(rj – rj”)/(|rj – rj'| |rj – rj”|)) = angolo
istantaneo tra i 2 legami,
θ0 = angolo di equilibrio
Termini di legame: torsione
Energia torsionale:
V t=
∑
torsions
kφ
2
φ −φ0
2
oppure
V t=
∑
torsions
A[1−cos n φ−φ0 ]
termine armonico (per torsioni rigide di piccola ampiezza)
+
termine sinusoidale (per torsioni “soffici” a 360°): modella la
presenza di barriere steriche attraversabili;
A = ampiezza curva,
n = tipo di simmetria e periodicità,
ϕ0 = angolo torsionale d'equilibrio
Interazioni a lunga distanza
•
Atomi non legati covalentemente, ad es. atomi della
stessa molecola ma separati tra loro da almeno due
legami covalenti, oppure atomi di molecole diverse
•
In buona approssimazione l'energia di “non-legame”
dipende dalle distanze reciproche tra tutte le coppie di
atomi non legati
•
Esempi principali di interazioni di non-legame: le forze
di Van der Waals e quelle elettrostatiche
•
Sono forze “a lungo raggio”: energie e forze decadono
con leggi di potenza a grande distanza tra le coppie di
atomi
Interazioni di Van der Waals
Una forza attrattiva (a lunga
distanza E ∝ –r-6), dovuta a
fluttuazioni nella distribuzione di
carica delle nuvole elettroniche.
A piccola distanza questa attrazione
di VdW (che divergerebbe a –∞,
facendo collassare la simulazione) è
bilanciata,
e
dominata
dalla
repulsione dovuta al sovrapporsi
delle nubi elettroniche (piccola
distanza E ∝ +r-12). Repulsione e
attrazione vanno a zero a distanza
interatomica infinita.
Bilanciamento ad una distanza
intermedia ottimale r*.
r*
Le interazioni di Van der Waals
e gli effetti di repulsione
vengono spesso modellizzati
usando il potenziale di
Lennard-Jones
Interazioni elettrostatiche
Potenziale di Coulomb: rappresenta l’interazione elettrostatica tra
una coppia di atomi carichi
1
V Coulomb r 1 , r 2 ,... , r N =
4 0 r
qj qj'
∑ ∣r −r ∣
j j '
j
j'
Interazioni di questo tipo dipendono
• dalla carica atomica qj presente sugli atomi non-legati
• dalla distanza tra atomi non-legati
• dalla natura del dielettrico interposto (può essere sia il solvente
che una parte della molecola stessa). Di solito si assegna un
valore costante allo schermo εr del dielettrico (tra 1 e 5); si
utilizzano valori linearmente dipendenti dalla distanza per
simulare effetti dovuti al solvente
Che ce ne facciamo delle Fj?
Cerchiamo il minimo dell'energia
configurazione più stabile.
potenziale,
cioè la
Oppure integriamo l'equazione di Newton e vediamo come la
materia si muove.
Minimizzazione energia libera:
guida il folding delle proteine
Ipotesi termodinamica di Anfinsen
Le proteine funzionali assumono spontaneamente
un'unica struttura tridimensionale, determinata dalla loro
struttura primaria (sequenza)
Questa struttura nativa è il minimo stabile dell'energia
libera ed un percorso ragionevolmente “liscio” porta dallo
stato unfolded alla struttura nativa.
Lo stato nativo è il minimo assoluto dell’energia libera F
della proteina.
Ricerca minimo energetico
Conformazione energeticamente preferita
minimo globale dell'energia potenziale
equilibrio stabile
stato più popolato a bassa T
... ma...
F=U–TS
L'energia libera F non coincide con l'energia U
È necessario tenere conto anche di termini entropici
Nei calcoli talvolta si usano delle energie potenziali
“efficaci” che tengono conto anche dell'entropia
Algoritmi di minimizzazione
Fanno discendere l'energia verso il minimo più vicino
Algoritmi principali:
lento ma robusto, senza complicazioni
• Simplex:
strutture di partenza rozze e ad elevata energia
descent: veloce, lavora bene
• Steepest
minimo quando il gradiente è grande
dà
lontano dal
lavora bene solo vicino al minimo;
• Newton-Raphson:
meno steps per convergere
⇒ meglio usare diversi minimizzatori a turno
Un rischio tipico della minimizzazione
I minimi locali: partendo da configurazioni a casaccio la minimizzazione
del potenziale rischia di intrappolarsi in configurazioni molto diverse dal
vero minimo
minimo
assoluto
configurazione
iniziale rischiosa
Un rischio reale: alcune malattie
(Alzheimer, BSE, Parkinson) sono
legate a misfolding di proteine!
Violazione dell'ergodicità: la proteina non
esplora tutto lo spazio delle configurazioni
Dinamica molecolare (MD)
Permette lo studio di processi dinamici complessi che
avvengono nei sistemi biologici. Può anche tornare utile a
trovare minimi globali (simulated annealing). Descrive allo
stesso tempo sia le vibrazioni locali che le transizioni
conformazionali, ad esempio:
stabilità delle proteine
variazioni conformazionali
folding proteico
trasporto ionico
Obiettivo della DM
Calcolare la traiettoria di un sistema molecolare
cioè
la configurazione molecolare in funzione del tempo,
cioè
come variano nel tempo le posizioni (e dunque anche le
velocità e le accelerazioni) di tutti gli atomi della/e molecola/e.
In realtà si parte proprio dalle accelerazioni:
a j = Fj / m j
Traiettoria
La traiettoria è generata da integrazioni simultanee dell’equazione del
moto di Newton
per tutti gli atomi del sistema,
a =F /m
j
j
j
nota la forza Fj esercitata dal sistema su ciascun atomo.
La forza è uguale alla derivata dell'energia potenziale:
Fj = – ∂V/∂rj
quindi
d2rj /dt2 = – mj-1 ∂V/∂rj
che collega la derivata dell'energia potenziale alle variazioni di
posizione in funzione del tempo
Ingredienti
•
le posizioni iniziali rj degli atomi (coordinate atomiche):
si ricavano ad es. da strutture sperimentali (cristallografia
raggi X, NMR ecc.) o ottenute con modeling;
•
le velocità iniziali vj spesso si ottengono dalla
distribuzione delle velocità caratteristica di una data
temperatura;
•
le accelerazioni aj lungo tutta la traiettoria sono
determinate dalle derivate dell'energia potenziale totale V
Distribuzione iniziale di velocità vi
Riscaldamento lento alla temperatura di simulazione
Riequilibrazione dell’energia tra gli atomi
Temperatura di simulazione desiderata
Lunga integrazione → energia libera del sistema
Algoritmi di integrazione
Non c'è soluzione analitica all'equazione del moto, data la
sua forma complessa (l'energia potenziale è funzione delle
3N coordinate atomiche di tutti gli atomi del sistema) ⇒
soluzione numerica ⇒ algoritmi numerici (es. velocity-Verlet e
Runge-Kutta).
Caratteristiche comuni fondamentali degli algoritmi di
integrazione:
●
deve valere la conservazione dell'energia e del momento
●
devono essere computazionalmente efficienti
●
●
devono permettere un tempo di integrazione il più lungo
possibile
si possono fare simulazioni a energia fissata (ensemble
microcanonico) oppure a temperatura fissata (ensemble
canonico)
Accuratezza metodo
L'accuratezza della simulazione è limitata da
●
qualità del force-field (= capacità di riprodurre i risultati
sperimentali)
●
simulazione dell'ambiente circostante
●
scelta della configurazione di partenza
●
lunghezza del periodo di tempo di equilibrazione
●
lunghezza del tempo di simulazione
●
(alcuni ns)
eventuale importanza di effetti quantistici (mecc. classica →
no livelli vibrazionali discreti, no spettroscopia!)
Un “esperimento” al computer
Evoluzione di proprietà locali (nucleasi dello stafilococco)
Un “esperimento” al computer
2.5 nm
Carbonio
Idrogeno
Azoto
Fluoro
Una simulazione di liquidi ionici: [bmim]+ [Ntf2]− a 300 K
Un “esperimento” al computer
Dynameomics: un pacchetto per analizzare le traiettorie della MD
Un “esperimento” al computer
(movie)
Simulazione MD di un peptide in solvente acquoso
Un “esperimento” al computer
(movie)
Simulazione “parallel tempering” o “replica exchange” MD del folding:
AK-peptide (sequenza AAAAKAAAAKAAAAKAAAAY) in solvente
acquoso (non mostrato ma simulato).
A=Alanina, K=Lisina, Y=Tirosina
Un “esperimento” al computer
(movie)
Simulazione replica-exchange MD del folding:
trp-cage miniprotein (sequenza NLYIQWLKDGGPSSGRPPPS) in
solvente acquoso (non mostrato ma simulato). È mostrata una
singola replica (di 40) che arriva alla struttura nativa ripiegata in circa
30-40 ns di tempo di simulazione