MOLECULAR DYNAMICS
Vediamo quali sono gli “ingredienti” per effettuare una dinamica molecolare:
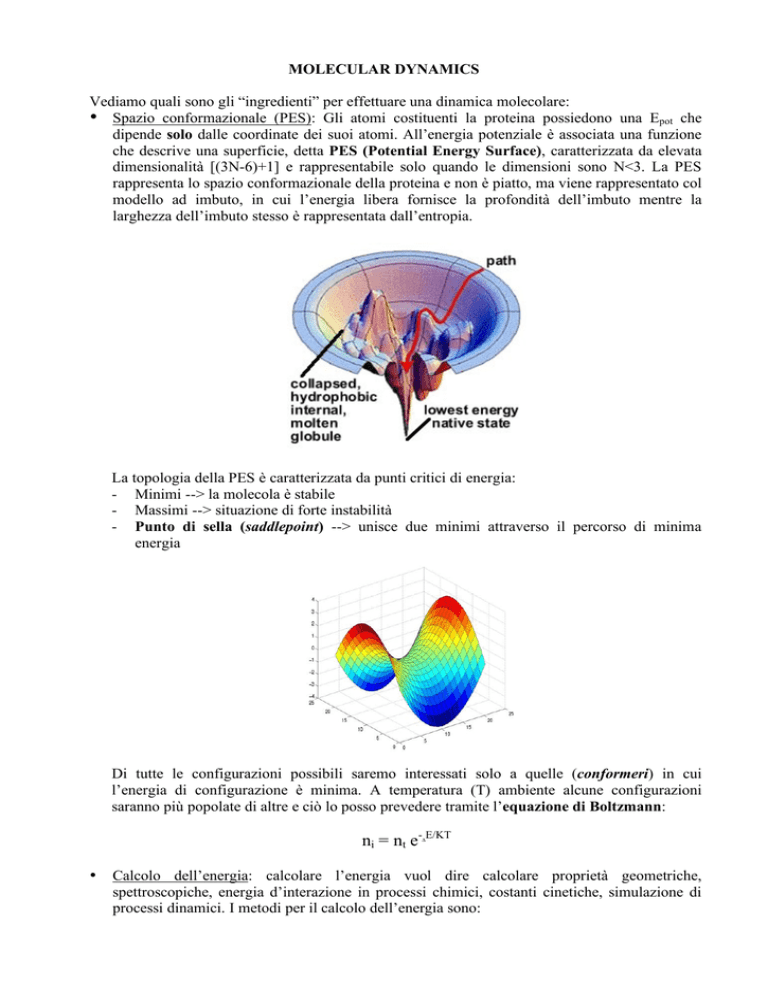
• Spazio conformazionale (PES): Gli atomi costituenti la proteina possiedono una Epot che
dipende solo dalle coordinate dei suoi atomi. All’energia potenziale è associata una funzione
che descrive una superficie, detta PES (Potential Energy Surface), caratterizzata da elevata
dimensionalità [(3N-6)+1] e rappresentabile solo quando le dimensioni sono N<3. La PES
rappresenta lo spazio conformazionale della proteina e non è piatto, ma viene rappresentato col
modello ad imbuto, in cui l’energia libera fornisce la profondità dell’imbuto mentre la
larghezza dell’imbuto stesso è rappresentata dall’entropia.
La topologia della PES è caratterizzata da punti critici di energia:
- Minimi --> la molecola è stabile
- Massimi --> situazione di forte instabilità
- Punto di sella (saddlepoint) --> unisce due minimi attraverso il percorso di minima
energia
Di tutte le configurazioni possibili saremo interessati solo a quelle (conformeri) in cui
l’energia di configurazione è minima. A temperatura (T) ambiente alcune configurazioni
saranno più popolate di altre e ciò lo posso prevedere tramite l’equazione di Boltzmann:
ni = nt e- E/KT
Δ
•
Calcolo dell’energia: calcolare l’energia vuol dire calcolare proprietà geometriche,
spettroscopiche, energia d’interazione in processi chimici, costanti cinetiche, simulazione di
processi dinamici. I metodi per il calcolo dell’energia sono:
-
Metodi quantomeccanici --> Utilizzano i principi della meccanica quantistica che tengono
conto di posizione e velocità di nuclei ed elettroni.
Metodi classici --> Utilizzano la teoria della meccanica classica in cui gli elettroni non
vengono considerati in modo esplicito, ma solo i nuclei. I metodi classici sono:
Meccanica molecolare : Cerca di descrivere le molecole tramite le leggi della fisica
classica. La meccanica molecolare trova la propria estrinsecazione nel force field,
cioè un potenziale che descrive le caratteristiche energetiche della molecola.
Applicando il campo di forze è possibile calcolare l’Epot di una data molecola che
possiede una certa conformazione considerando la somma di singoli contributi
energetici dati dal legame chimico covalente ed interazioni di non legame:
ETOT = ECOV + ENONCOV
Dove:
ECOV = ESTR + EBEND + ETORS
ESTR = ESTRETCHING = funzione per l’allungamento di un legame tra due atomi
EBEND = EBENDING = energia necessaria per deformare un angolo formato da tre atomi
ABC quando ci sia un legame tra A e B e tra B e C
ETORS = ETORSION = energia di torsione attorno ad un legame per formare un nuovo
angolo diedro.
ENONCOV = EEL + EVdW
EEL = energia elettrostatica, calcolata utilizzando il potenziale di Coulomb
EVdW = termine legato alle forze di Van der Waals, calcolato col potenziale di LennardJones.
Dinamica Molecolare: nella dinamica molecolare si adopera il force field al fine di
valutare il comportamento dinamico di una molecola, dunque si vede come si muovono
gli atomi in un determinato tempo. La proteina deve esplorare il bacino di attrazione
del minimo globale o di altri minimi. Se T≠ 0 si parla di energia cinetica e di energia
potenziale:
ETOT = EK + EPOT
Dove:
EK = Energia cinetica = ½ mv2 = 3/2 KBT
Fornendo energia alla molecola, questa si muoverà lungo la PES, descrivendo una
traiettoria. Considerando il sistema proteina:
abbiamo tante particelle che compongono il sistema
le particelle interagiscono e la forza che agisce su un atomo cambia ad ogni δt
perchè gli atomi si muovono e cambiano posizione.
Se le condizioni iniziali del moto a cui è sottoposta la particella sono note, possono
essere scritte le leggi del moto. Quindi ad un certo istante t si sa come calcolare l’EPOT,
ma nel t successivo la configurazione è un po’ cambiata perchè ci sono delle forze che
agiscono sull’atomo. La legge che governa il moto dei vari atomi è:
F = ma
Lo scopo è risolvere l’equazione del moto per ricavare la posizione r(t) e la velocità
v(t). Visto che ad ogni istante tutto cambia, non è possibile integrare l’equazione del
moto ma si segue l’evoluzione della traiettoria ad ogni δt. In che modo? La traiettoria è
continua ma si procede per intervalli discretizzati. Applicando la regola del trapezio,
più piccolo è il trapezio meglio approssimo l’integrale della curva. Quindi,
riassumendo, ad ogni passo vengono calcolati:
Forze come derivate di energia potenziale rispetto alle coordinate atomiche -->
Fi(t) = -∂EPOT/∂T
Aggiornamento delle velocità e delle coordinate atomiche mediante le forze
precedentemente ottenute.
Una dinamica molecolare può essere condotta in differenti ensembles. Con questo
termine si suole indicare l’idealizzazione di un gran numero di copie del sistema
considerate all’istante, ognuna delle quali rappresenta un possibile stato in cui il
sistema reale potrebbe essere. Nella molecola le particelle calde dovranno cedere
energia cinetica alle particelle fredde e allora compariranno delle forze: c’è un travaso
da una parte all’altra di energia cinetica e potenziale. Si può lavorare in:
NVE ensemble (microcanonical ensemble) --> E’ un sistema isolato in cui il
numero di particelle, velocità ed energia totale vengono conservati. NVE
corrisponde ad un processo adiabatico in cui non c’è scambio di calore.
Sfortunatamente, il microcanonical ensemble non corrisponde alle condizioni
alle quali avviene la maggior parte degli esperimenti. Infatti, se lavoro ad ETOT
costante, l’energia cinetica istantanea varia e cambia la T istantanea. Non ha
senso portare la proteina in condizioni di alta T; se per caso siamo andati in un
punto in cui le forze sono grandi e la PES s’impenna, le velocità aggiornate
saranno grandi e vado a finire in un regime ad alta T.
NVT ensemble (canonical ensemble) --> Abbandonata l’idea di lavorare ad
energia costante per i motivi esposti precedentemente, si decide di lavorare a
temperatura costante: in questo modo l’energia cinetica sarà costante ma varierà
l’energia potenziale, con la conseguenza che l’energia totale cambia. Questo è
l’approccio più fisiologico in quanto questo non è un sistema isolato; per
mantenere costante la T bisogna invocare un sistema di scambio di calore tra il
mio sistema e l’universo (bagno termostatico). Se l’aumento della T è molto
grande, il sistema impiegherà molto più tempo a ritermalizzarsi: ciò vuol dire
che la mia proteina rischia di stare per troppo tempo ad alta T. per mantenere
costante la T bisogna accoppiare il sistema proteina con un bagno termostatico
esterno, la cui T è circa costante ed è controllata da un termostato. L’ensemble
NVT è fondamentale nello studio di protein unfolding che richiede un controllo
preciso della T.
NPT ensemble --> In questo caso P e T vengono mantenuti costanti.
Una volta scelto l’ensemble opportuno, è necessario decidere se effettuare la
dinamica in solvente esplicito o implicito:
Solvente esplicito --> Vengono aggiunte molecole d’acqua al sistema
(solvation box) e la distanza che intercorre tra gli atomi della proteina e lo
spigolo della box dovrebbe essere compreso tra gli 8 e i 10 A. In caso di
proteine con cariche nette vengono aggiunti controioni per neutralizzare il
sistema.
Solvente implicito --> Consiste nell’inclusione di una costante elettrostatica
nel termine elettrostatico dell’energia potenziale (costante dielettrica efficiente).
Le molecole di solvente sono implicitamente considerate modificando
opportunamente le interazioni tra i nuclei come se essi subissero l'effetto
``medio'' cioe' statistico della presenza di un gran numero di molecole di
solvente. In questo caso il costo computazionale è notevolmente ridotto rispetto
alla trattazione con solvente esplicito.
A questo punto avviene una fase di minimizzazione energetica. Questa fase è
necessaria per eliminare quelli che vengono definiti come atom clashes. Solitamente si
effettuano alcuni cicli di steepest descent o steepest descent+conjugate gradients ed il
sistema viene portato alla temperatura desiderata. Dopo la minimizzazione avviene uno
step di equilibrazione del sistema, in cui si cerca di mantenere il sistema stesso alla
temperatura desiderata. In questa fase la temperatura viene costantemente monitorata e
se avvengono variazioni di un tot K (ad es. ±10K) rispetto alla T preimpostata, le
velocità vengono scalate per consentire la ritermalizzazione alla T voluta.
Successivamente alla fase di equilibrazione (della durata di circa 20ps) possono
prendere via i cicli produttivi (production runs). Dall’analisi della traiettoria (o dalle
traiettorie) ottenute da un esperimento di dinamica molecolare si ottengono importanti
informazioni:
RMSD: Come già visto precedentemente,rappresenta la vicinanza, in termini
strutturali, alla struttura nativa. Viene solitamente considerato l’RMSD dei C
Una distanza compresa tra 3 e 4A rispetto alla struttura iniziale è generalmente
usata come indicatore del raggiungimento del folding.
α
Cambiamento della SASA: il processo di folding è tipicamente accompagnato
da un decremento significativo della SASA (Solvent Accessible Surface Area).
La SASA dunque – o in alternativa il raggio di girazione – è un indicatore del
processo di folding.
Folding landscape: poichè il folding è un problema multidimensionale, le
analisi basate su coordinate di reazione monodimensionali offrono un quadro
incompleto del processo di folding. Le coordinate tipicamente adoperate sono
l’RMSD, il raggio di girazione e la percentuale di contatti nativi.
© Irene Righetto e Francesco Filippini, 2005-2017