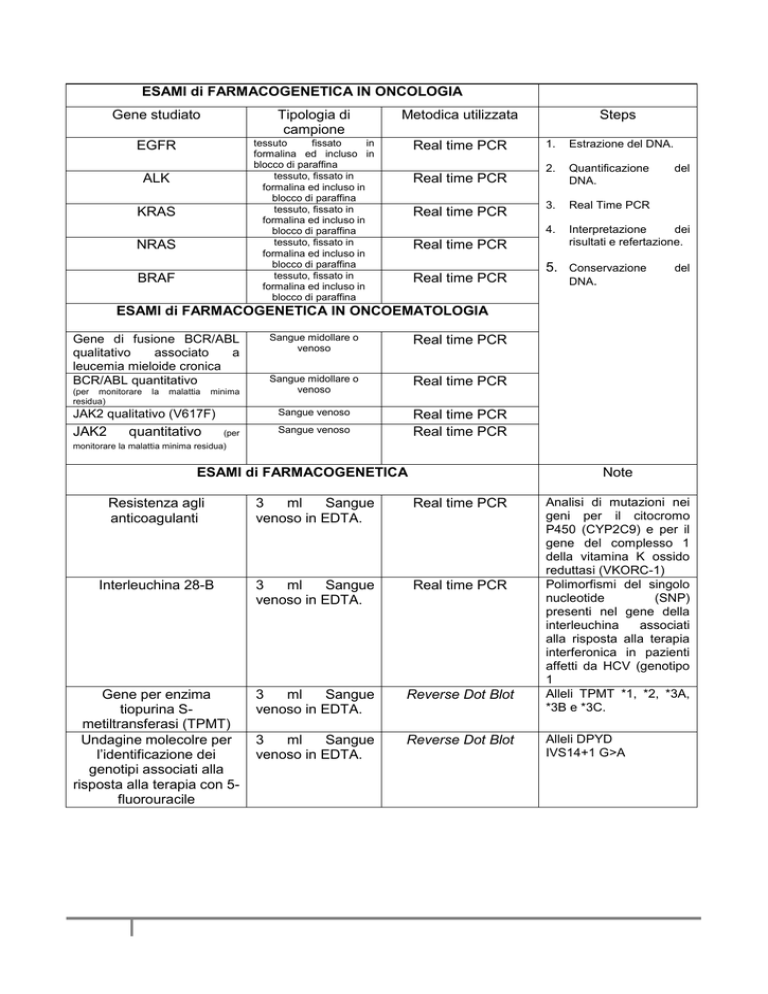
ESAMI di FARMACOGENETICA IN ONCOLOGIA
Gene studiato
Tipologia di
campione
Metodica utilizzata
EGFR
tessuto
fissato
in
formalina ed incluso in
blocco di paraffina
tessuto, fissato in
formalina ed incluso in
blocco di paraffina
tessuto, fissato in
formalina ed incluso in
blocco di paraffina
tessuto, fissato in
formalina ed incluso in
blocco di paraffina
tessuto, fissato in
formalina ed incluso in
blocco di paraffina
Real time PCR
ALK
KRAS
NRAS
BRAF
Real time PCR
Real time PCR
Real time PCR
Real time PCR
Steps
1.
Estrazione del DNA.
2.
Quantificazione
DNA.
3.
Real Time PCR
4.
Interpretazione
dei
risultati e refertazione.
5. Conservazione
del
del
DNA.
ESAMI di FARMACOGENETICA IN ONCOEMATOLOGIA
Sangue midollare o
venoso
Real time PCR
Sangue midollare o
venoso
Real time PCR
JAK2 qualitativo (V617F)
Sangue venoso
JAK2
Sangue venoso
Real time PCR
Real time PCR
Gene di fusione BCR/ABL
qualitativo
associato
a
leucemia mieloide cronica
BCR/ABL quantitativo
(per monitorare
residua)
la
malattia
minima
quantitativo
(per
monitorare la malattia minima residua)
ESAMI di FARMACOGENETICA
Note
Resistenza agli
anticoagulanti
3
ml
Sangue
venoso in EDTA.
Real time PCR
Interleuchina 28-B
3
ml
Sangue
venoso in EDTA.
Real time PCR
Gene per enzima
tiopurina Smetiltransferasi (TPMT)
Undagine molecolre per
l’identificazione dei
genotipi associati alla
risposta alla terapia con 5fluorouracile
3
ml
Sangue
venoso in EDTA.
Reverse Dot Blot
3
ml
Sangue
venoso in EDTA.
Reverse Dot Blot
Analisi di mutazioni nei
geni per il citocromo
P450 (CYP2C9) e per il
gene del complesso 1
della vitamina K ossido
reduttasi (VKORC-1)
Polimorfismi del singolo
nucleotide
(SNP)
presenti nel gene della
interleuchina
associati
alla risposta alla terapia
interferonica in pazienti
affetti da HCV (genotipo
1
Alleli TPMT *1, *2, *3A,
*3B e *3C.
Alleli DPYD
IVS14+1 G>A
ESAMI DI GENETICA MOLECOLARE
Gene studiato
Tipologia di campione
Metodica utilizzata
surrenalica
3 ml Sangue venoso in EDTA.
Analisi di mutazioni nel gene CYP21A2
definite mediante Reverse Dot Blot
HLA DQ2 e HLA DQ8
suscettibilità alla celiachia
3 ml Sangue venoso in EDTA.
Analisi di mutazioni definite mediante
Reverse Dot Blot
Iperplasia
congenita
ESAMI DI GENETICA MOLECOLARE
Gene studiato
Tipologia di campione
Disomia Uniparentale
(Sindromi di Prader-Willi,
Angelman, BeckwithWiedeman, Russell-Silver)
Analisi aneuploidie
cromosomi 13,18,21,X,Y
X-Fragile o Sindrome di
Martin-Bell (FRAXA/FRAXE)
Metodica utilizzata
3 ml Sangue venoso in EDTA.
15 ml di Liquido amniotico.
20 mg di Villi coriali.
Studio di marcatori genetici STR
mediante
PCR
fluorescente
e
genotipizzazione
3 ml Sangue venoso in EDTA.
15 ml di Liquido amniotico.
20 mg di Villi coriali.
CGH-ARRAY
3 ml Sangue venoso in EDTA.
15 ml di Liquido amniotico.
20 mg di Villi coriali.
Valutazione dell’espansione patologica
di triplette nucleotidiche associate alla
sindrome di Martin-Bell.
DIAGNOSTICA PRENATALE
Analisi
liquido amniotico.
Prelievo mediante
amniocentesi.
Campione
Note Tecniche
Tipo
INDICAZIONI
20 ml di Liquido
amniotico
-
Studio molecolare dei geni beta globinici.
-
Studio molecolare del gene CFTR
epoca di gestazione:
16-18 settimane.
Villi coriali: prelievo
mediante villocentesi
epoca di gestazione:
11-13 settimane.
Villi coriali
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali.
INDAGINI CITOGENETICHE/CITOGENOMICHE
Analisi
Campione
Note Tecniche
Tipo
Cariotipo da sangue
venoso
5 ml Sangue venoso in
eparina
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali.
Cariotipo da midollo
osseo
5 ml di sangue midollare
in eparina
Identificazione di riarrangiamenti cromosomici
correlati all’estrinsecazione di alcune patologie
oncoematologiche
Cariotipo da liquido
amniotico
20 ml di Liquido
amniotico
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali.
Cariotipo da materiale
abortivo
Materiale abortivo
20 mg Villi coriali
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali.
Cariotipo su villi coriali
20 mg Villi coriali
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali.
(metodo diretto +
coltura)
FISH con sonde
painting
FISH con sonde a
sequenza unica
Array-CGH 180k
(Array-based
Comparative Genomic
Hybridization)
10 ml sangue in eparina
5 ml Liquido amniotico
20 mg Villi coriali
20 mg Materiale abortivo
Caratterizzazione di riarrangiamenti cromosomici
strutturali.
10 ml sangue in eparina
5 ml Liquido amniotico
20 mg Villi coriali
20 mg Materiale abortivo
Identificazione delle principali sindromi determinate
da microdelezioni e/o micro duplicazioni
cromosomiche
5 ml Sangue in eparina
5 ml Sangue in EDTA
Analisi dello sbilanciamento del numero di copie di
sequenze genomiche.
Caratterizzazione di riarrangiamenti cromosomici
strutturali.
(In fase di acquisizione)
Analisi
Campione
Note Tecniche
Tipo
Diagnosi prenatale
invasiva precoce
mediante utilizzo della
metodica CGH-array 44k
5 ml di Liquido amniotico
Villi coriali
Identificazione di sindromi legate ad aberrazioni
cromosomiche numeriche e strutturali
submicroscopiche (<5Mb)