SCUOLA DI DOTTORATO DI RICERCA – XIII Ciclo
FACOLTA’ DI MEDICINA E CHIRURGIA
Curriculum Neuroscienze
Dipartimento di Scienze Cliniche Specialistiche ed Odontostomatologiche
Sezione di Scienze della Salute della Donna
ABORTIVITA’ SPONTANEA RICORRENTE: STUDIO DI
ESPRESSIONE ENDOMETRIALE DELLE CITOCHINE
INFIAMMATORIE
Relatore: Chiar.mo
Prof. Andrea Ciavattini
Dottoranda:
Dott.ssa Beatrice Landi
Correlatore: Chiar.ma
Prof.ssa Franca Saccucci
Anno Accademico 2014-2015
!
Al Prof. Andrea L. Tranquilli
"! #$%&'()*+'$,----------------------------------------------------------------------------------------------------.!
"-"! /0'&%+1+%2!34'$%5$,5!&+6'&&,$%,------------------------------------------------------------------------------------------.!
"-7! 8+%'69+$,!+$:+5;;5%'&+,!,!50'&%+1+%2!34'$%5$,5 -----------------------------------------------------------<!
"-.! =$(';,%&+'!,!50'&%+1+%2!34'$%5$,5 --------------------------------------------------------------------------------- ">!
7! ?4,&+;,$%5*+'$, ----------------------------------------------------------------------------------------- 7"!
7-"! @0+,%%+1'!(,AA'!3%)(+' ----------------------------------------------------------------------------------------------------------- 7"!
7-7! B5*+,$%+!,!;,%'(+-------------------------------------------------------------------------------------------------------------------- 7"!
7-.! C+3)A%5%+ ------------------------------------------------------------------------------------------------------------------------------------- >>!
7-D! E+36)33+'$,------------------------------------------------------------------------------------------------------------------------------- <F!
7->! 8'$6A)3+'$+ ------------------------------------------------------------------------------------------------------------------------------- GH!
I+0A+'J&5:+5------------------------------------------------------------------------------------------------------- "KK!
2
1 Introduzione
1.1 Abortività spontanea ricorrente
Si definisce aborto spontaneo l’evento clinico che porta all’interruzione
spontanea di una gravidanza clinicamente riconosciuta, con un feto che pesi
meno di 500 grammi oppure qualora il peso sia sconosciuto, con un feto che
misuri meno di 25 cm (lunghezza vertice-tallone) oppure che la gravidanza
sia durata meno di 22 settimane complete [1,2].
L’abortività spontanea ricorrente è definita come il verificarsi di due o più
aborti spontanei consecutivi [3,4]. Non rientrano nel computo degli aborti per
la definizione di aborto ricorrente né gli aborti procurati né quelli derivanti da
gravidanze iniziate con tecniche di fecondazione assistita (es.FIVET o ICSI).
Sono valutabili ai fini della definizione di aborto ricorrente solo gli aborti di
gravidanze clinicamente riconosciute. Ovviamente quindi non sono valutabili
come aborti le morti embrionali precocissime che avvengono prima
dell’annidamento e sono pertanto asintomatiche.
L’abortività spontanea ricorrente è un problema clinico comune nell’età
riproduttiva che si verifica in circa il 1-3% delle gravidanze [5,6].
Nella specie umana si calcola che su 100 concepimenti 16 si blocchino allo
stadio zigote, l5 si perdano ulteriormente prima dell’impianto nell’utero. Dei
rimanenti, 25 muoiono dopo l’impianto entro due settimane di età
concezionale, altri 10 muoiono tra 3 e 6 settimane e 3 muoiono tra 7 e 20
settimane di età concezionale. Dei feti che superano le 20 settimane di età
concezionale, uno muore prima del parto. Quindi si può ritenere che il 103
15% delle gravidanze clinicamente riconosciute esiti in aborto spontaneo, in
genere prima della 14° settimana gestazionale, ma che gli aborti in totale,
compresi quelli preclinici, si attestino attorno al 50%.
Una gravidanza esitata in aborto spontaneo predispone ad un elevato rischio
di sviluppare ulteriori aborti. Il rischio di abortività in una successiva
gravidanza cresce all’aumentare degli aborti precedenti: 24% dopo un aborto,
30% dopo due, 35% dopo tre aborti fino a circa 40% dopo quattro aborti
spontanei consecutivi [7].
L’aborto spontaneo è la più comune complicanza della gravidanza ed implica
un coinvolgimento della donna e della coppia con risvolti di ordine
psicologico. L’importanza epidemiologica della poliabortività risiede quindi
anche nell’alta morbidità psicologica [8].
Eziopatogenesi:
L`incidenza dell’abortività spontanea ricorrente osservata è molto più alta
(l%) di quella che ci si aspetterebbe se fosse determinata solo dal caso
(0,34%) [9,10].
Sebbene cause genetiche, ormonali ed anatomiche siano state implicate
nell’eziologia dell’abortività ricorrente, più del 60 per cento dei casi
rimangono tuttora inspiegati [11].
Le cause note riguardano fattori genetici, cromosomici, uterini, endocrini,
metabolici, infettivi, trombofilici, immunologici ed ambientali [12-15].
Anomalie genetiche e cromosomiche (3-6%): Le anomalie cromosomiche
rappresentano la causa più frequente di aborto spontaneo sporadico e si
ritrovano nel 50-70% degli aborti spontanei del primo trimestre e nel 5-10%
di tutte le gravidanze.
4
Diverso è il caso dell’aborto ricorrente; in questa condizione, un’anomalia
cromosomica, di cui uno dei partner è portatore (solitamente una
traslocazione bilanciata) è la sola causa indiscussa di abortività ricorrente
[16]. Per la diagnosi la sola anamnesi familiare non è sufficiente; occorre
effettuare uno studio più approfondito dei partners mediante analisi del
cariotipo su sangue periferico [3,17].
Alterazioni anatomiche (10-15%): Le anomalie possono essere congenite
(difetti di fusione dei dotti mulleriani, come utero unicorne, bicorne, didelfo,
setto ecc, anomalie vascolari dell’arteria uterina, esposizione a DES) o
acquisite (sinechie uterine per pregressi raschiamenti o endometriti,
leiomiomi, endometriosi/adenomiosi, incompetenza cervicale iatrogena o da
trauma). La diagnosi è principalmente strumentale.
Disturbi ormonali (10-20%): diabete mellito, disfunzioni tiroidee [18],
insufficienza della fase luteale, PCOS (sindrome dell’ovaio policistico) [1921].
Processi infettivi (5%): possono essere associati all’aborto sporadico, non ci
sono evidenze per quello ricorrente.
Agenti fisici e chimici (<1%)
Cause ematologiche (10-20%), in particolare difetti trombofilici. Studi
retrospettivi hanno riportato un aumento della prevalenza di abortività ripetuta
in pazienti con alterazione dei livelli sierici dei normali inibitori della
coagulazione [22-24], quali: antitrombina III, proteina C e proteina S,
iperomocisteinemia, resistenza alla proteina C attivata (APCR; generalmente
congenita, secondaria alla mutazione del gene del fattore V di Leiden)
[25,26].
Problemi immunologici (30-40%)
5
Il fattore immunitario sembra essere responsabile di una buona percentuale di
casi inspiegati, alcuni Autori riportano una prevalenza dell’80%, altri la
ritengono sovrastimata correggendo questa percentuale ad un 30-40%. Si
possono distinguere due condizioni:
- l’aborto autoimmune (la presenza di anticorpi antifosfolipidi è la sola causa
immunologica umorale scientificamente documentata di abortività ricorrente)
- l’aborto alloimmune, quando non si instraurano i meccanismi immunologici
di
protezione
della
gravidanza
[27,28].
Sono
numerosi
gli
studi
sull’incompatibilità HLA, sulle cellule NK [29-33] e sull’assetto di citochine
ed il loro polimorfismo [34], ma non esistono attualmente evidenze
sull’utilizzo di tali test diagnostici nella pratica clinica [35].
1.2 Citochine infiammatorie e abortività spontanea
1.2.1 Caratteristiche generali delle citochine
La superfamiglia proteica delle citochine è parte integrante del sistema di
comunicazione intercellulare (the signaling network) ed è essenziale nella
generazione e regolazione del sistema immunitario. Recentemente si sono
ottenuti notevoli progressi nell’interpretare come il sistema immunitario
comunica, ed è regolato, grazie alle citochine ed alle chemochine o citochine
chemiotattiche. L’interazione di questi segnali biologici permette lo
svolgimento di numerose attività, come regolare la crescita e lo sviluppo,
l’ematopoiesi,
il
reclutamento
linfocitario,
la
sottopopolazioni dei linfociti T e l’infiammazione.
6
differenziazione
delle
Una cellula immunitaria all’interno di un dato microambiente può rispondere
ai segnali ricevuti grazie ai propri recettori con un suo linguaggio proteico,
mediato appunto dalle citochine [36].
Le citochine si comportano pertanto da “mediatori” intercellulari o meglio da
“modificatori della risposta biologica”e le loro azioni possono influenzare la
cellula stessa (effetto autocrino) o altre cellule dell’organismo, vicine (effetto
paracrino) o lontane (effetto endocrino).
Le citochine sono piccoli fattori solubili con funzioni pleiotropiche e sono
prodotte da numerosi tipi di cellule come parte integrante di un pattern di
espressione genica che può influenzare e regolare il sistema immune.
Il termine “citochine” fu proposto da Cohen et al. nel 1974 [37] al posto di
“linfochine”, termine coniato alla fine degli anni ’60 per indicare le proteine
solubili con effetti immunologici derivate dai linfociti [38], contrapposto a
“monochine” per indicare quelle prodotte dai macrofagi. Dato che la
designazione “linfochine” lasciava intendere in modo fuorviante che i linfociti
potessero essere le uniche cellule in grado di secernere tali proteine, il termine
citochine divenne rapidamente il preferito. Seguendo tale impostazione
generica, al “Second International Lymphokine Workshop” del 1979 fu
proposto il sistema di nomenclatura “interleukin” (IL) per semplificare la
crescente lista di citochine, contrassegnandole con la sigla IL seguita da un
numero (progressivo man mano che vengono scoperte). Ironicamente però
questo sistema parzialmente adottato ha continuato a generare confusione in
quanto non tutte le interleuchine, che sono almeno 23, effettivamente
agiscono solamente tra linfociti. Quindi la moderna nomenclatura delle
citochine è un insieme di termini largamente accettati, anche se un
po’fuorvianti, denominazioni secondo il sistema interleuchine ed altre
proteine ancora note con i loro nomi originali [36].
7
Le citochine sono una famiglia di molecole molto eterogenee in cui si
possono identificare però caratteristiche comuni:
o
Sono molecole prodotte essenzialmente durante la fase di attivazione e
la fase effettrice sia dell’immunità innata che di quella specifica.
o
Sono proteine glicosilate, con p.m. compreso tra 10.000 e 60.000 Da.
o
Al pari di altri ormoni peptidici, esercitano la loro attività legandosi a
specifici recettori presenti sulla superficie delle cellule bersaglio. La cellula
bersaglio può essere la stessa produttrice (attività autocrina), una cellula
vicina (attività paracrina) o, come nel caso di altri ormoni, una cellula lontana
(attività endocrina). Le citochine possiedono quindi azione sia locale che
sistemica.
o
I recettori per le citochine hanno un’affinità molto elevata (Kd = 10-10
- 10-12), pertanto esse sono attive a bassissime concentrazioni.
o
L’espressione dei recettori è regolata da specifici segnali esterni alla
cellula, in genere costituiti dalla stessa o da altre citochine, o nel caso dei
linfociti, dal riconoscimento dell’antigene.
o
La secrezione delle citochine è un fenomeno breve ed autolimitato: le
citochine non sono accumulate in granuli, ma prodotte de novo in seguito allo
stimolo.
o
Numerose citochine sono prodotte da diversi tipi cellulari (si tratta
soprattutto di monociti, macrofagi e linfociti T, ma anche cellule endoteliali e
alcune cellule epiteliali possono produrre citochine) e ciascuna agisce su tipi
cellulari diversi (“pleiotropismo”).
o
L’attività delle citochine è spesso ridondante, cioè diverse citochine
esercitano lo stesso effetto.
8
o
Le citochine influenzano spesso la sintesi di altre citochine e/o la loro
attività; possono interagire tra loro antagonizzandosi o cooperando in modo
additivo o sinergico, potenziandosi a vicenda.
o
La risposta delle cellule bersaglio alla maggior parte delle citochine
consiste
in
modificazioni
dell’espressione
genica,
con
conseguente
acquisizione di nuove funzioni.
Esistono 2 eccezioni a questa regola: le chemochine, che promuovono una
rapida migrazione delle cellule senza bisogno dell’espressione di nuovi geni,
ed il fattore di necrosi tumorale (TNF) che induce morte della cellula
bersaglio, anche in questo caso senza bisogno dell’espressione di geni sino ad
allora silenti.
1.2.2 Classificazione funzionale delle citochine
Sono state identificate centinaia di citochine. Possono essere classificate
secondo la loro funzione principale in 3 grandi gruppi:
1.
Mediatori dell’immunità innata: dette anche citochine infiammatorie.
Sono prodotte principalmente dai fagociti mononucleati per potenziare o
inibire le reazioni infiammatorie. Gli stimoli per la produzione di tali
citochine sono prodotti batterici, come l’LPS e i peptidoglicani, prodotti
virali, come l’RNA a doppia elica, o citochine di derivazione T, come l’ IFN
gamma. Comprendono: TNF alfa, IL-1, IL-6, IL-10, IL-12, IL-15, IL-18,
chemochine, IFN alfa e IFN beta.
2.
Mediatori dell’immunità specifica: dette anche citochine immunitarie.
Sono prodotte soprattutto dai linfociti T in risposta ad un riconoscimento
antigenico specifico. Alcune svolgono funzione regolatrice sulla crescita e
differenziazione di varie popolazioni linfocitarie, mentre altre reclutano,
9
attivano e controllano cellule effettrici specializzate, come fagociti
mononucleati, neutrofili ed eosinofili. Comprendono: IL-2, IL-4, IL-5, (IL-6),
IL-13, IL-16, IL-17, LT, TGF beta e IFN gamma.
3.
Fattori di crescita del sistema ematopoietico. Prodotte da cellule
stromali del midollo osseo, leucociti ed altri tipi cellulari, stimolano la
crescita e la differenziazione dei leucociti immaturi. Comprendono: IL-3, IL-7
ed altri fattori a classificazione incerta, come SCF, GM-CSF, M-CSF e GCSF.
Il profilo di secrezione di caratteristiche citochine e chemochine ha permesso
di classificare le cellule T helper (Th) CD4+ in 2 grandi sottopopolazioni:
Th1 e Th2.
Le cellule Th1 secernono soprattutto IL-2, interferone gamma (IFN-!) e
tumor necrosis factor-" (TNF-") ed esprimono tra i recettori per chemochine
soprattutto CXCR3, mentre le cellule Th2 producono soprattutto IL-4, IL-5,
IL-6, IL-10 e IL-13 ed esprimono i recettori per chemochine CCR3 e CCR4.
L’IL-12 è il principale induttore delle cellule Th1, mentre l’IL-4 rappresenta il
principale stimolo per la differenziazione in senso Th2.
Le cellule Th1 supportano l’immunità cellulo-mediata e quindi promuovono
l’infiammazione, la citotossicità e l’ipersensibilità di tipo ritardato (DTH),
attraverso soprattutto l’attivazione di macrofagi e neutrofili.
Le cellule Th2 supportano invece l’immunità umorale e inibiscono le azioni
infiammatorie delle cellule Th1, sopprimendo soprattutto l’attivazione
macrofagica; sono responsabili inoltre dell’attivazione dei eosinofili e della
degranulazione dei mastociti [39-43].
10
Tuttavia le singole cellule T possono esprimere le più diverse combinazioni di
citochine ed esistono probabilmente numerosissime sottopopolazioni, con un
profilo di espressione di citochine estremamente eterogeneo).
1.2.3 La “teoria” immunologica nell’abortività spontanea ricorrente
La natura immunologica di aborti spontanei ricorrenti altrimenti inspiegati ha
suscitato notevole interesse e controversie.
Fin dal 1953, quando Medawar evidenziò che la sopravvivenza del prodotto
allogenico del concepimento dei mammiferi contraddice le leggi del trapianto
di tessuto [44], il feto è considerato un semi-trapianto dato il contributo
genetico da parte di ciascun genitore con un conseguente possibile disturbo
sui fattori alloimmuni nell’ambiente placentare. Pertanto il rigetto
immunologico
dell’embrione/feto
potrebbe
essere
una
conseguenza
dell’insuccesso nei meccanismi che normalmente salvaguardano il sistema
immunitario materno da parte dell’attacco degli antigeni paterni espressi nel
prodotto del concepimento.
Il feto offre alla propria madre un doppio stimolo immunogeno: la placenta
stessa e le cellule fetali che attraversano la barriera placentare.
Durante la gravidanza, il sistema immunitario materno affronta il concepito
con una reazione di difesa verso l’ospite basata sul riconoscimento degli
antigeni fetali e placentari di origine paterna. Per evitare il rigetto del prodotto
semi-allogenico, la risposta immunitaria materna durante la gravidanza viene
normalmente soppressa [45] con l'inibizione dell'attivazione delle cellule T
all’interfaccia materno-fetale [46]. Il meccanismo con cui l'aborto abituale sia
legato all’allo-immunità non è chiaro. Secondo Lim et al., tre meccanismi
potrebbero esserne responsabili: una condivisione dell’antigene leucocitario
11
umano (HLA), una carenza di anticorpi bloccanti, ed un meccanismo che
coinvolga mediatori immunitari e cellule soppressorie.
Le molecole del complesso maggiore di istocompatibilità (MHC, Major
Histocompatibility Complex) di classe I sono importanti nella presentazione
dell'antigene alle cellule T ed alle cellule NK [47], e ciò può sostenere la
teoria secondo la quale l'assenza di molecole MHC di classe I nel trofoblasto
lo possa proteggere da attacchi materni di cellule NK. Tuttavia, il trofoblasto
extravilloso esprime l’HLA-C, classico prodotto MHC di classe I, e le
molecole non-classiche HLA-G,-E e-F [48,49].
Un momento cruciale all'inizio della gravidanza è l'invasione delle cellule
fetali del trofoblasto extravilloso nella decidua. La funzione esatta del
prodotto del gene HLA-G non è ancora nota ma la sua espressione tessutospecifica in cellule placentari di citotrofoblasto extravilloso [50,51]
suggerisce che l’HLA-G possa giocare un ruolo importante nella tolleranza
tissutale materno - fetale. Il ruolo essenziale di queste molecole potrebbe
risiedere nel fatto che possono inibire l'attività di una cospicua popolazione di
cellule deciduali: le cellule NK.
Alla luce del fatto che la quantità di cellule NK uterine è notevole sia alla fine
della fase luteale che nel primo trimestre di gravidanza, si può ben prevedere
che una disturbata interazione tra molecole HLA-G e cellule NK possa
portare alla compromissione dei processi riproduttivi, sebbene studi che
hanno confrontato l'espressione di queste popolazioni cellulari tra donne con
abortività ricorrente e controlli hanno mostrato risultati molto diversi ed
incoerenti [52].
Inizialmente era stato suggerito che il riconoscimento materno dell’HLA di
origine paterna sia necessario, o almeno potrebbe esserlo, nella gravidanza
normale. Come risultato, i primi studi sull’HLA nell’abortività ricorrente si
12
basavano sull'ipotesi che la maggiore somiglianza HLA tra i partners
porterebbe a risposte immunitarie protettive materne inadeguate con
conseguente perdita del feto. Sebbene siano stati effettuati un numero
considerevole di studi sulla condivisione HLA nelle coppie con abortività
ricorrente, prove convincenti di questa ipotesi sono ancora scarse.
Anche le cellule T regolatorie (Treg), oltre ad un ruolo chiave nel controllo
delle cellule NK [53], svolgono un ruolo essenziale nel sopprimere l'immunità
materna contro l’alloantigene fetale di origine paterna [54]. Le cellule Treg
sono numerose nella decidua umana [55,56] e l’aumento del loro numero
durante il ciclo mestruale prima dell'impianto dell'embrione [57] e durante la
gravidanza sembra essere un segno distintivo della tolleranza materno-fetale
[55,58,59].
L’attivazione indotta dall’allotrapianto porta ad una risposta di rifiuto
attraverso la stimolazione di alcune molecole di superficie (CD25, CD69,
HLA-DR), nonché attraverso la produzione di citochine di tipo T helper 1
(Th1).
C'è un crescente interesse nel chiarire il ruolo immunologico e biologico delle
citochine durante la gravidanza [45], soprattutto nel sangue periferico materno
[60-62], ma anche nei tessuti utero-placentari [63-66]. Le sottopopolazioni dei
linfociti T helper (CD3 + / CD4 +) possono essere classificate come T helper
1 (Th1) e T helper 2 (Th2), a seconda del loro profilo di citochine.
L'equilibrio tra l'attività Th1 e Th2 può indirizzare quindi la risposta
immunitaria in direzione dell’immunità cellulo-mediata o di quella umorale
[67-70].
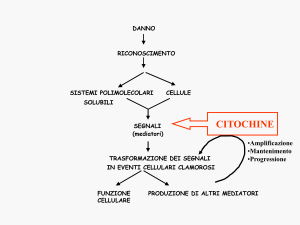
Vi è attualmente una crescente evidenza che le citochine siano anche
coinvolte come fattori autocrini, paracrini ed endocrini nel modulare la
risposta immunitaria materna e l'esito della gravidanza.
13
In particolare è stato proposto come l’andamento della gravidanza possa
dipendere dall’equilibrio delle risposte di tipo Th1/Th2 [71], anche se, più
recentemente, alcuni autori hanno suggerito che questa ipotesi rappresenta
un’eccessiva semplificazione della situazione [72].
Numerosi studi suggeriscono che una predominanza di risposte citochiniche
Th2 favorisca la gravidanza fisiologica, mentre un eccesso di risposte Th1
porti a eventi avversi della gravidanza [71,73]. E’ stato suggerito inoltre che
le citochine Th1 possano innescare processi trombotici/ infiammatori nei vasi
sanguigni materni utero-placentari attraverso l'attivazione delle cellule
endoteliali vascolari procoagulanti [74]. Al contrario, le citochine Th2
possono inibire la produzione del fattore tissutale dei monociti indotto dalle
citochine Th1 [75].
Esistono molti studi sul rilevamento nel siero materno di livelli
significativamente più alti di citochine Th2 nella gravidanza normale rispetto
a quelli degli aborti spontanei ricorrenti, così come di livelli molto più elevati
di citochine Th1 presenti nelle donne con aborti spontanei ricorrenti rispetto a
quelle con una gravidanza fisiologica [60,61,76-82].
Tuttavia le citochine sono secrete non solo dai linfociti sistemici ma anche da
altri compartimenti cellulari, come le cellule epiteliali, stromali, endometriali,
trofoblastiche e deciduali. Per questo, alcuni autori hanno indagato, seppur
con risultati contrastanti, l’espressione di citochine a livello dell'endometrio e
della decidua nelle donne con gravidanze normali e patologiche.
Anche il nostro gruppo ha precedentemente indagato il pattern di espressione
delle citochine infiammatorie e dei loro recettori, usando la tecnologia cDNA
macroarray, confrontando il tessuto trofoblastico proveniente da aborti
spontanei ricorrenti rispetto a quello da interruzioni volontarie della
gravidanza [83].
14
Sulla base di queste ipotesi immunologiche, varie terapie immunomodulanti
potrebbero essere utilizzate per inibire la citotossicità delle cellule NK, per
migliorare la formazione di anticorpi bloccanti, o per promuovere un
cambiamento di profilo citochinico Th1-Th2. Tali terapie includono
l’immunoterapia con linfociti allogenici e le immunoglobuline per via
endovenosa (IVIG). E’ stata proposta anche una procedura di induzione della
tolleranza timica per la prevenzione o il trattamento dell’abortività ricorrente
[84]. Tuttavia l'efficacia di queste terapie nella prevenzione dell'aborto
abituale dell'embrione euploide è controversa ed i risultati di studi clinici
randomizzati per valutare l'efficacia dell'immunoterapia sono stati contrastanti
[85-87].
1.3 Endometrio e abortività spontanea
L’espressione genica dei diversi tipi di cellule endometriali è regolata da
ormoni steroidei di origine ovarica e molecole paracrine secrete dalle cellule
circostanti. Come conseguenza di questa regolazione, l’endometrio passa
attraverso modificazioni cicliche che possono essere divise per semplicità in:
fase proliferativa, fase secretoria e fase mestruale. Un corretto impianto
dell’embrione dipende da tre fattori: qualità dell’embrione, stato di recettività
dell'endometrio e sincronia tra tessuto materno e blastocisti. È dunque
necessario caratterizzare lo stato di recettività dell’endometrio al fine di
prevenire un fallimento del meccanismo della riproduzione. Tuttora però non
è ancora stato trovato un singolo marker molecolare o istologico
specificamente rappresentativo di questo stato [88].
15
E’ nella fase secretiva che l’endometrio acquisisce il fenotipo recettivo che
consente l’impianto della blastocisti. Questo periodo di recettività è noto
come "Window of implantation" (WOI) e inizia al giorno diciannove e
ventesimo del ciclo con una durata di quattro o cinque giorni [89]. Se non si
verifica l'impianto, i livelli di progesterone e estrogeno diminuiscono,
contemporaneamente ad una vasocostrizione delle arterie spirali che porta a
involuzione dell’endometrio [90]. Se invece si verifica l’impianto, si va a
stabilire una sorta di dialogo tra tessuto materno e blastocisti, che risulta
possibile grazie ad una serie di complesse interazioni autocrine, paracrine e
endocrine tra le diverse molecole e i recettori che sono espressi in modo
perfettamente coordinato nell’endometrio e nell'embrione [91-94].
In seguito, la blastocisti entra in contatto con l’epitelio endometriale e vi
aderisce alla superficie e penetra nell’endometrio. Questo processo è mediato
da cellule immunitarie, citochine, fattori di crescita, chemochine e molecole
di adesione [95].
È per raggiungere questo obiettivo che l’endometrio necessita di disporre di
una regolazione genica appropriata e coordinata durante l’intero ciclo
endometriale. Proprio per questo motivo è emerso il bisogno di comprendere i
meccanismi genetici che sono alla base dei cambiamenti istologici che
avvengono durante il ciclo endometriale. Prima dell’era genomica, i
ricercatori si erano limitati a studiare “gene per gene” per determinare quali
fossero i cambiamenti molecolari responsabili delle alterazioni osservate.
I microarrays hanno permesso di tracciare un profilo dei trascritti dell’intero
genoma e sono stati ampiamente utilizzati per esplorare i diversi fenomeni
biologici, poiché permettono di misurare i livelli di espressione dell'RNA per
migliaia di geni contemporaneamente [96]. A tale scopo sono stati creati dei
16
database [97] in cui sono stati catalogati i dati relativi ai geni espressi
nell’endometrio.
Nell’era genomica le tecniche a nostra disposizione permettono di tracciare un
profilo di tutti i geni che vengono trascritti e anche tradotti [98]. Tra i più
recenti studi pubblicati in letteratura ci sono studi che identificano il profilo di
trascrizione genica in condizioni fisiologiche di ciascuna fase del ciclo
endometriale soprattutto localizzati sulla fase di recettività endometriale [99115], studi sul profilo genetico in pazienti affette da infertilità e poliabortività
[116-118], patologie endometriali [119,120], o che valutano l’alterazione dei
profili dei trascritti genici in seguito a diversi protocolli di stimolazione
ovarica [121,122].
La metodica che viene utilizzata per lo studio dei trascritti genici è quella dei
microarray.
La fase secretiva è la fase più studiata in quanto l’endometrio diviene
recettivo al fine di consentire l’impianto dell’embrione. È utile quindi
suddividerla in: iniziale, intermedia e tardiva [90]. La fase precoce è
caratterizzata dalla predominanza dei processi legati al metabolismo cellulare
(acidi grassi, lipidi, eicosanoidi e alcoli), al trasporto (con un’ampia
rappresentazione dei trasportatori delle molecole biologiche coinvolte in
questi processi metabolici), alla migrazione di cellule germinali (che può
facilitare il trasporto dello sperma e assicurare un ambiente asettico) e alla
down-regulation della proliferazione cellulare. L’incremento del metabolismo
è dovuto al fatto che questa fase è molto attiva da un punto di vista della
biosintesi, probabilmente in preparazione all’impianto dell’embrione, e
l’inibizione della mitosi è sostenuta da una down-regolazione di numerosi
fattori di crescita. Alcuni tra i principali geni iperespressi nella fase secretiva
precoce sono: MSX1 (che appartiene alla famiglia dei geni HOX, implicati
17
negli
eventi
morfogenetici),
l7ßHSD
(che
regola
la
permissività
dell’endometrio agli estrogeni), PIP5K1B (che regola un’ampia gamma di
processi cellulari, inclusi proliferazione, sopravvivenza e organizzazione del
citoscheletro) e MUC1 (che mantiene l'idratazione della superficie cellulare,
proteggendo le cellule contro i microrganismi e gli enzimi).
La fase secretoria intermedia è caratterizzata anch’essa dall’essere molto
attiva da un punto di vista metabolico e secretorio e dalla grande importanza
riposta nella risposta immunitaria di tipo innato [123]. Il cambiamento
dell'espressione genica in questa fase è dovuto al picco del progesterone, che
determina una maggiore espressione dei geni coinvolti nella regolazione del
ciclo cellulare, nel legame agli ioni, nelle proteine di trasporto dei segnali
cellulari, e nell’immunomodulazione. A differenza della fase precoce, i geni
iperespressi riguardano l’impianto dell’embrione, i processi di adesione
cellulare, il metabolismo, la risposta agli stimoli esterni, il signalling, la
risposta immune, la comunicazione cellulare e la down-regulation della
proliferazione. Una delle funzioni più rappresentative della fase secretoria
intermedia è l’adesione cellulare: l’entrata della blastocisti nell’endometrio,
divenuto recettivo, innesca la produzione di citochine da parte delle cellule
epiteliali. Queste citochine modulano la recettività dell’endometrio, tramite la
regolazione dell’espressione delle molecole di adesione.
In effetti probabilmente la de-regolazione dell’espressione delle citochine e
dei loro segnali porta a un fallimento nell’impianto o anomalie nella
formazione della placenta [124,125]. Un altro ruolo importante è giocato dalla
risposta immune: vengono iperespressi i geni coinvolti nell’attivazione della
risposta immune di tipo innato (complemento, peptidi antimicrobici, TLR),
nonché quelli coinvolti nella chemiotassi dei monociti, delle cellule T e delle
cellule NK. Un’altra importante funzione che viene svolta in questa fase è la
18
protezione dell’endometrio: troviamo infatti una up-regolazione dei geni che
traducono per antiossidanti, che proteggono la cellula dai radicali liberi e dai
metalli pesanti (metallotionina e GPXs) e di DAF (the decay accelerating
factor), la cui funzione è proteggere l’embrione dall’attacco mediato dal
complemento materno e prevenire la distruzione epiteliale.
Nella fase secretoria tardiva, i principali meccanismi che vengono messi in
atto sono vasocostrizione, contrazione delle cellule della muscolatura liscia,
emostasi e transizione da una fase di risposta immunitaria di tipo innato a una
risposta di tipo infiammatorio. I geni che vengono regolati in questa
transizione sono per la maggior parte correlati alla risposta immune
(iperespressione di recettori Fc, molecole MHC, cellule NK e T) e al sistema
immunitario di tipo innato, alla risposta immune di tipo umorale, all’emostasi,
alla coagulazione del sangue, alla biosintesi di ormoni steroidei e al
metabolismo delle prostaglandine. Il processo che si innesca nella fase
secretoria tardiva non favorisce l’impianto embrionale, quindi la transizione
dalla fase secretoria intermedia a quella tardiva definisce la fine della WOI e
il ritorno a un fenotipo endometriale non recettivo.
Come precedentemente citato, esistono numerosi studi di valutazione
dell’assetto citochinico, o più spesso di citochine isolate, a livello soprattutto
periferico [78,80-82], mentre date le peculiari funzioni di citochine e
chemochine sarebbe più corretto valutarle localmente, laddove agiscono.
Esistono pertanto alcuni lavori che hanno studiato il materiale trofoblastico
ottenuto in seguito all’aborto spontaneo [126].
Esistono pochi studi che hanno invece valutato il coinvolgimento delle
citochine a livello endometriale [127-131], in uno stato non gravidico, in
19
modo da poter indagare cosa avviene durante la fase secretiva al momento
dell’impianto della blastocisti.
Il ruolo dell’endometrio nel successo della gravidanza sembra essere sempre
più decisivo [132,133].
20
2 Sperimentazione
2.1 Obiettivo dello studio
L’obiettivo dello studio è stato quello di valutare il profilo di espressione
genica di citochine infiammatorie a livello del tessuto endometriale di donne
con pregressa abortività ricorrente rispetto a donne con anamnesi ostetrica
negativa, confrontabili per età e BMI, mediante le metodiche di PCR Array e
Real-Time PCR.
2.2 Pazienti e metodi
L’attività di ricerca è stata articolata in quattro momenti distinti:
• raccolta di campioni di materiale endometriale dalle pazienti reclutate
per lo studio
• purificazione dell’RNA totale dai tessuti recuperati;
• valutazione dei profili di espressione genica mediante PCR Array
• come verifica e come ulteriore valutazione dell’espressione genica
differenziale, alcuni geni sono stati selezionati e l’espressione del loro
RNA messaggero è stata analizzata anche con la metodica Real-time
PCR, ampliando inoltre ulteriormente la numerosità del campione.
2.2.1 Selezione delle pazienti
I soggetti partecipanti allo studio sono stati reclutati presso la Clinica
Ostetrica e Ginecologica dell’Università Politecnica delle Marche, Ospedale
21
materno-infantile Salesi, Ancona. Il campionamento è stato condotto nel
periodo di tempo compreso tra Gennaio 2012 e Gennaio 2013.
Il campionamento di tessuto endometriale è stato eseguito in:
• 5 pazienti con pregressa abortività spontanea ricorrente (aborti nel
primo trimestre e senza causa apparente)
• 5 pazienti con anamnesi ostetrica negativa (pregresse gravidanze
fisiologiche con parti a termine, assenza di pregressi aborti spontanei).
Con gli stessi criteri, il campione è stato ampliato a 12 casi e 12 controlli nel
caso della valutazione mediante Real Time PCR.
Tutte le pazienti reclutate erano di razza caucasica, avevano un ciclo
mestruale regolare.
Tra le pazienti con abortività ricorrente sono state escluse le pazienti portatrici
di condizioni mediche preesistenti, come la sindrome anticorpi antifosfolipidi,
disordini ormonali, diabete mellito, difetti cromosomici, trombofilia, PCOS,
malattie infettive e malformazioni uterine.
Criteri di esclusione per entrambi i gruppi sono stati: patologie croniche,
patologie autoimmuni, patologie connettivali e neoplastiche.
Tra i 2 gruppi non c’erano differenze significative riguardo all’età materna ed
al Body Mass Index (BMI) (Tab. 1).
Il prelievo del tessuto endometriale è stato eseguito nel corso di una
isteroscopia diagnostica, che nel caso delle pazienti con abortività spontanea
ricorrente aveva anche lo scopo di valutare la cavità endometriale dal punto di
vista anatomico, per evidenziare eventuali anomalie strutturali e la possibile
presenza di un quadro endometritico.
Il prelievo bioptico isteroscopio è stato eseguito durante la fase secretiva del
ciclo mestruale.
22
Il materiale prelevato è stato opportunamente conservato in soluzione RNAlater.
Pregressi
aborti
spontanei
ricorrenti (n=12)
CONTROLLI
(n = 12)
p
Età (anni)
36,4 + 3,5
34,6 + 5,2
N.S.
Body Mass Index
23,1 + 2,8
23,7 + 3,1
N.S.
Tab 1. Caratteristiche delle pazienti (Media ± DS)
2.2.2 Purificazione dell’RNA totale dai tessuti recuperati
Relativamente a ciascun campione l’RNA totale è stato isolato a partire da
tessuto endometriale (<5 mg), mediante l’impiego dell’RNeasy Micro Kit
(Qiagen). La liberazione dell’RNA avviene all’interno di dispositivi filtranti
centrifughi (forniti con il kit) attraverso una serie di quattro passaggi che
determinano in successione la lisi del tessuto, la denaturazione dei complessi
nucleoproteici, l’inattivazione dell’attività ribonucleasica endogena e la
rimozione di DNA e proteine contaminanti. Tale metodica combina le
proprietà distruttive e protettive della guanidina tiocianato (GTC) e del "mercaptoetanolo per inattivare le ribonucleasi presenti nell’estratto tissutale.
La GTC, infatti, assieme all’SDS provoca la distruzione dei complessi
nucleoproteici ed il rilascio dell’RNA in soluzione; la diluizione dell’estratto
tissutale con un’alta concentrazione di GTC favorisce la precipitazione delle
proteine, mentre l’RNA rimane in soluzione. L’aggiunta di etanolo provoca la
precipitazione dell’RNA che si lega al dispositivo filtrante della colonna
mentre il DNA (digerito per via enzimatica tramite impiego della DNasi I) e
le altre impurità contaminanti si raccolgono nel fondo della vial attraverso una
23
successione di lavaggi con uno specifico tampone salino. L’RNA viene eluito
dalla colonna con 14 µl di acqua “nuclease free” e raccolto in una nuova vial.
2.2.3 Analisi spettrofotometrica dell’RNA estratto
Per determinare la concentrazione e la presenza di eventuali contaminanti
(proteine, GTC, DNA) nell’RNA isolato da tessuto è stata effettuata
un’analisi spettrofotometrica: da ognuno degli estratti a disposizione è stata
prelevata un’aliquota di cui, previa opportuna diluizione in acqua sterile, è
stata determinata l’assorbanza a 260nm. La concentrazione dell’RNA estratto
è legata al valore dell’assorbanza secondo la seguente formula:
[RNA estratto] (µg/ µl)= Abs 260nm X Fattore di diluizione X 0.04
Dove:
0,04= coefficiente di estinzione molare per gli acidi nucleici a singolo
filamento.
2.2.4 Verifica dell’integrità dell’RNA estratto
Allo scopo di saggiarne l’integrità, una quantità pari a 1 µg di RNA estratto
da campioni di controllo e patologici, implementato del relativo tampone di
caricamento (Fermentas) contenente 0,015% di blu di bromofenolo, 0,015%
di xilene, 10% di glicerolo e EDTA 10 mM viene assoggettato a migrazione
elettroforetica sul gel di agarosio all’1% in TBE 1X (90 mM Tris-Borato e 2
mM EDTA, pH 8,0). Il gel si allestisce portando ad ebollizione una
sospensione di agarosio solido in un tampone acquoso (nel nostro caso TBE)
fino ad ottenere una soluzione limpida; a quest’ultima, raffreddata a 60°C, si
addiziona un volume di bromuro di etidio tale da avere una concentrazione
finale di 0,5 µg/ml. Questo colorante si intercala tra le basi azotate degli acidi
24
nucleici e li rende visibili se esposto ad una sorgente UV. La soluzione viene
quindi versata all’interno di uno stampo e raffreddandosi dà origine ad una
matrice la cui porosità è determinata dalla concentrazione del polimero stesso
nella soluzione iniziale. La migrazione elettroforetica avviene all’interno di
una vasca fornita di due elettrodi (catodo e anodo) collegati ad un generatore
di corrente. Al gel, una volta immerso nel tampone di corsa (TBE 1X), viene
applicata una differenza di potenziale di 50-70 V che dà inizio al processo
elettroforetico. Nell’intervallo di pH (7,5-7,8) sia del tampone di corsa che del
gel, l’RNA e il DNA risultano carichi negativamente e migrano dal catodo
verso l’anodo con una mobilità elettroforetica che è condizionata
esclusivamente dalle loro dimensioni dal momento che la densità di carica
(quantità di carica per unità di lunghezza) dovuta ai gruppi fosfato rimane
costante: i frammenti più lunghi mostrano maggiore difficoltà al passaggio
attraverso i pori del gel mentre i più piccoli migrano più velocemente.
2.2.5 Reazione di retro trascrizione
Il processo di retrotrascrizione si svolge grazie all’attività della trascrittasi
inversa, enzima isolato per la prima volta dai retrovirus eucariotici. Tale
enzima è in grado di sintetizzare una molecola di DNA utilizzando come
stampo un filamento di RNA; per questa ragione la trascrittasi inversa può
essere considerata una DNA polimerasi RNA-dipendente. In generale le DNA
polimerasi consentono lo svolgimento della reazione di polimerizzazione di
una catena polinucleotidica complementare ad un singolo filamento di DNA
impiegato come stampo (nella retrotrascrizione il templato è rappresentato da
una molecola di RNA); l’enzima catalizza la formazione di un legame
fosfodiesterico tra il gruppo 3’-OH del deossiribosio relativo all’ultimo
25
nucleotide ed il fosfato in 5’ del dNTP da aggiungere alla catena. Nessuna
DNA polimerasi nota può iniziare la sintesi di un filamento di DNA ex novo;
questi enzimi infatti possono solo catalizzare l’addizione dei dNTPs ad un
filamento preesistente. Per tale ragione si rende necessario l’impiego di
oligonucleotidi (primers) forniti di estremità 3’-idrossilate rivolte in direzione
del frammento da sintetizzare; i primers costituiscono un innesco per la
polimerasi che può così sintetizzare filamenti di DNA in direzione 5’#3’.
Una quantità pari a 2µg di RNA totale viene sottoposto ad una reazione di
retrotrascrizione
mediante
l’impiego
del
kit
RT2
First
Strand
(SABiosciences), secondo le istruzioni indicate nel seguente protocollo
operativo.
Miscela di digestione del DNA genomico
RNA totale # x µl
Buffer GE # 2 µl
42° C per 5 minuti
H2O RNase-free fino ad un volume di 10 µl
4°C fino alla fase successiva
RT cocktail
Buffer BC3 # 4 µl
Buffer P2 # 1 µl
Buffer RE3 # 2 µl
H2O RNase-free # 3 µl
L’RT cocktail (10 µl) viene aggiunto alla miscela precedente (10 µl). L’intera
miscela (20 µl) viene riscaldata a 42°C per 15 min, a 95°C per 5 min e
successivamente mantenuta a 4°C. Alla miscela di retrotrascrizione (20 µl)
vengono aggiunti 91 µl di H2O RNase-free.
26
2.2.6 Verifica mediante PCR del cDNA sintetizzato
La tecnica della reazione a catena della DNA polimerasi (Polymerase Chain
Reaction), ideata da Kary Mullis alla metà degli anni '80, offre la possibilità
di produrre un enorme numero di copie di specifiche sequenze di DNA senza
ricorrere al clonaggio. La PCR sfrutta alcune peculiarità della duplicazione
del DNA. La DNA polimerasi impiega un DNA a filamento singolo come
stampo per la sintesi di un nuovo filamento complementare. Gli stampi di
DNA a filamento singolo sono prodotti per denaturazione della doppia elica
mediante alta temperatura. Per avviare la sintesi, la DNA polimerasi necessita
anche di una piccola regione a doppio filamento. Quindi il punto di inizio
della sintesi può essere specificato fornendo come innesco un oligonucleotide,
il primer complementare allo stampo proprio in quel punto. La PCR consente
quindi di indirizzare la polimerasi a sintetizzare una regione specifica di
DNA. E' una tecnica così sensibile che può dare un amplificato partendo da
una singola molecola di DNA presente in una miscela o compresa in un
filamento più lungo e consentire poi la visualizzazione su gel d'agarosio.
Per avere risultati ottimali si può agire su vari parametri fisici e chimici. La
concentrazione consigliata della Taq polimerasi è normalmente compresa tra
1 e 2.5 unità per 100 µl di reazione, quella dei deossinucleotidi tra 20 e 200
µM. Una concentrazione troppo elevata di enzima porterebbe alla formazione
di prodotti secondari non specifici; una quantità ridotta consentirebbe invece
una scarsa amplificazione. E' importante ottimizzare anche la concentrazione
di magnesio in quanto può avere effetti sull'accoppiamento dei primers con il
filamento stampo, sulla temperatura di denaturazione, sulla specificità dei
prodotti, sull'attività e sulla fedeltà della polimerasi. Anche la scelta dei
primers è guidata da alcune regole: il contenuto in G+C deve essere del 50% e
si devono evitare ripetizioni successive della stessa base che potrebbero
27
facilitare appaiamenti errati. Si devono considerare anche le eventuali
strutture secondarie e la formazione di dimeri di primers che influirebbero
negativamente sul risultato dell'amplificazione. La distanza tra i siti di legame
dei primers sui filamenti del DNA deve essere appropriata, in funzione della
capacità di separazione del gel e dell'attività limite dell'enzima: non deve
essere inferiore alla lunghezza minima dei frammenti di DNA individuabili
nel gel di separazione utilizzato e nemmeno superiore alla massima lunghezza
di sequenza nucleotidica efficientemente sintetizzata dall'enzima DNA
polimerasi (circa 2000 nucleotidi). Alla provetta contenente il DNA da
amplificare vengono aggiunti i 2 oligonucleotidi che servono da primer, la
DNA polimerasi e una mistura dei 4 nucleotidi precursori.
La DNA polimerasi più usata è la Taq, un enzima inizialmente estratto e
purificato dal batterio termofilo Thermus aquaticus ma ora disponibile in
forma ingegnerizzata. Questa miscela di reazione è riscaldata a circa 94°C. A
questa temperatura le doppie eliche si sono completamente separate. La
temperatura viene poi abbassata per permettere ai primers oligonucleotidici di
appaiarsi alle sequenze complementari presenti nelle molecole di DNA.
Questa temperatura di appaiamento (annealing) è un parametro variabile
capace di determinare la specificità della PCR. Per la tappa successiva la
temperatura è portata a 72° per consentire all'enzima termostabile Taq
polimerasi di copiare il DNA. Ripetendo le tre fasi (denaturazione,
appaiamento, allungamento) nel tempo (Fig. 1), il processo può essere reso
ciclico. I cicli sono compiuti automaticamente in uno strumento chiamato
"termocycler", programmato per passare alle diverse temperature richieste nei
tempi previsti. Il numero dei cicli dipende dalla concentrazione iniziale del
DNA bersaglio e ad ogni ciclo si ha il doppio delle copie di DNA amplificato.
Come tutti i processi biochimici, la duplicazione del DNA mediante PCR non
28
è un processo esente da errori visto che, nel corso della reazione, vengono
prodotte delle copie non fedeli all’originale. Può succedere infatti che la DNA
polimerasi incorpori nella catena in allungamento nucleotidi errati; questo
accade poiché la Taq polimerasi manca dell’attività esonucleasica 3’#5’,
cioè non può svolgere la funzione di “correzione di bozze” (proofreading).
Normalmente nelle copie di DNA tali errori vengono introdotti con una
frequenza di un nucleotide sbagliato ogni 2 $ 104 nucleotidi incorporati. Se
l’errore di incorporazione avviene nelle fasi iniziali del processo di
amplificazione, si avrà un maggior numero di copie mutate rispetto a quando
la mutazione avviene nel corso degli ultimi cicli. Tale inconveniente può
essere circoscritto dando inizio alla PCR con un numero elevato di molecole
di DNA stampo; in tal caso il numero di cicli di amplificazione richiesto è
minore e tale sarà anche la quota di errori introdotti nelle copie neosintetizzate.
29
Fig. 1 Rappresentazione schematica della reazione a catena della polimerasi. Nella figura sono
rappresentati 4 cicli di PCR articolati nelle fasi di (1) Denaturazione a 94°C (2) Annealing a 55°C- 60°C (3)
Estensione a 72°C. Al DNA templato (linea blu) si appaiano i primers (linea rossa) che vengono poi estesi
dalla Taq Polimerasi (cerchio verde) per aumentare il numero delle copie della sequenza bersaglio (linea
verde).
Un’aliquota di cDNA, pari a 6 %l della miscela di retrotrascrizione (111 %l)
ottenuta al termine della procedura descritta nel paragrafo precedente, è stata
utilizzata come templato in una reazione di PCR. Tale analisi è stata realizzata
utilizzando dei primers le cui sequenze nucleotidiche sono qui di seguito
riportate e che sono stati progettati in maniera tale da condurre alla sintesi di
amplificati relativi ad una regione del cDNA per la "-actina pari a 116 bp.
30
I primers utilizzati sono i seguenti:
5’-ctcttccagccttccttcct-3’ (forward),
5’-agcactgtgttggcgtacag -3’ (reverse).
La reazione di PCR è stata eseguita in un volume di 25 µl contenente buffer
1X addizionato di Mg2+, 200 µM di ciascuno dei quattro dNTPs, 300 nM di
ciascuno dei due primers, 0,02 U/µl di DNA polimerasi Sigma Aldrich
(D4585) ed 6 µl di cDNA, quale stampo.
Il saggio di PCR è stato condotto per impiego del termociclatore PCR Express
Thermohybaid con i seguenti parametri di amplificazione:
- denaturazione iniziale a 94°C per 3’
- denaturazione a 94°C per 30’’
35 cicli di amplificazione
- annealing dei primers e polimerizzazione a 56°C per 30’’
- polimerizzazione finale a 72°C per 5’
Per verificare l’avvenuta amplificazione del retrotrascritto è stato allestito un
gel di agarosio al 2% in TAE 1X (45 mM Tris-Acetato, 1 mM EDTA, pH 8,0)
e nel gel di corsa, oltre alle miscele di PCR, è stato caricato un opportuno
marcatore di peso molecolare in grado di segnalarci se l’amplificato ottenuto
era di taglia opportuna.
2.2.7 Valutazione dei profili di espressione mediante PCR array
La Real-Time PCR è una tecnica che consente la simultanea amplificazione e
quantificazione del DNA stampo attraverso il monitoraggio in tempo reale
dell’intensità di fluorescenza che si libera dal prodotto di amplificazione
durante la reazione a catena della DNA polimerasi: questo risulta possibile
mediante l’impiego di marcatori fluorescenti il cui accumulo, a livello del
prodotto di reazione, segue la stessa cinetica della PCR. Il colorante
31
fluorescente, intercalato nel DNA a doppio filamento ed eccitato da una luce
laser, emette energia luminosa ad una specifica lunghezza d’onda. La luce
raccolta ed analizzata da uno spettrografo viene poi convogliata in una
telecamera CCD. Il detector CCD integra il segnale e lo converte in un dato
numerico che viene conservato dal software gestito da un personal computer
interfacciato al termociclatore. L’ampia disponibilità sul mercato di
fluorocromi con diversi spettri di assorbimento ed eccitazione ha reso
possibile lo sviluppo di diverse chimiche di reazione che consentono di
rilevare in modo più o meno specifico i prodotti di amplificazione e possono
essere teoricamente applicate a tutte le strumentazioni disponibili per lo
svolgimento di analisi mediante Real-Time PCR. I metodi comuni di
quantificazione del DNA amplificato mediante Real-Time PCR includono
l'impiego di molecole fluorescenti che si intercalano in maniera aspecifica al
DNA a doppio-filamento (SYBR Green) e di sonde ad ibridazione, ovvero
oligonucleotidi modificati (marcati con molecole fluorescenti) che, una volta
ibridati in maniera specifica al frammento amplificato del gene di interesse,
emettono fluorescenza (sonde TaqMan).
2.2.8 Curva di amplificazione e ciclo soglia
Durante lo svolgimento di una reazione di amplificazione del DNA mediante
Real-Time PCR, la quantità di prodotto che si forma dopo un ciclo termico
raddoppia al ciclo successivo. Misurando l’incremento di fluorescenza,
dovuto alla formazione di nuovo prodotto di reazione, all’avanzare dei cicli, è
possibile ricavare la curva di amplificazione del templato di interesse. Tale
diagramma esibisce un andamento tipicamente sigmoide, caratterizzato
fondamentalmente da tre fasi:
32
una prima fase in cui al susseguirsi dei cicli di amplificazione non corrisponde
un significativo incremento di fluorescenza; ciò è dovuto al fatto che la
quantità di prodotto amplificato genera un segnale fluorescente che si
mantiene al di sotto della soglia di rilevabilità dello strumento;
una fase esponenziale in cui, con l’avanzare dei cicli termici, inizia a
diventare significativo l’incremento di fluorescenza dovuto all’accumulo di
prodotto di amplificazione; in questa fase tutti i reagenti sono nel loro
rapporto ottimale di concentrazione e per questo la quantità di amplificato che
si forma risulta direttamente proporzionale alla quantità di templato iniziale;
una fase di plateau in cui l’intensità di fluorescenza non aumenta in maniera
significativa al progredire del numero dei cicli a causa di una ridotta
disponibilità dei substrati consumati nel corso della reazione: la quantità di
amplificato che si forma non è più direttamente proporzionale alla quantità di
DNA stampo.
La
metodica
dell’amplificato
della
nella
PCR
fase
“classica”
di
plateau
consente
di
valutare
assoggettando
a
l’entità
migrazione
elettroforetica le miscele di reazione a termine del processo di amplificazione;
la tecnica della Real-Time PCR, invece, consente di misurare la quantità di
amplificato che si forma durante la fase esponenziale del processo di
amplificazione grazie alla possibilità di monitorare in “tempo reale”
l’emissione di fluorescenza relativa al templato di interesse. In questa fase,
quindi, sarà possibile individuare un ciclo di amplificazione ottimale a cui
corrisponde un valore di fluorescenza che è misura della quantità di prodotto
che si è formato. Tale ciclo di amplificazione prende il nome di ciclo soglia
(Ct) e graficamente corrisponde al punto in cui la curva di amplificazione
interseca la linea di base (Threshold line). A questa linea virtuale, che
distingue la fluorescenza di fondo da segnali imputabili a reali amplificazioni,
33
corrisponde un valore ben definito di fluorescenza, al di sopra del quale
diventa significativo qualsiasi incremento di fluorescenza (Fig. 2).
Fig. 2 Curva di amplificazione
2.2.9 Efficienza di amplificazione e curva di Melting
Nella messa a punto delle condizioni di reazione negli esperimenti di RealTime PCR, sussistono dei passaggi fondamentali quali la produzione di
templati puliti e la progettazione di primers opportuni. In tal senso, nello
svolgimento di questo tipo di indagine biologica, risulta di fondamentale
importanza non prescindere da condizioni essenziali quali l’accuratezza e la
riproducibilità degli esperimenti. Se l’accuratezza è imputabile per gran parte
all’operatore che pianifica e realizza l’esperimento, la riproducibilità dipende
essenzialmente dall’efficienza del processo di amplificazione. Per determinare
il valore di efficienza di amplificazione relativo ad un esperimento di RealTime PCR, si sfrutta la relazione lineare che intercorre tra il valore di Ct e il
logaritmo in base 10 (Log o log10) del numero di copie del templato iniziale.
Pertanto, allestendo miscele di amplificazione con diluizioni seriali del DNA
34
target, è possibile ricavare per ciascuna un valore di Ct. Interpolando i valori
di Ct e del Log del numero delle copie di templato iniziale è possibile
costruire una retta di regressione lineare. La pendenza ricavata dall’equazione
di tale retta può essere direttamente correlata all’efficienza della reazione di
amplificazione mediante la seguente legge:
E = [10(-1/pendenza)] - 1
Tale valore di efficienza solitamente espresso in termini percentuali, da un
lato sancisce la riproducibilità ed esprime l’attendibilità del risultato
sperimentale, dall’altro definisce il range dinamico di linearità della quantità
di templato analizzabile con successo, in definite condizioni sperimentali. In
generale, un valore di efficienza compreso tra 80% e 120% risulta accettabile
per lo svolgimento di esperimenti di Real-Time PCR, assicurando una buona
riproducibilità dei dati, garanzia fondamentale per la standardizzazione della
metodica. Diversi sono i parametri che influiscono sull’efficienza di
amplificazione tra cui le condizioni di reazione (valore di pH, concentrazione
dei dNTPs, concentrazione di Mg++), l’efficienza della DNA polimerasi, il
numero dei cicli termici, la sequenza dei primers e la loro concentrazione, la
temperatura di annealing. Negli esperimenti di Real-Time PCR condotti
mediante l’impiego di sostanze fluorescenti che si intercalano in modo
aspecifico alla doppia elica del DNA amplificato, al termine del processo di
amplificazione, il software elabora la curva di Melting. Tale curva fornisce,
infatti, importanti indicazioni sulla “purezza” del prodotto di amplificazione e
quindi sull’attendibilità del risultato ottenuto. La necessità di elaborare la
curva di Melting fa riferimento a diverse considerazioni:
i coloranti fluorescenti impiegati negli esperimenti di Real-Time PCR (tra i
quali il SYBR Green rappresenta il più utilizzato e quello che consente di
35
operare in condizioni di massima sensibilità), se intercalati alla doppia elica,
emettono una fluorescenza di gran lunga superiore a quella emessa quando
sono liberi in soluzione;
il DNA a doppio filamento si denatura con l’incremento della temperatura;
a seguito della denaturazione, la molecola intercalante abbandona la doppia
elica tornando in soluzione e ciò determina un decremento della fluorescenza
del mezzo di reazione.
La temperatura di Melting (Tm) di un frammento di DNA a doppio filamento
si definisce come la temperatura alla quale, nella miscela di reazione, il 50%
del DNA presente è organizzato a doppio filamento. Ciascun frammento di
DNA a doppio filamento avrà uno specifico valore di Tm che dipende dalla
lunghezza e dalla composizione in basi del frammento in esame. Quindi,
conoscendo la Tm dell’amplicone che si deve quantizzare mediante Real-Time
PCR, è possibile indagare sulla specificità del prodotto di reazione. La
costruzione della curva di Melting è resa possibile impostando, a livello del
termociclatore, lievi incrementi di temperatura (0,5°C) al termine del processo
di amplificazione. In questa maniera, all’aumentare della temperatura, si
assiste ad un graduale decremento della fluorescenza generato dalla
denaturazione del prodotto di reazione (DNA a doppio filamento) e dal
conseguente rilascio nel mezzo di reazione del colorante intercalante. A
livello grafico, la Tm è rappresentata da un punto di flesso della curva di
Melting (Fig. 3). Riportando in ascissa la temperatura e in ordinata la derivata
1a del rapporto tra l’intensità di fluorescenza rispetto alla temperatura (dI/dT), il punto di flesso viene convertito in un picco: il picco di Melting. La
curva di Melting consente quindi di verificare la presenza di fluorescenza
36
aspecifica generata da dimeri di primers o da amplificati diversi
dall’amplicone di interesse (Fig. 4).
Tm
Fig. 3 Curva di Melting. A livello grafico i valori di Tm sono rappresentati dai punti di flesso della curva.
37
A
B
Fig. 4 (A) Rappresentazione della curva di Melting di due diversi amplificati con differenti Tm. (B)
Rappresentazione della curva di Melting, in presenza di un solo amplificato.
2.2.10 Quantizzazione e geni housekeeping
La molteplicità dei campi di indagine biomedica in cui tale metodica trova
applicazione rende la Real-Time PCR estremamente versatile; tuttavia,
38
l’analisi dell’espressione genica risulta il settore di ricerca in cui questa
tecnica di biologia molecolare mostra il suo più ampio impiego.
La Real-Time PCR consente infatti di effettuare una valutazione dei livelli di
espressione di un gene di interesse grazie alla determinazione quantitativa del
relativo RNA messaggero, dopo la sua conversione a cDNA. I prodotti di
amplificazione possono essere quantizzati in modo assoluto o relativo. La
quantizzazione assoluta è basata sulla costruzione di una curva standard in cui
vengono riportati i valori di Ct relativi all’amplificazione di diluizioni seriali
di DNA plasmidico, o altre forme di DNA, contenenti nella loro sequenza
nucleotidica l’amplicone relativo al gene di interesse. Verificata la
coincidenza dei livelli di efficienza di amplificazione tra campioni incogniti e
standard è possibile risalire al numero di copie del prodotto di amplificazione
del gene di interesse presenti nel campione indagato per interpolazione del
suo valore di Ct sulla curva standard di riferimento. Il metodo di
quantizzazione relativa consente invece di valutare le differenze nei livelli di
espressione di un gene tra un campione ed il relativo controllo. In questo
senso tale approccio quantitativo, pur risultando più semplice dal punto di
vista procedurale, non consente la determinazione del numero di copie del
prodotto di amplificazione (relativo al gene di cui si intende analizzare
l’espressione), bensì il semplice confronto tra i cicli soglia di amplificazione
del gene in esame tra il campione ed il suo controllo. Per poter effettuare tale
confronto è necessario disporre di uno “standard interno”, di un riferimento,
comune sia al campione, sia al controllo, rappresentato da un gene diverso dal
gene di interesse (GOI), ed espresso costitutivamente nel campione e nel
controllo. Tali geni costitutivi, denominati housekeeping (HKG), sono
caratterizzati dal fatto che la loro espressione segue l’attività trascrizionale
della cellula: ne consegue che la quantità di RNA e quindi di cDNA utilizzato
39
quale templato è ad essa proporzionale. In questo senso, prima di valutare la
differenza tra i Ct del GOI nel campione e nel controllo, è necessario
determinare per ciascuno i valori di Ct del HKG. Calcolati tali valori, si può
procedere alla determinazione dell’espressione relativa del GOI secondo lo
schema che segue:
GOI
campione
GOI
Ct
controllo
HKG Ct
Ct
HKG Ct
S!C
C!Ct
t
espressione relativa = 2
- ( S&Ct - C&Ct)
Tale valore, che risulta attendibile a parità di efficienza di amplificazione del
GOI e dell’HKG, esprime dunque l’entità dell’espressione differenziale del
gene d’interesse tra il campione ed il suo controllo.
La valutazione quantitativa dell’espressione dei geni selezionati è stata
condotta utilizzando uno strumento costituito da un termociclatore provvisto
di un lettore ottico per la lettura della fluorescenza.
40
2.2.11 PCR Array
Il nostro progetto di ricerca, volto a valutare l’eventuale flogosi endometriale
alla base della poliabortività, si è basato su uno studio realizzato mediante
PCR Array. Tale indagine è stata condotta con lo scopo di valutare i
cambiamenti nel pattern di espressione di geni coinvolti nella risposta
infiammatoria, analizzando i profili di espressione genica dei campioni
cellulari in esame.
L’analisi mediante PCR Array è una tecnologia che permette la simultanea
valutazione di numerosi geni in un singolo esperimento con una capacità
informativa significativamente maggiore dell’analisi separata di singoli o di
soli pochi markers.
La tecnica è articolata in 4 steps:
sintesi del cDNA (fase precedentemente descritta),
allestimento della miscela di PCR,
amplificazione e analisi di Melting,
raccolta ed elaborazione dei dati.
Per quanto concerne la fase di allestimento della miscela di PCR, un aliquota
pari a 102 %l del cDNA sintetizzato in precedenza viene addizionata a 1350 %l
di RT Real-Time SYBR Green/Fluorescein PCR master mix e a 1248 %l di
H2O sterile. Tale miscela viene quindi trasferita in aliquote da 25 %l
all’interno di pozzetti raggruppati in un supporto solido costituito da una
piastra (Figura 5 A).
A tale scopo sono state impiegate piastre contenenti 96 pozzetti prodotte dalla
SABiosciences e indicate con la sigla RT2 Profiler PCR Array System Human Cytokines & Chemokines PCR arrays (PAHS – 150D). All’interno di
ciascun pozzetto è presente in forma liofilizzata un set di primers per
l’amplificazione di un gene specifico.
41
(A)
(B)
Fig. 5 (A) Immagine relativa ad una piastra PCR Array contenente 96 pozzetti. (B) Rappresentazione
schematica della dislocazione dei pozzetti in cui avviene l’amplificazione dei geni di interesse, dei geni
housekeeping e delle sequenze per il controllo della contaminazione da DNA genomico, dell’efficienza di
retrotrascrizione e di amplificazione mediante PCR.
In particolare nelle piastre da 96 pozzetti è possibile esplorare l’espressione di
84 geni di interesse relativi ad uno specifico pathway (nel nostro caso i geni
42
coinvolti nella risposta immunitaria) e di 5 geni housekeeping, utilizzati per la
normalizzazione dei dati sperimentali, indispensabile per poter effettuare la
valutazione dell’espressione genica differenziale nei vari campioni. Inoltre è
possibile valutare la presenza di DNA genomico contaminante il cDNA
sintetizzato, nonché di saggiare l’efficienza dei processi di retrotrascrizione e
di amplificazione mediante PCR (Figura 5 B).
L’analisi mediante PCR Array delle piastre relative ai campioni presi in
esame è stata condotta utilizzando iCycler, quale termociclatore accoppiato
allo strumento di acquisizione CFX96 Real-Time PCR Detection System
(Bio-Rad).
L’amplificazione ha previsto l’adozione delle seguenti condizioni:
1. denaturazione iniziale a 95°C per 10’
2. denaturazione a 95°C per 15’’
40
cicli
di
amplificazione
3. annealing dei primers e polimerizzazione a 60°C
Al termine della reazione di amplificazione l’allestimento delle curve di
Melting relative a ciascun amplificato è stato condotto secondo il seguente
protocollo:
1. mantenimento delle miscele a 95°C per 10’’
2. incrementi consecutivi di 0,5°C (da 65°C a 95°C) compiuti ogni 5’’.
L’acquisizione della fluorescenza relativa agli amplificati è stata effettuata al
termine della fase 3 di ciascun ciclo termico del processo di amplificazione e
al compimento di ogni singolo incremento relativo alla fase di Melting.
Di seguito viene riportato l’elenco dei geni la cui espressione viene esplorata
nella piastra RT2 Profiler PCR Array System - Human Cytokines &
Chemokines (PAHS – 150D), prodotta dalla SABiosciences. I geni vengono
43
rappresentati secondo l’ordine di dislocazione nei pozzetti sulla piastra (Tab.
2), in raggruppamento funzionale (Tab. 3) e successivamente in elenco
dettagliato (Tab. 4).
Tabella 2: Array Layout
ADIPOQ
BMP2
BMP4
BMP6
BMP7
C5
CCL1
CCL11
CCL13
CCL17
CCL18
CCL19
A01
A02
A03
A04
A05
A06
A07
A08
A09
A10
A11
A12
CCL2
CCL20
CCL21
CCL22
CCL24
CCL3
CCL7
CCL8
CD40LG
CNTF
CSF1
B08
B09
B10
B11
B12
CCL5 B07
B01
B02
B03
B04
B05
B06
CSF2
CSF3
CX3CL1
CXCL1
CXCL10
CXCL11
CXCL12
CXCL13
CXCL16
CXCL2
CXCL5
CXCL9
C01
C02
C03
C04
C05
C06
C07
C08
C09
C10
C11
C12
FASLG
GPI
IFNA2
IFNG
IL10
IL11
IL12A
IL12B
IL13
IL15
IL16
IL17A
D01
D02
D03
D04
D05
D06
D07
D08
D09
D10
D11
D12
IL17F
IL18
IL1A
IL1B
IL1RN
IL2
IL21
IL22
IL23A
IL24
IL27
IL3
E01
E02
E03
E04
E05
E06
E07
E08
E09
E10
E11
E12
IL4
IL5
IL6
IL7
IL8
IL9
LIF
LTA
LTB
MIF
MSTN
NODAL
F01
F02
F03
F04
F05
F06
F07
F08
F09
F10
F11
F12
OSM
PPBP
SPP1
TGFB2
THPO
TNF
TNFRSF11B
TNFSF10
TNFSF11
TNFSF13B
VEGFA
XCL1
G01
G02
G03
G04
G05
G06
G07
G08
G09
G10
G11
G12
ACTB
B2M
GAPDH
HPRT1
RPLPO
HGDC
RTC
RTC
RTC
PPC
PPC
PPC
H01
H02
H03
H04
H05
H06
H07
H06
H09
H10
H11
H12
44
Tabella 3: Functional gene grouping
Chemokines: CCL1, CCL11, CCL13, CCL17, CCL18, CCL19, CCL2,
CCL20, CCL21, CCL22, CCL24, CCL3, CCL5, CCL7, CCL8, CX3CL1,
CXCL1, CXCL10, CXCL11, CXCL12, CXCL13, CXCL16, CXCL2,
CXCL5, CXCL9, PF4, PPBP, XCL1.
Interleukins: IL10, IL11, IL12A, IL12B, IL13, IL15, IL16, IL17A, IL17F,
IL18, IL1A, IL1B, IL1RN, IL2, IL21, IL22, IL23A, IL24, IL27, IL3, IL4,
IL5, IL6, IL7, IL8, IL9.
Interferons: IFNA2, IFNG.
Growth Factors: BMP2, BMP4, BMP6, BMP7, CNTF, CSF1, CSF2, CSF3,
GPI, LIF, MSTN, NODAL, OSM, THPO, VEGFA.
TNF Superfamily: CD40LG, FASLG, LTA, LTB, TNF, TNFRSF11B,
TNFSF10, TNFSF11, TNFSF13B.
Other Cytokines: ADIPOQ, MIF, SPP1, TGFB2.
Anti-Inflammatory Cytokines: CCL18, CCL19, CCL21, IL10, IL11,
IL12A, IL12B, IL13, IL18, IL2, IL22, IL23A, IL24, IL4, IL6, TGFB2.
Tabella 4: Gene Table
Position
UniGene
RefSeq
Symbol
A01
Hs.80485
NM_004797
ADIPOQ
A02
Hs.73853
NM_001200
A03
Hs.68879
A04
A05
Description
Gene Name
Adiponectin, C1Q and collagen domain ACDC, ACRP30, ADIPQTL1,
containing
ADPN, APM-1, APM1, GBP28
BMP2
Bone morphogenetic protein 2
BMP2A
NM_130851
BMP4
Bone morphogenetic protein 4
Hs.285671
NM_001718
BMP6
Bone morphogenetic protein 6
VGR, VGR1
Hs.473163
NM_001719
BMP7
Bone morphogenetic protein 7
OP-1
45
BMP2B, BMP2B1, MCOPS6,
OFC11, ZYME
CPAMD4,
A06
Hs.494997
NM_001735
C5
Complement component 5
A07
Hs.72918
NM_002981
CCL1
Chemokine (C-C motif) ligand 1
A08
Hs.54460
NM_002986
CCL11
Chemokine (C-C motif) ligand 11
A09
Hs.414629
NM_005408
CCL13
Chemokine (C-C motif) ligand 13
FLJ17816,
FLJ17822, MGC142298
I-309, P500, SCYA1, SISe,
TCA3
MGC22554, SCYA11
CKb10, MCP-4, MGC17134,
NCC-1,
NCC1,
SCYA13,
SCYL1
A-152E5.3,
A10
Hs.546294
NM_002987
CCL17
Chemokine (C-C motif) ligand 17
ABCD-2,
MGC138271,
MGC138273,
SCYA17, TARC
A11
A12
Hs.143961
Hs.50002
NM_002988
NM_006274
CCL18
CCL19
Chemokine
(C-C
motif)
ligand
(pulmonary and activation-regulated)
Chemokine (C-C motif) ligand 19
18
AMAC-1, AMAC1, CKb7, DCCK1, DCCK1, MIP-4, PARC,
SCYA18
CKb11,
ELC,
GDCF-2,
B01
Hs.303649
NM_002982
CCL2
Chemokine (C-C motif) ligand 2
MGC34433,
MIP-3b, MIP3B, SCYA19
HC11,
MCAF,
HSMCR30,
MCP-1,
MCP1,
MGC9434, SCYA2, SMC-CF
B02
Hs.75498
NM_004591
CCL20
Chemokine (C-C motif) ligand 20
B03
Hs.57907
NM_002989
CCL21
Chemokine (C-C motif) ligand 21
CKb4, LARC, MIP-3a, MIP3A,
SCYA20, ST38
6Ckine,
CKb9,
MGC34555,
ECL,
SCYA21,
SLC,
TCA4
B04
Hs.534347
NM_002990
CCL22
Chemokine (C-C motif) ligand 22
B05
Hs.247838
NM_002991
CCL24
Chemokine (C-C motif) ligand 24
B06
Hs.514107
NM_002983
CCL3
Chemokine (C-C motif) ligand 3
ABCD-1, DC, B-CK, MDC,
MGC34554, SCYA22, STCP-1
Ckb-6,
MPIF-2,
G0S19-1, LD78ALPHA, MIP1-alpha, MIP1A, SCYA3
D17S136E,
B07
Hs.514821
NM_002985
CCL5
Chemokine (C-C motif) ligand 5
MPIF2,
SCYA24
MGC17164,
RANTES,
SCYA5,
SISd,
TCP228
FIC, MARC, MCP-3, MCP3,
B08
Hs.251526
NM_006273
CCL7
Chemokine (C-C motif) ligand 7
MGC138463,
MGC138465,
NC28, SCYA6, SCYA7
B09
Hs.271387
NM_005623
CCL8
Chemokine (C-C motif) ligand 8
46
HC14,
MCP-2,
SCYA10, SCYA8
MCP2,
CD154, CD40L, HIGM1, IGM,
B10
Hs.592244
NM_000074
CD40LG
CD40 ligand
IMD3,
T-BAM,
TNFSF5,
TRAP, gp39, hCD40L
B11
Hs.715806
NM_000614
CNTF
Ciliary neurotrophic factor
HCNTF
B12
Hs.591402
NM_000757
CSF1
Colony stimulating factor 1 (macrophage)
MCSF, MGC31930
C01
Hs.1349
NM_000758
CSF2
C02
Hs.2233
NM_000759
CSF3
Colony stimulating factor 2 (granulocyte- GMCSF,
macrophage)
MGC131935,
MGC138897
C17orf33,
Colony stimulating factor 3 (granulocyte)
ABCD-3,
C03
Hs.531668
NM_002996
CX3CL1
CSF3OS,
GCSF,
C3Xkine,
CXC3,
MGC45931
Chemokine (C-X3-C motif) ligand 1
CXC3C, NTN, NTT, SCYD1,
fractalkine, neurotactin
Chemokine
C04
Hs.789
NM_001511
CXCL1
(melanoma
(C-X-C
growth
motif)
ligand
stimulating
alpha)
C05
Hs.632586
NM_001565
CXCL10
Chemokine (C-X-C motif) ligand 10
C06
Hs.632592
NM_005409
CXCL11
Chemokine (C-X-C motif) ligand 11
1
activity,
FSP, GRO1, GROa, MGSA,
MGSA-a, NAP-3, SCYB1
C7, IFI10, INP10, IP-10,
H174,
I-TAC,
IP-9,
MGC102770,
IP9,
SCYB11,
SCYB9B, b-R1
C07
Hs.522891
NM_000609
CXCL12
Chemokine (C-X-C motif) ligand 12
C08
Hs.100431
NM_006419
CXCL13
Chemokine (C-X-C motif) ligand 13
C09
Hs.708201
NM_022059
CXCL16
Chemokine (C-X-C motif) ligand 16
C10
Hs.590921
NM_002089
CXCL2
Chemokine (C-X-C motif) ligand 2
IRH, PBSF, SCYB12, SDF1,
SDF1A, SDF1B, TLSF, TPAR1
ANGIE,
ANGIE2,
BCA-1,
BCA1, BLC, BLR1L, SCYB13
CXCLG16,
SR-PSOX,
SRPSOX
CINC-2a,
GRO2,
GROb,
MGSA-b,
MIP-2a,
MIP2,
MIP2A, SCYB2
C11
Hs.89714
NM_002994
CXCL5
Chemokine (C-X-C motif) ligand 5
C12
Hs.77367
NM_002416
CXCL9
Chemokine (C-X-C motif) ligand 9
D01
Hs.2007
NM_000639
FASLG
Fas ligand (TNF superfamily, member 6)
ENA-78, SCYB5
CMK, Humig, MIG, SCYB9,
crg-10
APT1LG1, CD178, CD95-L,
CD95L, FASL, TNFSF6
AMF,
D02
Hs.466471
NM_000175
GPI
Glucose-6-phosphate isomerase
DKFZp686C13233,
GNPI, NLK, PGI, PHI, SA-36,
SA36
D03
Hs.211575
NM_000605
IFNA2
Interferon, alpha 2
47
IFN-alphaA,
IFNA,
INFA2,
MGC125764, MGC125765
D04
Hs.856
NM_000619
IFNG
Interferon, gamma
D05
Hs.193717
NM_000572
IL10
Interleukin 10
IFG, IFI
CSIF,
IL-10,
MGC126450,
IL10A,
MGC126451,
TGIF
D06
Hs.467304
NM_000641
IL11
D07
Hs.673
NM_000882
IL12A
Interleukin 11
Interleukin
AGIF, IL-11
12A
(natural
killer
cell
stimulatory factor 1, cytotoxic lymphocyte
maturation factor 1, p35)
Interleukin
D08
Hs.674
NM_002187
IL12B
12B
(natural
killer
cell
stimulatory factor 2, cytotoxic lymphocyte
maturation factor 2, p40)
CLMF, IL-12A, NFSK, NKSF1,
P35
CLMF,
CLMF2,
ALRH,
D09
Hs.845
NM_002188
IL13
Interleukin 13
IL-12B,
NKSF, NKSF2
BHR1,
MGC116786,
IL-13,
MGC116788,
MGC116789, P600
D10
Hs.654378
NM_000585
IL15
Interleukin 15
D11
Hs.459095
NM_004513
IL16
Interleukin 16
IL-15, MGC9721
FLJ16806,
FLJ42735,
FLJ44234,
LCF,
NIL16,
PRIL16, prIL-16
D12
Hs.41724
NM_002190
IL17A
Interleukin 17°
CTLA8, IL-17, IL-17A, IL17
E01
Hs.272295
NM_052872
IL17F
Interleukin 17F
IL-17F, ML-1, ML1
E02
Hs.83077
NM_001562
IL18
E03
Hs.1722
NM_000575
E04
Hs.126256
E05
Hs.81134
Interleukin 18 (interferon-gamma-inducing IGIF,
IL-18,
IL-1g,
IL1F4,
factor)
MGC12320
IL1A
Interleukin 1, alpha
IL-1A, IL1, IL1-ALPHA, IL1F1
NM_000576
IL1B
Interleukin 1, beta
IL-1, IL1-BETA, IL1F2
NM_000577
IL1RN
Interleukin 1 receptor antagonist
DIRA, ICIL-1RA, IL-1RN, IL1ra, IL-1ra3, IL1F3, IL1RA,
IRAP, MGC10430, MVCD4
E06
Hs.89679
NM_000586
IL2
Interleukin 2
IL-2, TCGF, lymphokine
E07
Hs.567559
NM_021803
IL21
Interleukin 21
IL-21, Za11
IL-21, IL-22, IL-D110, IL-TIF,
E08
Hs.287369
NM_020525
IL22
Interleukin 22
ILTIF,
MGC79382,
MGC79384, TIFIL-23, TIFa,
zcyto18
E09
Hs.98309
NM_016584
IL23A
Interleukin 23, alpha subunit p19
E10
Hs.411311
NM_006850
IL24
Interleukin 24
48
IL-23,
IL-23A,
IL23P19,
MGC79388, P19, SGRF
C49A, FISP, IL10B, MDA7,
MOB5, ST16
IL-27, IL-27A, IL27A, IL27p28,
E11
Hs.528111
NM_145659
IL27
Interleukin 27
E12
Hs.694
NM_000588
IL3
F01
Hs.73917
NM_000589
IL4
F02
Hs.2247
NM_000879
IL5
F03
Hs.654458
NM_000600
IL6
Interleukin 6 (interferon, beta 2)
BSF2, HGF, HSF, IFNB2, IL-6
F04
Hs.591873
NM_000880
IL7
Interleukin 7
IL-7
F05
Hs.624
NM_000584
IL8
Interleukin 8
IL30, MGC71873, p28
Interleukin 3 (colony-stimulating factor, IL-3,
multiple)
MCGF,
MGC79398,
MGC79399, MULTI-CSF
BCGF-1,
Interleukin 4
BCGF1,
BSF-1,
BSF1, IL-4, MGC79402
Interleukin 5 (colony-stimulating factor,
eosinophil)
EDF, IL-5, TRF
CXCL8, GCP-1, GCP1, LECT,
LUCT,
LYNAP,
MDNCF,
MONAP, NAF, NAP-1, NAP1
F06
Hs.960
NM_000590
IL9
Interleukin 9
F07
Hs.2250
NM_002309
LIF
F08
Hs.36
NM_000595
LTA
F09
Hs.376208
NM_002341
LTB
F10
Hs.407995
NM_002415
MIF
F11
Hs.41565
NM_005259
MSTN
Myostatin
GDF8
F12
Hs.370414
NM_018055
NODAL
Nodal homolog (mouse)
MGC138230
G01
Hs.248156
NM_020530
OSM
Oncostatin M
MGC20461
Leukemia
HP40, IL-9, P40
inhibitory
factor
(cholinergic
alpha
(TNF
superfamily,
beta
(TNF
superfamily,
differentiation factor)
Lymphotoxin
member 1)
Lymphotoxin
member 3)
Macrophage
migration
inhibitory
(glycosylation-inhibiting factor)
factor
CDF, DIA, HILDA
LT, TNFB, TNFSF1
TNFC, TNFSF3, p33
GIF, GLIF, MMIF
B-TG1,
Beta-TG,
CTAP-III,
CTAP3, CTAPIII, CXCL7, LAG02
Hs.2164
NM_002704
PPBP
Pro-platelet basic protein (chemokine (C-X- PF4, LDGF, MDGF, NAP-2,
C motif) ligand 7)
PBP, SCAR10, SCYB7, TC1,
TC2, TGB, TGB1, THBGB,
THBGB1
G03
Hs.313
NM_000582
SPP1
Secreted phosphoprotein 1
G04
Hs.133379
NM_003238
TGFB2
Transforming growth factor, beta 2
G05
Hs.1166
NM_000460
THPO
Thrombopoietin
49
BNSP,
BSPI,
ETA-1,
MGC110940, OPN
MGC116892, TGF-beta2
MGC163194, MGDF, MKCSF,
ML, MPLLG, TPO
G06
Hs.241570
NM_000594
TNF
G07
Hs.81791
NM_002546
TNFRSF11B
G08
Hs.478275
NM_003810
TNFSF10
G09
Hs.333791
NM_003701
TNFSF11
G10
G11
Hs.525157
Hs.73793
NM_006573
NM_003376
TNFSF13B
VEGFA
Tumor necrosis factor
Tumor necrosis factor receptor superfamily,
member 11b
DIF,
TNF-alpha,
TNFA,
TNFSF2
MGC29565, OCIF, OPG, TR1
Tumor necrosis factor (ligand) superfamily, APO2L, Apo-2L, CD253, TL2,
member 10
Tumor necrosis factor (ligand) superfamily,
member 11
Tumor necrosis factor (ligand) superfamily,
member 13b
Vascular endothelial growth factor A
TRAIL
CD254, ODF, OPGL, OPTB2,
RANKL, TRANCE, hRANKL2,
sOdf
BAFF, BLYS, CD257, DTL,
TALL-1,
TALL1,
THANK,
TNFSF20, ZTNF4
MGC70609, MVCD1, VEGF,
VPF
ATAC, LPTN, LTN, SCM-1,
G12
Hs.546295
NM_002995
XCL1
Chemokine (C motif) ligand 1
SCM-1a,
SCM1,
SCM1A,
SCYC1
H01
Hs.520640
NM_001101
ACTB
Actin, beta
PS1TP5BP1
H02
Hs.534255
NM_004048
B2M
Beta-2-microglobulin
-
H03
Hs.592355
NM_002046
GAPDH
Glyceraldehyde-3-phosphate dehydrogenase
G3PD, GAPD, MGC88685
H04
Hs.412707
NM_000194
HPRT1
Hypoxanthine phosphoribosyltransferase 1
HGPRT, HPRT
H05
Hs.546285
NM_001002
RPLP0
Ribosomal protein, large, P0
H06
N/A
SA_00105
HGDC
Human Genomic DNA Contamination
HIGX1A
H07
N/A
SA_00104
RTC
Reverse Transcription Control
RTC
H08
N/A
SA_00104
RTC
Reverse Transcription Control
RTC
H09
N/A
SA_00104
RTC
Reverse Transcription Control
RTC
H10
N/A
SA_00103
PPC
Positive PCR Control
PPC
H11
N/A
SA_00103
PPC
Positive PCR Control
PPC
H12
N/A
SA_00103
PPC
Positive PCR Control
PPC
50
L10E,
LP0,
MGC111226,
MGC88175, P0, PRLP0, RPP0
2.2.12 Raccolta ed elaborazione dei dati
L’analisi dei dati al termine della reazione di amplificazione e
dell’allestimento delle curve di Melting è stata condotta mediante l’impiego
del CFX Manager Software, Version 1.5 (Bio-Rad). Negli esperimenti relativi
ai campioni presi in esame la threshold è stata posizionata sempre al
medesimo valore di fluorescenza (100). Tale procedura ha consentito di
determinare per ciascun gene il valore di Ct. Sono stati considerati non
attendibili (negative calls) tutti i segnali a cui corrispondeva un valore di ciclo
soglia pari a N/A o maggiore di 35. In merito all’analisi di Melting sono stati
considerati attendibili gli amplificati con un picco simmetrico rispetto al
proprio asse mediano, con una base non superiore a 4-5°C, che sul grafico
descrivesse una punta (spike) e la cui temperatura non fosse inferiore a 77°C.
Successivamente per ciascuna piastra è stato verificato che:
il Ct relativo all’amplificato di controllo per la contaminazione da DNA
genomico (GDC) fosse N/A o ! 35,
la differenza tra la media dei Ct relativi agli amplificati di controllo
dell’efficienza della retrotrascrizione (RTC) e la media dei Ct relativi agli
amplificati di controllo dell’efficienza della PCR (PPC) fosse ' 5,
il Ct medio relativo agli amplificati di controllo dell’efficienza della PCR
(PPC) fosse 20 ± 2.
Relativamente ai campioni presi in esame, per ciascun gene è stato
determinato il &Ct, adottando la beta actina quale housekeeping. Per calcolare
l’espressione relativa di ciascun gene in un campione rispetto al suo controllo
è stata applicata la formula 2-&&Ct secondo le modalità descritte in precedenza.
51
2.2.13 Real Time PCR
Come ulteriore valutazione dell’espressione genica differenziale, alcuni geni
sono stati selezionati e l’espressione del loro RNA messaggero è stata
analizzata anche con la metodica Real-time PCR. Per questa ulteriore
valutazione è stato inoltre aumentato il campione in studio: sono stati
analizzate biopsie endometriali provenienti da 12 pazienti con pregressa
abortività spontanea ricorrente e 12 pazienti con anamnesi ostetrica negativa.
Nell’ambito dei 33 geni, ne abbiamo selezionati 6 diversamente up- o downregolati nel gruppo patologico rispetto al gruppo di controllo (BMP7,
CD40LG, CCL19, CCL20, CCL21, PPBP) e li abbiamo singolarmente
indagati mediante Real-Time PCR.
Per ottenere templati utili alla valutazione quantitativa mediante Real-Time
PCR dell’espressione del gene prescelto, sono stati sintetizzati i cDNA
corrispondenti a partire dagli RNA purificati come precedentemente descritto.
La reazione di retrotrascrizione, eseguita utilizzando il kit M-MLV RT
Promega, è una procedura articolata in 2 steps:
al campione di RNA totale (2 µg) vengono aggiunti i nonameri random quali
primers (0,1 µg/µl) e la soluzione, del volume finale di 15 µl, viene incubata a
70°C per 5’ e poi posta in ghiaccio;
vengono quindi aggiunti alla soluzione un enzima inibitore delle ribonucleasi
(25 U), un opportuno tampone, la miscela di dNTP (acronimo di
deossiribonucleotiditrifosfato) (10 mM) e da ultimo l’enzima della trascrittasi
inversa M-MLV (200 U) in un volume finale di 25 µl; tale miscela viene
quindi incubata a 37°C per 60’ e infine la temperatura viene abbassata a 4°C.
52
Gene
Descrizione
Homo
BMP7
CDS
sapiens
morphogenetic
bone
protein
7
(BMP7)
CD40LG
CCL19
CCL20
CCL21
TCGTCAACCTCGTGGAAC
55.1
ATCCGGAACGTCTCAT
TGTC
CCDS146
GAAGGCTTTGTGAAGGAT
ATCCTTTTTCAGCCCA
(CD40LG)
59.1
ATAATG
CTGT
Homo sapiens chemokine (C-
CCDS657
CCGCAGCAGTTAACCTAT
CGCTCACACTCACACT
C motif) ligand 19 (CCL19)
0.1
GAC
CACA
Homo sapiens chemokine (C-
CCDS465
C motif) ligand 20 (CCL20)
36.1
Homo sapiens chemokine (C-
CCDS657
CCCAGCTATCCTGTTCTTG
TCCTTTCTTGCCAGTCT
C motif) ligand 21 (CCL21)
1.1
C
TGG
CCDS356
AACCAAGTCGAAGTGATA
CCGCAGCAGTTAACCT
3.1
GCC
ATGAC
Homo sapiens actin, beta
CCDS534
AAATCTGGCACCACACCT
CATGATCTGGGTCATC
(ACTB)
1.1
TC
TTCTC
basic protein (chemokine (CX-C motif) ligand 7) (PPBP)
ACTB
CCDS134
Primer reverse (5'-3')
Homo sapiens CD40 ligand
Homo sapiens pro-platelet
PPBP
Primer forward (5'-3')
CATCAATGCTATCATCTTT
CACAC
TGGGCTATGTCCAATT
CCAT
Tab.5. Primers impiegati per la validazione del profilo di espressione.
Il cDNA, ottenuto in tal modo, è stato quindi utilizzato quale stampo nelle
analisi mediante Real-Time PCR condotte al fine di validare i dati
precedentemente ottenuti.
Per ridurre ulteriormente la possibilità di falsi positivi, attribuibili alla
amplificazione di DNA genomico contaminante nella preparazione del
cDNA, i primers sono stati costruiti utilizzando il software Primer3
disponibile on-line (http://frodo.wi.mit.edu/cgibin/primer3/primer3_www.cgi)
53
a partire dalla CDS (coding sequence) dell’RNA messaggero maturo
depositata in GeneBank (Tab.5).
!
Tutti i primers da noi costruiti sono stati testati prima in PCR classica per la
loro specificità nell’amplificare il tratto di interesse e poi in Real-Time PCR
per verificarne l’efficienza.
La composizione delle miscele di reazione e il dosaggio dei geni:
Nello studio di Real-Time PCR sono state allestite miscele di reazione di 25
µl (in triplicato per ciascun campione) contenente:
12,5 µl di iQTM Sybr Green Super Mix (BIORAD)
1 µl di cDNA quale stampo
Primer specifici ad una concentrazione finale di 200nM e di 300nM
rispettivamente per lo studio dei geni di interesse (GOI) e della "-actina
(ACTB).
L’espressione relativa dei GOI (BMP7, CD40LG, CCL19, CCL20, CCL21,
PPBP) rispetto al controllo (ACTB) è stata calcolata come descritto in
precedenza.
In questo studio l’indagine di Real-Time PCR è stata da noi condotta tramite
l’impiego
del termociclatore
CFX96
accoppiato
allo
strumento
di
acquisizione CFX96 Real-Time PCR Detection System (Bio-Rad).
2.2.14 Analisi Statistica
Tutte le analisi statistiche sono state condotte usando GraphPad Prism
Software version 5.00 for Windows (GraphPad Software, San Diego, CA,
USA). Tutti i valori sono stati espressi come Media ± Deviazione Standard. Il
test di Mann-Whitney è stato effettuato per valutare la significatività
statistica, con p inferiore al valore di 0.05.
54
2.3 Risultati
2.3.1 Analisi spettrofotometrica dell’RNA estratto
Al fine di verificare la qualità e determinare la concentrazione dell’RNA
isolato secondo le modalità descritte in precedenza è stata effettuata un’analisi
spettrofotometrica. Dallo spettro di assorbimento relativo è possibile ricavare
i valori di assorbanza a lunghezze d’onda di 230, 260 e 280 nm, utili sia per
calcolare la concentrazione degli acidi nucleici in soluzione sia per verificare
la purezza dell’RNA estratto. In Fig.6 è riportato lo spettro di uno dei
campioni
analizzati;
come
si
può
notare
l’RNA
non
presentava
contaminazioni di natura proteica, infatti:
a 260 nm si osserva il massimo valore di assorbanza,
e non si riscontrano picchi di assorbimento nell’intorno di 280 nm (imputabili
ad aminoacidi aromatici) che sono indicativi di una contaminazione da
proteine.
Una conferma ulteriore di tali risultati è stata ottenuta calcolando i seguenti
rapporti:
assorbanza a 260 nm/assorbanza a 280 nm = 1,9,
assorbanza a 260 nm/assorbanza a 230 nm = 1,7.
Il numero puro risultante da entrambi i rapporti deve essere compreso
nell’intervallo 1,7-2,1. Relativamente al primo rapporto qualsiasi valore
inferiore indica la presenza di un’eventuale contaminazione da proteine. Per
quanto concerne il secondo rapporto, un basso quoziente depone a favore di
una contaminazione da guanidina tiocianato, che potrebbe interferire con le
applicazioni successive. I valori relativi ad entrambi i parametri sono risultati
di fatto compresi nell’intervallo teorico, testimoniando così la purezza
55
dell’RNA ottenuto dall’estrazione. Considerando che 1 unità di assorbanza a
260 nm equivale a 40 µg/ml di RNA è stato possibile calcolare la
concentrazione dell’acido nucleico in soluzione.
Fig. 6. Spettro di assorbimento (210-400 nm) dell’RNA estratto
2.3.2 Verifica dell’integrità dell’RNA estratto
Allo scopo di indagare sull’integrità dell’RNA totale estratto, un’aliquota di
RNA totale è stata assoggettata ad elettroforesi su gel di agarosio. Avvenuta
la migrazione, il gel, dopo colorazione con bromuro di etidio, viene posto al
transilluminatore, assoggettato ad irraggiamento UV e fotografato (Fig. 7).
Dall’analisi degli rRNA si è stabilita l’integrità dell’RNA estratto; infatti le
bande relative ai ribosomiali da 28S e 18S, rispettivamente di taglia 4,8 e 1,8
kb, erano presenti nel giusto rapporto 2:1, a favore dell’rRNA da 28S. Non
era inoltre evidenziabile lo smear in corrispondenza degli esemplari
ribonucleici a basso peso molecolare, confermando che l’RNA si era
mantenuto integro nel corso del suo isolamento.
56
Fig. 7. Campione di 1 µg di RNA totale estratto da tessuto, su gel di agarosio all’1% in TBE 1X e sottoposto
ad elettroforesi. A margine della foto sono indicate le taglie corrispondenti agli RNA ribosomiali da 28S (4,8
kb) e 18S (1,8 kb)
57
2.3.3 Verifica mediante PCR del cDNA sintetizzato
Allo scopo di verificare la qualità del cDNA sintetizzato secondo le modalità
descritte in precedenza, quest’ultimo è stato impiegato quale templato per
un’amplificazione mediante PCR di una sequenza nucleotidica di 116 bp
corrispondente alla beta actina. L’immagine relativa al gel di agarosio dopo
elettroforesi
della
miscela
di
amplificazione
mostra
la
presenza
dell’amplificato di interesse (Fig. 8).
Fig. 8. Elettroforesi su gel di agarosio. Prodotto di amplificazione del cDNA investigato, relativo alla beta
actina
58
2.3.4 Risultati PCR Array
I risultati ottenuti dalle analisi mediante PCR Array, condotti su 5 campioni di
controllo e 5 patologici con abortività ricorrente, sono riassunti nel grafico
(Fig. 9) e nella tabella seguenti (Tab. 6).
Fig. 9. Rappresentazione 3D dei profili di espressione relativamente ai 96 geni presenti su piastra nei
campioni tissutali prelevati da endometrio di donne con gravidanza complicata da abortività ricorrente.
Nella seguente tabella sono riportati i geni la cui espressione risultava variata
nei campioni patologici rispetto ai controlli.
Media !Ct (Ct(GOI) Media Ct (HKG))
Espressione
2^-!Ct
differenziale
Simbolo
gene
Campioni
Campioni
Campioni
patologici
di controllo patologici
Campioni
o
(n.
di volte)
Campioni
patologici
patologici
di controllo /Campioni
Down
regolazione
Campioni
controllo
59
Up-
di /Campioni
controllo
di
BMP2
12,93
13,58
1,3E-04
8,2E-05
1,57
1,57
BMP4
10,07
9,77
9,3E-04
1,1E-03
0,81
-1,23
BMP6
9,13
9,45
1,8E-03
1,4E-03
1,25
1,25
BMP7
10,15
12,44
8,8E-04
1,8E-04
4,90
4,90
CCL11
15,11
13,76
2,8E-05
7,2E-05
0,39
-2,55
CCL19
14,32
8,68
4,9E-05
2,4E-03
0,02
-49,86
CCL2
7,02
6,15
7,7E-03
1,4E-02
0,55
-1,83
CCL20
12,54
9,25
1,7E-04
1,6E-03
0,10
-9,79
CCL21
8,46
10,17
2,8E-03
8,7E-04
3,26
3,26
CCL22
11,54
9,42
3,4E-04
1,5E-03
0,23
-4,35
CCL24
13,90
10,95
6,5E-05
5,1E-04
0,13
-7,72
CCL3
10,14
8,81
8,9E-04
2,2E-03
0,40
-2,52
CCL5
9,01
7,82
1,9E-03
4,4E-03
0,44
-2,28
CCL7
14,80
13,76
3,5E-05
7,2E-05
0,48
-2,06
CCL8
12,41
10,34
1,8E-04
7,7E-04
0,24
-4,21
CD40LG
14,54
11,07
4,2E-05
4,7E-04
0,09
-11,11
CSF1
8,37
7,30
3,0E-03
6,3E-03
0,48
-2,09
CSF2
15,06
13,76
2,9E-05
7,2E-05
0,41
-2,47
CSF3
13,25
13,76
1,0E-04
7,2E-05
1,42
1,42
CX3CL1
11,61
10,15
3,2E-04
8,8E-04
0,36
-2,74
CXCL1
9,29
8,39
1,6E-03
3,0E-03
0,54
-1,86
IFNA2
14,18
13,76
5,4E-05
7,2E-05
0,75
-1,34
IFNG
14,19
11,94
5,3E-05
2,5E-04
0,21
-4,76
IL11
13,64
11,29
7,8E-05
4,0E-04
0,20
-5,10
60
IL13
15,20
13,76
2,7E-05
7,2E-05
0,37
-2,71
IL16
12,06
10,83
2,3E-04
5,5E-04
0,43
-2,35
IL17A
15,20
13,76
2,7E-05
7,2E-05
0,37
-2,71
IL17F
15,20
13,76
2,7E-05
7,2E-05
0,37
-2,71
IL18
9,91
7,81
1,0E-03
4,5E-03
0,23
-4,27
IL1B
11,42
11,29
3,6E-04
4,0E-04
0,91
-1,10
IL1RN
10,62
11,05
6,3E-04
4,7E-04
1,35
1,35
IL2
14,66
13,76
3,9E-05
7,2E-05
0,54
-1,87
IL23A
11,81
9,88
2,8E-04
1,1E-03
0,26
-3,81
IL24
10,89
8,52
5,3E-04
2,7E-03
0,19
-5,18
IL27
13,33
11,94
9,7E-05
2,5E-04
0,38
-2,63
IL4
15,20
13,76
2,7E-05
7,2E-05
0,37
-2,71
IL5
12,28
10,63
2,0E-04
6,3E-04
0,32
-3,14
IL6
13,12
12,82
1,1E-04
1,4E-04
0,81
-1,24
IL7
13,48
10,84
8,7E-05
5,5E-04
0,16
-6,24
IL8
9,04
8,96
1,9E-03
2,0E-03
0,94
-1,06
IL9
15,20
13,76
2,7E-05
7,2E-05
0,37
-2,71
LTA
14,16
12,72
5,5E-05
1,5E-04
0,37
-2,71
MIF
1,63
1,05
3,2E-01
4,8E-01
0,67
-1,50
NODAL
13,93
11,66
6,4E-05
3,1E-04
0,21
-4,83
OSM
14,06
13,23
5,9E-05
1,0E-04
0,56
-1,78
PPBP
9,70
12,46
1,2E-03
1,8E-04
6,75
6,75
THPO
12,71
11,91
1,5E-04
2,6E-04
0,58
-1,74
TNFRSF11B 10,57
10,24
6,6E-04
8,3E-04
0,79
-1,26
TNFSF10
8,13
2,8E-03
3,6E-03
0,80
-1,25
8,46
61
TNFSF13B
12,13
11,34
2,2E-04
3,9E-04
0,58
-1,73
VEGFA
7,60
6,92
5,1E-03
8,3E-03
0,62
-1,61
Tab. 6. Elenco dei geni sia up- che down-regolati nei campioni tissutali prelevati da endometrio di donne
con gravidanza complicata da abortività ricorrente.
62
L’espressione di un determinato gene è stata considerata significativamente
alterata solo se, una volta normalizzata rispetto alla "-actina, il rapporto tra
l’espressione nel patologico e l’espressione nel controllo risultava maggiore o
uguale a 1,5 (geni up-regolati) o inferiore o uguale a 0,5 (geni down-regolati).
Nei campioni di endometrio di donne con gravidanza complicata da abortività
ricorrente, applicando tale cut-off sono stati pertanto messi in evidenza 33
geni, di cui 4 up-regolati e 29 down-regolati (Fig. 10).
Fig. 10. Risultati PCRarray in campioni normali e patologici.
Profilo d’espressione di geni codificanti per citochine e chemochine. I valori
sono stati normalizzati rispetto alla "-actina. L’orientamento della freccia
indica l’up-regolazione (freccia su) o la down-regolazione (freccia giù) del
gene.
63
2.3.5 Risultati Real-Time PCR
Nell'ambito dei 33 geni, a carico dei quali era stata rilevata un’alterazione
nell’espressione nei campioni patologici rispetto ai controlli, ne sono stati
selezionati 6 (BMP7, CD40LG, CCL19, CCL20, CCL21, PPBP) per la
valutazione mediante Real-Time PCR.
L’attendibilità dei risultati ottenuti mediante l’analisi PCR Array è stata
verificata, prendendo in esame un numero più elevato di campioni (12
controlli e 12 patologici). Le analisi di Real-Time PCR successivamente
condotte hanno confermato i dati ottenuti mediante la tecnica del PCR Array e
hanno consentito una valutazione quantitativa più accurata delle alterazioni
nell’espressione preliminarmente osservate.
I risultati ottenuti evidenziano un aumento dell’espressione a carico dei geni
BMP7, CCL21, e PPBP. I geni CCL19, CCL20 e CD40LG risultano invece
down-regolati (Tab.7 e 8).
I risultati ottenuti vengono mostrati come "Percentuale di espressione",
assumendo, per comodità, che la percentuale di espressione nel gruppo di
controllo sia pari al 100%.
!
Geni
Percentuale di espressione
BMP7
1163
CCL21
256
PPBP
220
Tab.7 Percentuale di espressione dei geni up-regolati
Geni
Percentuale di espressione
CCL19
49
CCL20
58
CD40LG
71
Tab.8 Percentuale di espressione dei geni down-regolati
!
64
!
L’analisi statistica condotta sui risultati ottenuti mediante Real-Time PCR ha
dimostrato che differenze di espressione tra campioni di controllo e campioni
patologici sono statisticamente significative per quanto riguarda i geni BMP7,
CCL19, CCL21, e CD40LG (* p< 0.05). Non statisticamente significativa è
risultata invece essere la differenza nei livelli di espressione tra campioni di
controllo e campioni patologici per i geni CCL20 e PPBP (Fig.11 e 12).
!
!
!
!
!
!
!
!
!
!
!
!
!
!
65
!
!
!
Fig.11 Percentuale di espressione dei geni up-regolati BMP7, CCL21 e PPBP.
!
!
!
!
!
!
!
!
!
!
!
66
!
!
!
Fig.12 Percentuale di espressione dei geni down-regolati CCL19, CCL20 e CD40LG.
67
2.4 Discussione
Analizzeremo dapprima singolarmente le singole citochine risultate ad
espressione aumentata o diminuita tra i campioni di pregressa abortività
ricorrente in esame ed i controlli.
GENI UP-REGOLATI
BMP2
Le bone morphogenetic proteins (BMPs) appartengono alla famiglia del
fattore di crescita trasformante tipo !
(TGF!) e svolgono funzioni
pleiotropiche non solo durante l’embriogenesi, ma anche dopo la nascita:
sono essenziali per lo sviluppo del cuore, del sistema nervoso, della
cartilagine e nello sviluppo postnatale dell’osso. Queste proteine promuovono
infatti la differenziazione delle cellule mesenchimali in condrociti e
osteoblasti, che sono i protagonisti dello sviluppo e del metabolismo
dell’osso. Tra i vari membri della famiglia delle BMPs, le BMP2, BMP4,
BMP6 e BMP7 hanno dimostrato la potenzialità di indurre la formazione di
osso ectopico, qualora impiantato nel tessuto muscolare. Infatti le BMPs
inibiscono la differenziazione delle cellule mioblastiche in cellule muscolari
mature, deviando il percorso di maturazione verso il fenotipo osteoblastico.
In particolare, BMP2 regola la funzione delle proteine Gremlin 1 e Gremlin 2,
che sono degli antagonisti della funzione delle BMPs, down-regolando
l’espressione di Gremlin 1 e up-regolando l’espressione di Gremlin 2 con il
risultato finale di indurre la differenziazione osteoblastica.
In sostanza, BMP2 inibisce l’espressione di Grem1 che viene indotta durante
la differenziazione mioblastica; mentre attiva l’espressione di Grem2 che
68
viene indotta durante la differenziazione osteoblastica. La regolazione
dell’espressione genica di Grem 1 e Grem2 può spiegare le fondamentali
funzioni di questi geni durante la differenziazione osteoblastica [134]. BMP2
modula anche le popolazioni cellulari dei sinoviociti, in particolar modo in
caso di sinovite, poiché i livelli di BMP2 vengono incrementati dalle
citochine pro infiammatorie e infatti riscontrati in caso di artrite reumatoide e
spondiloartrite [135].
BMP2 inoltre promuove la proliferazione, la differenziazione e l’apoptosi
delle cellule uterine del ratto durante il ciclo mestruale e molte BMPs sono
espresse nella placenta umana [136-138].
A livello del sistema nervoso centrale, BMP2, in sinergia con LIF, agisce
sulle cellule progenitrici neurali fetali per indurre la crescita degli astrociti
[139] e per determinare il destino della cellula staminale neurale in senso
simpatico o parasimpatico [140]. A livello delle cellule staminali neurali,
BMP2 agisce sopprimendone la differenziazione e mantenendone quindi la
multipotenzialità [141].
A livello del timo, BMP2 esplica la propria azione poichè nel timo sono
espressi i recettori per questa proteina, che riveste quindi un ruolo anche nella
differenziazione delle cellule T [142].
Infine BMP2 e BMP4 regolano il processo di rigenerazione del follicolo
pilifero a livello dermico, mostrando, anche in questo caso, un’azione
sull’omeostasi delle cellule staminali locali dell’organo da parte del
macroambiente circostante. Da sottolineare il fatto che l’espressione di BMP2
nel tessuto cutaneo, così come negli adipociti, indica una fisiologica
integrazione tra i due organi coinvolti nella termoregolazione [143].
69
BMP7
BMP7 è un membro della superfamiglia del fattore di crescita trasformante
!(TGF!). La sua funzione è quella di stimolare la crescita delle cellule
dendritiche nei neuroni centrali e periferici [144]. Infatti i geni che trascrivono
per BMPs, per gli antagonisti di BMP e per i recettori di BMPs sono espressi
nel cervello dell’adulto e trascrivono per proteine implicate in vari aspetti
dello sviluppo del sistema nervosa. Per esempio BMP7 regola la migrazione
neuronale, riduce gli effetti dell’infarcimento cerebrale indotto da ischemia,
promuove la sopravvivenza cellulare nelle cellule granulocitarie del
cervelletto e ha una funzione neuroprotettiva nelle cellule corticali. La perdita
di BMP7 nel topo causa difetti nello sviluppo dello scheletro, del rene, del
palato e dei denti e provoca morte neonatale a causa dell’uremia dovuta a
malfunzionamento renale [145].
Le proteine TGF! hanno mostrato anche diverse funzioni a livello della
cornea: per esempio, l’attivina A e TGF!1 hanno dimostrato un’attività di
regolazione nei confronti della differenziazione dei cheratociti e potrebbero
avere un ruolo durante la trans differenziazione miofibroblastica; ma ciò non
risulta vero per quanto riguarda BMP7 [146].
BMP7 ha però dimostrato, come gli altri membri della famiglia delle BMP, la
capacità di stimolare tutti i marker di differenziazione osteoblastica nelle
cellule staminali mesenchimali e pluripotenti, ma risulta meno potente rispetto
ad altre BMP, quali BMP2, BMP6 e BMP9 [147].
Nel tessuto cardiaco, BMP7 agisce preservando il fenotipo endoteliale,
inibendo cioè la transizione endotelio-mesenchimale e la conseguente
progressione della fibrosi cardiaca, al contrario del TGF! che svolge invece
un’azione profibrotica [148].
70
Infine BMP7 attiva il programma di adipogenesi del tessuto adiposo bruno
promuovendo la sua differenziazione e la termogenesi e candidandosi quindi
come un potenziale nuovo approccio terapeutico nel trattamento dell’obesità
[149].
Nel ricercare una correlazione specifica tra patologie ostetriche e
iperespressione delle proteine BMP, sono emersi in particolare alcuni studi. Il
primo è uno studio cinese [150], che si prefiggeva di analizzare la relazione
tra BMP2 e la rottura prematura delle membrane (PROM). In questo studio
l’espressione di BMP2 nella rottura prematura delle membrane a termine
risultava più alta che nel gruppo con travaglio a termine e l’espressione nella
rottura prematura delle membrane pretermine risultava più alta che nel gruppo
con travaglio pretermine. Lo studio conclude dunque che l’espressione di
BMP2 nelle membrane corioamniotiche sia strettamente correlato alla PROM.
Questi risultati sono in sintonia con quelli di un altro studio [151] che,
partendo dal presupposto che gli studi genici delle membrane corioamniotiche
avevano inaspettatamente riscontrato un incremento dell’espressione della
BMP2 nel travaglio spontaneo di parto a termine, si prefiggeva di determinare
se l’mRNA di BMP2 e l’espressione proteica fosse alterata nelle membrane
corioamniotiche di pazienti con travaglio a termine, travaglio pretermine e
rottura prematura pretermine delle membrane (pPROM). Il risultato è che
l’mRNA di BMP2 è significativamente più alto nelle membrane di pazienti
con travaglio a termine che nelle membrane di pazienti non in travaglio e che
è incrementato in pazienti con travaglio pretermine e riscontri istologici di
corioamnionite rispetto a pazienti senza corioamnionite; infine non risultava
una differenza statisticamente significativa nell’espressione dell’mRNA di
BMP2 in pazienti con pPROM, a prescindere dalla corioamnionite.
71
Kodama et al. [152] hanno invece valutato l’espressione di BMP7 a livello
endometriale ed hanno studiato il suo ruolo sulla decidualizzazione e
proliferazione delle cellule endometriali stromali, suggerendone un ruolo
chiave in questi processi [153].
CCL21
CCL21 agendo attraverso il recettore CCR7, ricopre un ruolo essenziale nel
veicolare i linfociti naive e le cellule dendritiche ai tessuti linfoidi secondari
[154]. CCL21 è infatti una chemochina che svolge la funzione fondamentale
della chemiotassi; viene prodotta da cellule che sono disseminate all’interno
delle aree extrafollicolari e agisce tramite il recettore CCR7, che viene
specificamente espresso dalle cellule T naive, da alcune cellule T della
memoria, dalle cellule B e dalle cellule dendritiche mature. Durante la
chemiotassi delle cellule B e T, il citoscheletro viene rimodellato
dinamicamente e questa riorganizzazione produce la forza necessaria per
l’attivazione e la migrazione di queste cellule [155]. Le cellule B, per
esempio, richiamate dalle chemochine, vengono attirate verso le zone T al
fine di interagire con le cellule T helper [156]. Il percorso seguito dai linfociti,
che è critico per l’efficacia della risposta immunitaria, è infatti strettamente
regolato dall’espressione di molecole di adesione e recettori della
chemioattrazione espresso sui linfociti, in combinazione con l’espressione dei
ligandi per questi recettori espressi di volta in volta da una varietà di cellule
tissutali diverse. Le cellule B esprimono diversi recettori per le chemochine,
inclusi CXCR4, CXCR5, CCR6 e CCR7 e rispondono ai loro ligandi
corrispettivi CXCL12, CXCL13, CCL20 e CCL21 [157].
L’importanza delle chemochine però non risiede solamente nel richiamare le
cellule immunitarie da un sito ad un altro, ma anche nel contribuire allo
72
sviluppo dei tessuti linfoidi e ad iniziare ed espandere la risposta immune
adattativa [158].
Alcuni recettori delle chemochine hanno mostrato un importante ruolo nelle
metastasi da tumori: le chemochine CCL19, CCL20 e CCL21 e i loro recettori
CCR6 e CCR7 sono stati accertati come potenziali biomarker di
disseminazione metastatica nel tumore primitivo della mammella [159]. Studi
simili sono stati svolti anche per quanto riguarda il tumore della laringe, della
testa e del collo. Per quanto riguarda il melanoma, studi condotti sui topi
[160] hanno rivelato che l’espressione di CCL21 da parte del tumore era
associata ad un’immunotolleranza del microambiente, caratterizzato da
induzione di uno stroma reticolare simil-linfocitario, alterato milieu
citochinico e reclutamento dei popolazioni leucocitarie. Questa scoperta
permetteva di ipotizzare che, alterando il microambiente tumorale, i tumori
secernenti CCL21 decretavano uno shift della risposta immunitaria dell’ospite
da immunogena ad immunotollerante, in modo da facilitare la progressione
tumorale.
Tornando ad analizzare il reclutamento al sito dell’infiammazione delle
cellule T effettrici mediato dalle chemochine, sappiamo che esso rappresenta
un carattere centrale della risposta immunitaria. L’accumulo delle cellule T
effettrici però non è permesso all’interno della decidua, in parte proprio a
causa del silenziamento epigenetico dei geni che trascrivono per le
chemochine infiammatorie nelle cellule stromali della decidua. Queste
scoperte gettano luce sul meccanismo dell’immunotolleranza feto materna e
rivelano le modificazioni epigenetiche che avvengono nelle cellule stromali al
fine di limitare il reclutamento di cellule T effettrici [161].
Risulta infatti che, al contrario, in caso di villite di origine sconosciuta, una
lesione infiammatoria che distrugge i villi della placenta, si sviluppi una
73
iperespressione degli mRNA che trascrivono per le chemochine e i loro
recettori (CXCL9, CXCL10, CXCL11, CXCL13, CCl4, CCL5, CXC3, CCR5
[162] .
PPBP (Pro-Platelet Basic Protein)
Pro-platelet basic protein è il precursore di due proteine, platelet basic protein
(PBP) e connective tissue-activating peptide III (CTAP3). Al momento
dell’attivazione delle piastrine, essi vengono rilasciati e ulteriormente
processati nel plasma in beta-thromboglobulin (TGB) e neutrophil-activating
peptide-2 (NAP2).
CTAP3 è un fattore di crescita derivato dalle piastrine che stimola una varietà
di attività cellulari e metaboliche specifiche, incluse la capacità di indurre
mitosi, la sintesi della matrice extracellulare, il metabolismo del glucosio, la
sintesi dell’attivatore del plasminogeno nelle culture di fibroblasti [163,164].
GENI DOWN-REGOLATI
Chemochine
Le chemochine sono proteine coinvolte nei processi infiammatori e di
immunoregolazione. Le chemochine sono proteine di 8-12 kD con 4 residui
cisteinici conservati in tutti i membri della famiglia. La famiglia delle
chemochine è suddivisa in base a dove sono localizzati i primi due residui
cisteinici: se sono semparati da un singolo amminoacido, appartengono alla
famiglia " e vengono chiamati C-X-C; se invece sono adiacenti l’uno
all’altro, appartengono alla famiglia ! e vengono chiamati C-C. I membri
della famiglia " sono organizzati in un cluster all’interno del cromosoma 21;
mentre i membri della famiglia ! sono organizzati in un cluster all’interno del
74
cromosoma 32. Il principale ruolo delle chemochine è quello di regolare
l’infiltrazione leucocitaria all’interno di un tessuto infiammato. Alcune
chemochine, come ad esempio MIP1" inibiscono anche la proliferazione dei
progenitori delle cellule ematopoietiche.
Il ruolo delle chemochine nel ciclo mestruale e nell’impianto embrionale è
stato ampiamente studiato [165-169].
CCL19
oltre
all’azione
esplicata
nell’infiammazione
e
nell’immunoregolazione, CCL19 potrebbe agire anche nel normale ricircolo
ed homing dei linfociti: infatti determina la migrazione dei linfociti T verso il
timo e dei linfociti B verso gli organi linfoidi secondari [156]. È un potente
chemioattrattore dei linfociti T e B, ma non dei granulociti e dei macrofagi.
CCL19 è coinvolta anche nella migrazione delle cellule dendritiche dalla cute
ai linfonodi, poiché media l’azione del leucotriene C4 sul suo recettore MRP1
[170].
Il suo recettore specifico è CCR7 (lo stesso a cui si lega CCL21, la citochina
che abbiamo visto essere up-regolata nell’aborto spontaneo).
CCL19 viene espressa abbondantemente nel timo e nei linfonodi, in modo
moderato nella trachea e nel colon, e a bassi livelli nello stomaco,
nell’intestino tenue, nel polmone, nel fegato e nella milza.
CCL19, infatti, insieme a CXCL13 e CCL21, viene costitutivamente espresso
negli organi linfoidi secondari ed è inducibilmente espresso nel polmone in
seguito ad infezione da parte del virus dell’influenza. In assenza di milza e
linfonodi, l’espressione a livello polmonare dei ligandi di CCR7 (CCL19 e
CCL21) diviene un elemento critico per la risposta immunitaria al virus
dell’influenza. Infatti essi sono essenziali per indurre la formazione di tessuto
linfoide associato al bronco. Dunque CCL19, così come CCL21, viene
espresso nei siti di infiammazione e contribuisce allo sviluppo del tessuto
75
linfoide locale e all’inizio e al protrarsi della risposta immunitaria adattativa
[158].
Il recettore di CCL19, CCR7, è il segnale necessario richiesto perché le
cellule leucemiche T si vadano a localizzare all’interno del sistema nervoso
centrale. Il silenziamento di CCR7 o del suo ligando CC19 quindi inibisce
l’infiltrazione nervosa, potendo rappresentare una nuova strategia terapeutica
mirata [171].
CCL19 però, non lega solamente CCR7, ma anche CCRL2 con un’affinità
comparabile a quella che ha verso CCR7. Ma il legame con CCRL2 non esita
in alcuna attivazione intracellulare, come la mobilizzazione del calcio o la
migrazione. CCRL2 potrebbe quindi rappresentare un recettore coinvolto nel
modulare l’attività di homing dei linfociti svolta da CCL19 [172].
CCL24 ha un’azione chemiotattica sui linfociti T a riposo e sugli eosinofili,
una bassa attività sui neutrofili e nessuna attività sui monociti e sui linfociti T
attivati. CCL24 è inoltre un potente soppressore dei fattori di crescita formanti
colonie, che rappresentano progenitori ematopoietici multipotenti [173].
La sua attività sugli eosinofili è dimostrata dall’eosinofilia che viene indotta
quando si inietta CCL24 intradermica. Anche i basofili e i neutrofili si
accumulano nel sito di iniezione. Ciò a dimostrazione del fatto che CCL24
induce un infiltrato infiammatorio ed eosinofilo importante e compatibile con
la de granulazione dei mastociti indotta dalla chemochina stessa [174].
CCL24 ha infatti il ruolo di reclutare e attivare le cellule che esprimono il
recettore CCR3, vale a dire gli eosinofili, i mastociti e i linfociti Th2, essendo
quindi uno dei maggiori responsabili dei disordini di tipo allergico. I livelli di
CCL24 risultano infatti incrementati nei pazienti asmatici rispetto ai controlli
sani [175].
76
Per quanto riguarda CCL20, risulta essere iperespressa nella psoriasi. I
cheratinociti che eprimono CCL20 si localizzano nella zona di cute affetta da
psoriasi assieme ai linfociti T, a dimostrazione del fatto che CCL20 è un
fattore chemiotattico per le cellule T. CCL20 viene prodotto nella cute in
risposta a una varietà di mediatori pro infiammatori, inclusi TNF/IL1B,
CD40LG, IFNG e IL17 [176].
CCL20 potrebbe inoltre rappresentare un asse evolutivo conservato che regola
l’ingresso e la disseminazione delle cellule T all’interno del sistema nervoso
centrale [177].
CCL11 è un potente induttore della chemiotassi degli eosinofili, che
comporta migrazione degli eosinofili in vitro e accumulo di essi in vitro.
Questi effetti sono mediati dal recettore CCR3, che è altamente espresso negli
eosinofili, nei basofili e nei linfociti Th2 e quindi coinvolto nella reazione
infiammatoria allergica. CCL11 può agire anche sui monociti tramite i
recettori CCR2 e CCR5 [178].
CCL11 inoltre induce la migrazione delle cellule endoteliali in vitro e
l’angiogenesi in vivo e può dunque contribuire all’angiogenesi nelle
condizioni in cui essa sia accompagnata da infiltrazione di eosinofili, come ad
esempio nel linfoma di Hodgkin, nei polipi nasali, nell’endometriosi e nella
diatesi allergica [179].
A CCL11 può infine essere attribuito il declino della neurogenesi e il
deterioramento delle funzioni cognitive che si osservano con l’avanzare
dell’età [180].
CCL22 è un fattore chemiotattico per le cellule dendritiche e per le cellule
NK attivate da IL2, ma non per i neutrofili [181]. Le concentrazioni di CCL22
risultano molto elevate nel plasma di soggetti infettati dal virus HTLV-1 in
quanto CCL22 determina il ritardo nella progressione della malattia
77
infiammatoria associata ad HTLV-1 e contribuisce all’immunosoppressione
osservata nell’infezione da HTLV-1 [182].
CCL3/MIP-1-", è una chemochina coinvolta nella risposta infiammatoria
acuta e nel reclutamento, in particolare, dei polimorfonucleati. È inoltre uno
dei più potenti fattori soppressivi del virus di HIV secreti dalle cellule T CD8
positive [183].
CCL5/RANTES (“regulated on activation, normal T-cell expressed and
secreted”) è una chemochina molto potente, implicata nei processi di
immunoregolazione e infiammazione dell’artrite reumatoide. Le chemochine
vengono principalmente prodotte nei tessuti periferici a livello dei siti di
infiammazione e rivestono un duplice ruolo nell’infiammazione: in primo
luogo, richiamano i neutrofili, i monociti, le cellule dendritiche immature, le
cellule B all’articolazione infiammata e, in secondo luogo, regolano una
reclutazione differenziale dei linfociti T helper (Th1 e Th2) [184].
Nel caso dell’infezione da virus HIV, sono proprio i linfociti T CD8+ ad
essere coinvolti nel controllo dell’infezione, grazie alla produzione delle
chemochine RANTES, MIP1alfa e MIP1beta, che sono i maggiori fattori HIV
soppressivi. L’effetto delle chemochine sul virus dell’HIV può essere sia
diretto, grazie all’azione anti-lentiretrovirale, o indiretto, grazie alla capacità
di attrarre linfociti T e monociti al sito di infezione. Ma quest’ultimo
meccanismo può anche avere l’effetto opposto di generare nuovi target per
l’HIV [183].
In conseguenza di ciò, si è cercato di investigare se un derivato di RANTES
potesse essere un possibile agente terapeutico nei confronti dell’infezione da
HIV e si è scoperto che il derivato della chemochina mostrava ancora le
capacità di bloccare l’infezione da HIV, pur con dei difetti nelle proprietà di
attivazione del proprio recettore [185].
78
La concentrazione dell’mRNA di RANTES, unica tra le chemochine, risulta
incrementata inoltre anche con l’avanzare dell’età a livello degli astrociti.
RANTES regola infatti la crescita e la sopravvivenza degli astrociti. Ciò a
dimostrazione del fatto che le chemochine non sono solo mediatori
dell’infiammazione,
ma
sono
anche
importanti
regolatori
della
differenziazione durante lo sviluppo [186].
A livello endometriale è stato studiato l’effetto dei ligandi PPAR-gamma
sulla trascrizione e la traduzione di RANTES nelle cellule stromali
endometriali. Tre elementi responsivi a PPAR-gamma sono stati trovati sul
promotore di RANTES, i quali determinano l’inibizione di questo promotore.
L’uso di PPAR-gamma dunque riduce la produzione delle chemochine e la
conseguente infiammazione, cosa che potrebbe fungere da razionale per una
futura strategia terapeutica nei confronti di patologie endometriali, quali
l’endometriosi [187].
Nel caso del fegato, l’espressione di CCL5 risulta bassa nel fegato normale,
mentre risulta incrementata significativamente in seguito ad infezione da virus
dell’epatite C, in particolare nelle aree che istologicamente risultano infiltrate
da linfociti [188].
Il ruolo di CCL5 si esplica non solo nel caso di infezione da virus HCV, ma
anche nel corso di altre infezioni virali, in quanto le cellule infettate dal virus
devono essere rimosse da parte dei linfociti citotossici e questa rimozione
richiede che i macrofagi non vengano a loro volta uccisi dal virus. Questo
processo che impedisce l’apoptosi dei macrofagi è mediato proprio da CCL5,
tramite l’attivazione del recettore CCr5 e la conseguente attivazione della via
intracellulare proteina G"-PI3K-AKT e proteina G"-MEK-ERK, tant’è vero
che topi knock-out per il gene CCL5 dimostrano una clearence virale
ritardata, un’eccessiva infiammazione delle vie aeree e vanno più facilmente
79
incontro a morte in seguito ad infezione da parte del virus della parainfluenza
murina o dell’influenza umana [189].
CCL5 risulta coinvolto, infine, anche nel processo di metastatizzazione del
tumore della mammella. È risultato infatti che le cellule staminali
mesenchimali di derivazione dal midollo osseo incrementano la capacità
metastatica delle cellule tumorali in quanto secernono CCL5 che potenzia la
motilità, la capacità invasiva e la capacità metastatica delle cellule tumorali
[190].
CCL7 attrae specificamente i monociti e regola la funzione macrofagica.
Viene prodotta da alcune linee cellulari tumorali e dai macrofagi.
CCL8 è un fattore chemiotattico per svariati tipi di cellule immunitarie,
inclusi i mastociti, gli eosinofili e i basofili (che sono implicati nella risposta
allergica), i monociti, le cellule T e le cellule NK. Esplica il suo effetto
legandosi a una serie di differenti recettori cellulari sulla superficie delle
diverse cellule, inclusi CCR1, CCR2B e CCR5 [191]. È anch’esso un potente
inibitore di HIV poiché lega con alta affinità il recettore CCR5, uno dei più
importanti co-recettori di HIV [192].
CX3CL1 rappresenta una classe unica di chemochine e può costituire parte
del controllo del traffico leucocitario all’endotelio [193]. Viene infatti
particolarmente
espressa
nell’aterosclerosi
a
livello
delle
placche
ateromasiche delle arterie coronarie, dove agisce da mediatore della
migrazione delle cellule muscolari lisce, più che da mediatore del
reclutamento delle cellule infiammatorie [194]. L’espressione di CX3CL1 è
stata accertata anche in campioni di tessuto endometriale, lungo tutto il ciclo
mestruale, durante la fase precoce della gravidanza e nelle donne che
utilizzano contraccettivi esclusivamente progestinici. CX3CL1 è localizzato
prevalentemente nell’epitelio ghiandolare e nelle cellule stromali della
80
decidua, soprattutto durante la fase secretoria del ciclo mestruale e nella
gravidanza iniziale. I leucociti che esprimono sulla loro membrana il recettore
per CX3CL1 (macrofagi e neutrofili) erano particolarmente abbondanti
durante la fase mestruale. Dunque CX3CL1 riveste certamente un ruolo di
primaria importanza nell’endometrio, nella fase secretoria, nella fase iniziale
della gravidanza e nelle donne che utilizzano contraccettivi progestinici [195].
81
Interleuchine
Le interleuchine sono un gruppo di citochine secrete da cellule del sistema
immunitario (linfociti, cellule NK, fagociti, cellule dendritiche) e, in alcuni
casi, anche da cellule endoteliali e cellule epiteliali, durante la risposta
immunitaria. Esse promuovono lo sviluppo e la differenziazione delle cellule
T, B ed ematopoietiche. Grazie alla secrezione di interleuchine, le cellule del
sistema immunitario possono regolare l'attività di altre cellule, innescando
uno dei più importanti meccanismi di comunicazione cellulare a livello del
sistema immunitario. Il loro nome infatti deriva proprio da “inter”, che sta ad
indicare la comunicazione che vanno a stabilire tra le varie cellule, e “leukin”,
poiché vengono prodotte dai leucociti e agiscono sui leucociti. La loro azione
può essere autocrina, paracrina e, in rari casi, endocrina.
IL 11 è una proteina che stimola la megacariocitopoiesi, con conseguente
incremento della funzione piastrinica, anche in seguito a trombocitopenia
indotta da chemioterapici. Ha inoltre svariati altri effetti, come quello di
indurre la produzione di proteine della fase acuta, modulare la risposta
antigene-anticorpo, partecipare alla regolazione della proliferazione e della
differenziazione degli osteociti, tanto che potrebbe essere utilizzata come
terapia dell’osteoporosi. In aggiunta, agisce anche su molti altri tessuti, inclusi
cervello, intestino e testicolo [196]. Alcune tra le sue funzioni più importanti
includono la formazione della placenta e della decidua: infatti IL 11 ha un
importante ruolo durante l’impianto della blastocisti nell’endometrio
dell’utero; si è infatti dimostrato che IL 11 viene espressa sia dalla decidua
che dai villi coriali per regolare fino a che punto la placenta debba estendersi
all’interno dell’utero, al fine di garantire il benessere della madre, ma anche la
sopravvivenza del feto [197,198].
82
IL 13 è una citochina con funzioni pleiotropiche che potrebbe essere
importante nella regolazione della risposta infiammatoria ed immunitaria.
Inibisce la produzione di citochine infiammatorie e agisce sinergicamente con
IL 2 nel regolare la sintesi di IFN#. In aggiunta a queste funzioni, IL 13 è
soprattutto implicato come mediatore centrale dei fisiologici cambiamenti
indotti in molti tessuti dall’infiammazione di origine allergica: IL 13 può
indurre la sintesi di IgE dalle cellule B attivate, ma più che agire come una
citochina nei confronti dei linfociti, IL 13 prevalentemente funge da ponte
molecolare tra le cellule della reazione infiammatoria allergica e le cellule di
natura non immune. Nonostante sia soprattutto coinvolta nell’innescare le
patologie delle vie aeree, ha anche proprietà antinfiammatorie: le MMPs delle
vie aeree vengono stimolate da IL 13 come parte di un meccanismo che
protegge le vie aeree contro un’eccessiva infiammazione [199]. L’espressione
genica dell’IL13 a livello endometriale è stata studiata da Chegini et al. in
donne con pregressa abortività spontanea, risultando elevata rispetto ai
controlli [200].
IL 16 è anch’essa una citochina pleiotropica che chemioattrattore per le
cellule che esprimono CD4 (linfociti, monociti, eosinofili e cellule
dendritiche), modulatore dell’attivazione dei linfociti T e inibitore della
replicazione di HIV (il segnale di questa interleuchina è infatti mediato da
CD4) [201].
IL 17 è una citochina che agisce da potente mediatore delle reazioni
immunitarie di tipo ritardato, incrementando la produzione di chemochine in
vari tessuti per reclutare monociti e neutrofili al sito di infiammazione, con
azione simile a quella dell’IFN#. IL 17 è prodotta dalle cellule Thelper in
risposta all’invasione dell’organismo da parte di un patogeno extracellulare al
fine di distruggere la matrice cellulare del patogeno [202]. Le cellule T che
83
producono IL17 sono coinvolte nei processi infiammatori cronici ed alcuni
autori hanno anche valutato il loro ruolo negli aborti spontanei ricorrenti
[203].
IL 18 è una citochina che appartiene alla famiglia delle IL 1 ed è prodotta dai
macrofagi. Grazie al legame con IL 18R, e in sinergia con IL 2, stimola
l’immunità cellulo-mediata in seguito ad un’infezione con prodotti microbici,
come LPS. Dopo stimolazione da parte di IL 18, le cellule NK e le cellule T
rilasciano un’altra importante citochina, IFN# che andrà ad attivare i
macrofagi. Inoltre IL 18 inibisce la produzione di IgE e IgG1 mediata da IL 4.
E’ documentato un suo coinvolgimento, sia come polimorfismo che come
ridotto livello sierico, nelle donne con abortività spontanea ricorrente [204] e
tale dato è conforme con i nostri dati.
IL 23 è una parte importante della risposta infiammatoria che si innesca
contro le infezioni. Promuove l’up-regulation della MMP9, incrementa
l’angiogenesi e riduce l’infiltrazione delle cellule T CD8 positive. Inoltre
stimola le cellule T CD4 positive naive a differenziarsi in un nuovo sottotipo
cellulare, chiamato Th17, che è distinto da Th1 e Th2 e produce IL 17 [205].
IL 24 controlla la sopravvivenza e la differenziazione cellulare, inducendo
una rapida attivazione dei fattori di trascrizione Stat-1 e Stat-3. È secreta da
monociti attivati, macrofagi e cellule Th2 e agisce su tessuti non
ematopoietici come la cute, il polmone e i tessuti riproduttivi. Ha un ruolo
importante nella guarigione delle ferite, nella psoriasi e nel cancro. Molti
studi hanno infatti dimostrato che l’apoptosi delle cellule tumorali è
conseguente a esposizione a IL 24 [206].
IL 27 riveste un ruolo importante nel regolare l’azione dei linfociti B e T.
IL 4 induce la differenziazione delle cellule Th naive in cellule Th2, che
dunque iniziano a secernere IL 4. Oltre a questo, ha molti altri ruoli biologici,
84
inclusa la stimolazione della proliferazione delle cellule B e T attivate e la
differenziazione delle cellule B in plasmacellule, inducendo lo switch in IgE.
Regola la risposta immunitaria umorale e adattativa. Riduce la produzione di
cellule Th1, macrofagi, IFN# e cellule dendritiche. La sua overespressione è
associata a sviluppo di allergie [207].
IL 5 è un’interleuchina prodotta dai linfociti Th2 e dai mastociti. Le sue
funzioni sono di stimolare la crescita delle cellule B e aumentare la secrezione
di immunoglobuline. È anche un mediatore chiave dell’attivazione degli
eosinofili. Clinicamente è considerato causa di molte malattie allergiche,
come la rinite allergica e l’asma [208].
IL 7 stimola la differenziazione delle cellule staminali ematopoietiche multi
potenti nei progenitori delle cellule linfoidi (al contrario dei progenitori
mieloidi, la cui differenziazione è stimolata da IL3).
IL 9 è specificamente prodotta dai linfociti T CD4 positivi e agisce da
regolatore di una varietà di cellule ematopoietiche. Stimola la proliferazione
cellulare e previene l’apoptosi. Questo gene è stato identificato come un gene
candidato per lo sviluppo dell’asma, poiché è determinante per lo sviluppo di
iperresponsività bronchiale [209].
Altre citochine
CD40LG codifica per una proteina che viene espressa sulla superficie delle
cellule T e regola la funzione delle cellule B, legandosi a CD40 espresso sulla
superficie dei linfociti B. Un difetto nell’espressione di questo gene determina
un’incapacità a fare lo switch tra le varie classi immunoglobuliniche ed è
associato a sindrome da iper-IgM. Media la proliferazione delle cellule B ed è
appunto coinvolto nello switch delle catene immunoglobuliniche [210,211].
85
Il rilascio di CD40L solubile da parte delle piastrine è in parte regolato da GP
IIb/IIIa e potrebbe dunque essere utilizzato come marker nei pazienti con
sindrome coronarica acuta.
CSF 1 è il macrophage colony-stimulating factor, o M-CS; è un fattore di
crescita ematopoietico che stimola le cellule staminali ematopoietiche a
differenziarsi in macrofagi, quindi è coinvolto nella proliferazione,
differenziazione e sopravvivenza dei monociti, dei macrofagi e delle cellule
progenitrici degli osteociti. A livello osseo stimola gli osteoclasti e determina
quindi innalzamento dei livelli di calcio plasmatici. Può anche essere
coinvolto nello sviluppo della placenta: alti livelli di CSF 1 infatti sono
osservati nell’epitelio dell’endometrio dell’utero gravidico, così come alti
livelli del suo recettore CSF1R vengono espressi nel trofoblasto. Studi hanno
dimostrato che l’attivazione di CSFR1 nel trofoblasto da parte di alti livelli
locali di CSF 1 è essenziale per il normale impianto dell’embrione e per lo
sviluppo della placenta [212]. Più recentemente è stato dimostrato che CSF-1
e CSF1R sono implicati nello sviluppo della ghiandola mammaria durante il
normale sviluppo e durante la crescita neoplastica [213].
CSF 2 è il granulocyte-macrophage colony-stimulating factor (GM-CSF); è
un fattore di crescita per i granulociti e macrofagi: CSF 2 stimola le cellule
staminali a produrre granulociti (neutrofili, eosinofili e basofili) e monociti. I
monociti escono dal circolo e migrano nei tessuti, dove poi matureranno in
macrofagi e cellule dedritiche. Rappresenta una parte della cascata
infiammatoria /immune, grazie alla quale l’attivazione di un piccolo numero
di macrofagi può rapidamente portare a aumento del loro numero, un
proocesso cruciale per sconfiggere le infezioni.
IFNG codifica per IFN#, una citochina critica per la risposta immunitaria
innata e adattativa contro le infezioni virali e batteriche intracellulari e per il
86
controllo dei tumori. L’importanza di IFN# nel sistema immunitario è dovuta
in parte alla sua abilità nel’impedire la replicazione virale in modo diretto, ma
soprattutto ai suoi effetti immunostimolanti e immunomodulatori.
INF# è prodotto soprattutto dalle cellule NK, come parte della risposta innata,
e dalle cellule Th1 CD4 positive e CD8 positive, una volta che la risposta
immunitaria antigene-specifica si sia instaurata.
Al contrario di IFN" e IFN! che possono essere espressi da tutte le cellule,
IFN# è secreto dalle cellule Th1, dalle cellule T citotossiche e dalle cellule
NK.
Le sue principali funzioni sono antivirale, immunoregolatoria e antitumorale e
vengono esplicate alterando la trascrizione di 30 geni e producendo così una
varietà di effetti fisiologici e di risposte cellulari. Tra gli effetti più importanti:
promuove l’attività delle cellule NK, incrementa la presentazione antigenica e
l’attività lisosomica dei macrofagi, attiva l’ossido nitrico sintasi inducibile,
induce la produzione di IgG2 e IgG3 dalle cellule B attivate, promuove la
differenziazione Th1 e sopprime la differenziazione Th2, stimola le cellule
normali ad aumentare l’espressione di MHCI e le cellule presentanti
l’antigene ad aumentare l’espressione di MHCII, promuove l’adesione
richiesta per la migrazione leucocitaria, induce l’espressione di fattori di
difesa intrinseci [214].
LTA codifica per la proteina Lymphotoxin-alpha, un membro della famiglia
dei tumor necrosis factor. È una citochina prodotta dai linfociti, altamente
inducibile, che media un’ampia gamma di risposte infiammatorie,
immunostimolanti ed antivirali. È anche coinvolta nella formazione degli
organi linfoidi secondari durante lo sviluppo e gioca un importante ruolo
nell’apoptosi, tanto che topi knock out per LTA non sviluppano le placche di
Peyer né i linfonodi [215].
87
NODAL codifica per una proteina membro della superfamiglia TGF!. Come
molti altri membri di questa superfamiglia, è coinvolto nella differenziazione
cellulare. È essenziale per la formazione del mesoderma e per la conseguente
organizzazione delle strutture lungo l’asse mediano nel corso dello sviluppo
embrionale [216]. Infatti l’asimmetria destra-sinistra delle vertebre nel corpo
è determinata da un processo che utilizza la proteina NODAL. NODAL è
espresso dal lato sinistro dell’organismo nelle fasi iniziali dello sviluppo. La
non espressione di NODAL comporta l’assenza del primitivo scheletro assile
e il fallimento della formazione del mesoderma, con conseguente arresto dello
sviluppo appena dopo la gastrulazione [217]. NODAL è una citochina
ampiamente distribuita: non è limitata solo ai vertebrati.
Nel nostro studio è stato analizzato il profilo di espressione genica di
citochine infiammatorie nel tessuto endometriale in pazienti affette da
abortività spontanea ricorrente.
La gravidanza nella specie umana è un processo inefficiente perché il 70% dei
concepimenti fallisce e gli specifici meccanismi molecolari e/o cellulari che
sottintendono il mantenimento di una normale gravidanza e l’induzione
dell’aborto rimangono ancora poco studiati.
Visto che il tessuto fetale è parzialmente allogenico rispetto alla madre, studi
recenti hanno avanzato l’ipotesi di una patogenesi immunologica nell’aborto
spontaneo [218]. Questi studi suggeriscono in particolare che in donne
gravide le citochine prodotte dalle cellule Th-2 predominano su quelle
prodotte dalle cellule Th-1 e ciò determina l’evoluzione positiva della
gravidanza, mentre una predominante produzione di citochine Th-1 rispetto
alle Th-2 determina più frequentemente un’evoluzione in aborto [71,218].
88
Molti studi hanno valutano i livelli di citochine materne nell’aborto spontaneo
ricorrente [219-224], ma sono molto pochi quelli che hanno valutato
l’espressione citochimica a livello endometriale nella cosiddetta “finestra di
impianto” [127-131].
I nostri dati fanno ipotizzare e sono, in considerazione del numero e del tipo
di metodica applicata, una prova dell’attivazione massiccia e a tutti i livelli
del sistema immunitario che trova poi nell’evento aborto il suo epifenomeno
clinico.
Circa l’aumento del gene codificante per la PPBP, la proteina codificata da
questo gene è un fattore di crescita delle piastrine che appartiene alla famiglia
delle CXC chemochine. E’un importante attivatore dei neutrofili e del
plasminogeno oltre che modulatore di importanti processi intracellulari come
la sintesi di DNA, la mitosi, l’accumulo di AMPc. Ma soprattutto segnaliamo
il suo ruolo cruciale nell’indurre secrezione di prostaglandine che come noto
inducono contrazioni uterine e che fase iniziale di gravidanza renderebbero
l’utero poco recettivo rispetto all’impianto [225].
A questo punto è interessante notare come all’aumento di questo fattore si sia
riscontrato parallelamente una diminuzione dell’IL 11 che ha un notevole
effetto difensivo sui tessuti [226] e una funzione di accelerare la produzione
delle piastrine. Probabilmente l’up-regolazione della PPBP assume anche un
ruolo compensativo nell’ambito dell’omeostasi di tale linea cellulare.
Tra i geni aumentati vi sono anche le BMP, proteine morfogenetiche
dell’osso, che giocano un ruolo importante nello sviluppo di diversi tessuti ed
organi [227]. Le BMP sono una famiglia di fattori multifunzionali che
appartengono a loro volta alla grande famiglia del TGF-" [228].
Gli studi effettuati hanno dimostrato che le BMP giocano un ruolo importante
sia nello sviluppo embrionale che nell’organismo adulto, sono infatti
89
implicate nella formazione e nel mantenimento del tessuto cardiaco,
neuronale, osseo e cartilagineo [228].
Le BMP inoltre stimolano la migrazione, la proliferazione e il
differenziamento delle cellule staminali mesenchimali durante l’embriogenesi
e durante la riparazione e la rigenerazione tissutale nell’organismo adulto
[229]. Ad oggi sono stati identificati e caratterizzati circa 20 membri di questa
famiglia, tra i più studiati ricordiamo le BMP dalla 2 alla 8. Mediante
l’utilizzo di animali transgenici si è potuto capire l’importanza dei geni delle
BMP nello sviluppo di un organismo sano, in particolare la mancanza dei geni
per le BMP-2 e 4 porta all’aborto spontaneo per il mancato sviluppo dello
scheletro e degli annessi placentari [230]. La mancanza del gene per BMP-7
porta alla morte pretermine per gravi difetti dello scheletro, dei reni e degli
occhi [231].
Apparentemente in contrasto con quanto descritto da questi autori, crediamo
che la iperespressione genica da noi riscontrata, nel gruppo di pazienti con
poliabortività, possa essere espressione di un meccanismo di compenso
attivato dall’organismo per supportare lo sviluppo degli annessi e quindi
l’impianto.
Per quanto riguarda i geni down regolati, abbiamo trovato una down
regolazione di molte interleuchine, in particolare della IL 4, IL 5, IL 7, IL 9,
IL 11, IL 12, IL 12B, IL 13, IL 16, IL 17, IL 18, IL 22, IL 23, IL 24, IL 27.
Ogni interleuchina svolge un ruolo più o meno specifico ma tutte sono
strettamente correlate, in particolare la IL 4 è definito in letteratura fattore-1
stimolante le cellule B, per la sua attività di fattore di crescita per tali cellule,
e nei Linfociti B induce "switch isotipico" verso IgE e inibisce il
differenziamento verso i linfociti Th1. Dunque una sua down regolazione
implicherebbe una maggiore espressione del cluster Th1, che verrebbe
90
confermata da una diminuzione dell’IL 5 che è promotrice della
differenziazione Th2. Questo risultato, circa le IL 4, IL 5 e IL 9, è in accordo
con i dati della recente letteratura che evidenziavano l’esistenza di un pattern
Th1 nell’aborto interno. Molti meccanismi possono influenzare positivamente
o negativamente l’azione di queste citochine nell’evoluzione del primo
trimestre di gravidanza. I meccanismi immunologici ed endocrini coinvolti
nel primo trimestre di gravidanza potrebbero ricondursi ad una relazione tra il
trofoblasto umano e i linfociti della decidua: le cellule Th-2 della decidua
producono citochine Th-2 che inducono il rilascio di hCG dal trofoblasto.
L’hCG stimola la produzione di progesterone da parte del corpo luteo, e poi,
potenzialmente attraverso il fattore inibente l’induzione del progesterone
(PIBF), un aumento della secrezione delle citochine Th-2 e una riduzione
delle citochine Th-1 [218]. Questa teoria può essere appropriata anche per la
minaccia d’aborto con esito positivo. I nostri dati supportano l’ipotesi che
l’aborto possa essere associato ad un’aumentata reattività Th-1 mentre, la
gravidanza con esito positivo, avrebbe una reattività Th-1 non aumentata
[232].
Le altre interleuchine risultate down-regolate sono segno di un sistema
alterato, come la IL 13, che ha un ruolo inibitorio delle citochine
infiammatorie, agendo sui linfociti nel sangue periferico o la IL 16, proteina
chemoattraente per i linfociti, e la IL 17 al centro di molti studi, per il suo
ruolo di attivatore di flogosi. Oppure la IL 18, che ha compiti simili alla prima
forma, dove si divide nelle funzioni immunitarie e infiammatorie. Negli
ultimi anni si sta studiando la loro funzione per un possibile utilizzo contro le
malattie infiammatorie [233]. In caso di infezione sistemica, l'interleuchina 27
agisce in maniera simile al TNF-alfa, stimolando la produzione nel fegato
delle cosiddette proteine di fase acuta. ha un importante compito di
91
regolazione dell'attività dei linfociti B e T, ed ha importanti proprietà
immunosoppressive. La minore espressione di tutte queste molecole
compromette globalmente queste funzioni creando i presupposti per un
ambiente sfavorevole all’impianto embrionale.
Il ruolo delle interleuchine nell’eziopatogenesi dell’aborto è stato studiato
anche da Wilson et al. [221] che tuttavia, hanno osservato livelli di IL-12
significativamente più elevati negli aborti ricorrenti rispetto alle donne con
gravidanza normale.
E’ interessante notare come nel gruppo di studio le pazienti mostrino una
down-regolazione di altre interleuchine, in particolare la IL 22 e la ILB12.
Questi dati non sono in contrasto con quanto detto prima, semplicemente
perché la famiglia delle interleuchine è talmente vasta che numerose e
eterogenee sono le funzioni. La down-regolazione dell’interleuchina IL 22 è
da mettere in relazione alla sua attività di promotrice dei fenomeni
angiogenetici. E’ prodotta da TH17 CD4 e della cellule T, il suo ruolo è
quello di cooperare con altre interleuchine la IL 17, e la IL 23 [234]. Queste
interleuchine mediando l’angiogenesi potrebbero giocare un ruolo chiave al
momento dell’impianto dell’embrione e nella formazione dei villi e quindi
della placenta e la loro espressione deficitaria nel gruppo di studio potrebbe
giustificare l’esito negativo della gravidanza.
Per quanto riguarda il risultato dell’interleuchina 12, nota agli addetti ai lavori
come IL-12, è una citochina, cioè una specie di “ormone” prodotto dal corpo
umano che regola la comunicazione tra cellule, ed è dotata di numerosi e
importanti effetti sul sistema immunitario, in particolare attiva cellule natural
killer (NK).
IL 12 induce cellule NK e linfociti T a produrre IFN! che andrà ad attivare
nei macrofagi meccanismi battericidi. IL 12 assieme a IFN! induce il
92
differenziamento in Th1 dei CD4 le quali a loro vola producono IFN!. IL-12
potenzia inoltre l'attività citotossica dei CTL.
Risulta evidente come questa citochina rappresenti un importante punto di
collegamento tra immunità innata ed adattativa stimolandole entrambe.
Potremmo ipotizzare che un suo deficit alteri questo delicato equilibrio
determinando un alterazione incompatibile con il proseguimento della
gravidanza.
In base alle conoscenze attuali sappiamo, infatti, che la sopravvivenza del feto
semi-allogenico è dovuta alla immunosoppressione materna.
Il trofoblasto riesce ad eludere l’attacco da parte del sistema immunitario
materno con vari meccanismi locali, tra cui la particolare configurazione
dell’espressione delle molecole HLA di superficie che, come risaputo, sono
coinvolte nella presentazione antigenica al sistema immunitario materno.
In particolare il trofoblasto non esprime molecole MCH di classe Ia (HLA-A
e HLA-B) con l’eccezione di molecole HLA-C, ma soprattutto esprime
molecole non classiche quali le HLA-G e le HLA-E. Il ruolo delle HLA-G è,
ad oggi, parzialmente conosciuto ma, si presuppone, secondo l’ipotesi più
accreditata, che l’HLA-G, riveste un ruolo preminente nella resistenza del
trofoblasto non villoso alla lisi mediata dalle cellule NK (natural killer),
presenti in gran numero a livello uterino, inibendone l’attività citolitica e la
loro migrazione transplacentare. Anche le molecole HLA-E inibirebbero le
cellule NK a livello dell’interfaccia materno fetale.
Secondo tale teoria, una risposta immunitaria materna eccessiva contro gli
antigeni paterni, porterebbe alla genesi abnorme di cellule immunitarie e di
citochine responsabili di abortività ripetuta. Più precisamente, secondo studi
recenti, esistono differenze significative dei livelli di cellule NK nel sangue
periferico nelle donne con abortività ripetuta/ricorrente rispetto ai gruppi di
93
controllo. Cellule NK sono state rinvenute anche nell’endometrio e nella
decidua di donne con abortività ripetuta, anche se non si conosce il loro ruolo
nel meccanismo di placentazione.
Secondo queste ipotesi, le cellule NK della mucosa uterina contribuiscono
alla risposta citochinica a livello dell’interfaccia materno fetale.
Tale risposta è sia di tipo T-helper-1 (Th1) con sintesi di IL 2, IFN
(interferone), TNF-alfa, sia di tipo T-helper-2(Th2) con sintesi di IL 4, IL 6 e
IL 10.
Oltre ai numerosi tipi di interleuchine che appaiono diminuite possiamo
sottolineare come il nostro studio abbia evidenziato una down-regolazione
anche di molti elementi della famiglia delle chemochine.
L’analisi di espressione genica del tessuto ha mostrato un grande
coinvolgimento delle molecole chemochine, sia in senso di up che di downregolazione. Il profilo genetico così tracciato ha mostrato un iper-espressione
per la chemochina CCL21 e una down-espressione per CCL24, CCL19,
CCL20, CCL22, CCL11, CCL1, CCL3, CCl5, CCL7, CCL8 e della CX3CL1.
Le chemochine sono una famiglia di proteine che modula la migrazione
direzionale nel luogo del bisogno dei linfociti. Il movimento è una
caratteristica cruciale del sistema immunitario: senza la capacità di migrare
rapidamente verso la zona interessata dal processo patologico, verrebbe meno
la possibilità di una risposta adeguata ad insulti patogeni.
La modificata espressione dei geni per le chemochine CCL21 e CCL19 attesta
un’attivazione del sistema immunitario a tutti i livelli. Le due chemochine
attraverso il recettore CCR7, espresso delle cellule dendritiche mature,
guidano la cellula fin dentro l’area T dei linfonodi dove presenterà l’antigene
al linfocita.
94
Inoltre le chemochine agiscono anche come molecole solubili generando il
“gradiente chemochinico” la cui concentrazione nei fluidi segna un po’ la
strada ai linfociti.
Nel gruppo delle pazienti affette da poliabortività si riscontra una diminuzione
delle chemochine CCL1, CCL24, CCL19, se pensiamo che tra le attività di
queste molecole vi è, in aggiunta a quanto sopra, anche il controllo della
differenziazione Th1/Th2 potremmo ipotizzare che la down-regolazione di
queste molecole comporti uno sbilanciamento in senso Th1.
Alla famiglia del TNF, invece, appartiene anche il gene ipo-espresso LTA,
numerosi studi indicano una sua associazione con lo sviluppo di malattie
autoimmunitarie .Un suo deficit comprometterebbe la migrazione di cellule
immunitarie quali macrofagi e linfociti verso il sito di infezione.
Vediamo inoltre la diminuzione di espressione del gene CD40LG, quindi
della proteina da esso codificata, sulla superficie dei linfociti T con
conseguente mancata regolazione delle funzioni dei linfociti B. Un suo deficit
è stato associato allo sviluppo di sindromi mieloproliferative monolocali per
incapacità delle immunoglobuline di effettuare un corretto switch maturativo.
Tra i geni ipo-espressi nel tessuto endometriale di donne affette da
poliabortività vi è anche un gene detto NODAL, la proteina codificata da tale
gene è un membro della TGF-beta superfamiglia. Studi sui topi suggeriscono
un ruolo centrale nella formazione del mesoderma e nella seguente
formazione delle strutture assiali nello sviluppo embrionale ai primissimi
stadi [235,236]; inoltre una sua alterazione è stata evidenziata in donne che
sviluppavano complicanze gestazionali tra cui la preeclampsia [237]. Per
ultimo, ma non per importanza, segnaliamo la down regolazione del gene
CSF-1. I suoi livelli sono normalmente elevati in gravidanze fisiologiche a
livello dell’epitelio endoteliale e alti sono normalmente i livelli del suo
95
recettore CSF1R nel trofoblasto. L’attivazione di tale recettore ad opera della
proteina codificata da CSF-1 è essenziale nello sviluppo della normale
placentazione e ancora prima del processo di impianto.
Il nostro studio ha valutato in maniera estensiva la situazione locale
endometriale delle citochine infiammatorie in una fase del ciclo mestruale
antecedente all’eventuale impianto, aggiungendo un tassello alla complessità
della regolazione dell’impianto e dei rapporti materno-fetali [238,239].
Tra i vari studi esistono considerevoli differenze che potrebbero essere dovute
alla diversa popolazione di studio, alla diversa tempistica di campionamento,
e al fatto se siano state misurate citocine in tutto il tessuto in esame o solo in
una determinata popolazione cellulare; la presenza inoltre di complicati
networks e le loro attività biologiche spesso sovrapponibili rendono
l’interpretazione di tali dati molto complessa.
96
2.5 Conclusioni
L'impianto è un processo biologico complesso che si fonda sulla
comunicazione a due vie tra l'embrione e l'endometrio. Questo dialogo
materno-embrionale
richiede
cambiamenti
progesterone-dipendenti
nell'endometrio per renderlo ricettivo ai segnali provenienti dall’embrione. I
fattori che conferiscono la recettività endometriale sono, nel processo
dell’evoluzione, altamente conservati. I principali regolatori del processo di
impianto sono i fattori di crescita, le citochine e i loro recettori e le molecole
di adesione cellulare come il recettore per il progesterone.
I nostri dati supportano l’ipotesi di un’alterazione a molti livelli del sistema
immunologico alla base dell’evento aborto. Il vasto numero di geni che
appaiono alterati suggerisce parallelamente la grande complessità delle
interazioni molecolari che si realizzano dopo il concepimento e che rendono
l’impianto fenomeno estremamente complesso e a noi per lo più sconosciuto.
Vi è ampia evidenza clinica che la refrattarietà dell'endometrio durante la
finestra di impianto è causa di sub-fertilità e fallimento del trattamento di
riproduzione assistita. Il nostro lavoro è volto a tracciare un profilo
proteomico
(valutazione
dell’insieme
delle
proteine
coinvolte
nella
funzionalità dei meccanismi biologici di specifiche attività cellulari) per
determinare le caratteristiche molecolari di un endometrio ricettivo al fine di
sfruttare queste conoscenze per fini diagnostici e terapeutici.
Mentre capire il profilo molecolare che conferisce la recettività endometriale
è importante nel contesto della subfertilità, altrettanto importante può essere
capire i segnali che terminano la finestra di impianto per prevenire la perdita
precoce della gravidanza. Le cellule dell'endometrio sono biosensori di
qualità degli embrioni. Ci siamo chiesti quale sia la natura dei segnali
97
provenienti da embrioni anomali e quale sia il meccanismo che permette alle
cellule
endometriali
di
percepire
e
rispondere
provocando
la
decidualizzazione. Queste domande rimangono ancora senza risposta. Ci sono
stati numerosi tentativi di stabilire una base immunologica per la mancata
gravidanza persistente anche se sinora non è stato possibile stabilire con
certezza un nesso causale. Alcuni dati sembrano suggerire che i segnali della
regolare differenziazione delle cellule dell’endometrio in cellule specializzate
deciduali sono alterati nelle donne con aborti ripetuti. L’alterazione di uno o
più segnali endocrini coinvolti in questa regolazione potrebbe essere causa
dell’abortività ripetuta.
I nostri risultati sembrano suggerire che la regolazione immunologica ed
infiammatoria
dell'ambiente
endometriale
gioca
un
ruolo
chiave
nell’abortività spontanea ricorrente. Il pattern di espressione genica
endometriale delle citochine conferma una modifica tra il profilo Th1 e Th2
all’interfaccia materno-fetale negli aborti ricorrenti, in coerenza con la teoria
immunologica. L'aspetto immunologico è stato già indagato, ma molto spesso
gli studi presenti in Letteratura si riferiscono a livelli sierici di singole
citochine scelte in base a studi precedenti o ad ipotetici meccanismi
patogenetici. Il nostro studio ha valutato a livello locale, piuttosto che a livello
sistemico, gli aspetti immunologici e infiammatori dell’abortività ricorrente
analizzando nel complesso il coinvolgimento di tutte le citochine
infiammatorie.
L’attuale comprensione dei meccanismi patogenetici degli aborti spontanei
ricorrenti inspiegati è ancora limitata; negli ultimi anni l’espansione della
ricerca nella biologia molecolare ci sta permettendo di addentrarci più
profondamente nella comprensione di alcuni tipi di aborti spontanei. In questo
98
orizzonte le citochine appaiono molto promettenti in quanto hanno un ruolo
chiave nel sistema endocrino e immunitario.
L’abortività spontanea ricorrente è spesso un problema inspiegabile e sono
stati fatti molti tentativi da parte dei ricercatori per dare un potenziale aiuto
alla pratica clinica, ma la nostra comprensione dei meccanismi che portano
all’interruzione della gravidanza è ancora incompleta. Nonostante i recenti
progressi nel campo della medicina riproduttiva, la diagnosi, la gestione, e
ancor di più la terapia, per le coppie con abortività ricorrente sono ancora
settori di ricerca.
99
Bibliografia
1. WHO Recommended Definitions. Terminology and format for statistical
tables related to the prenatal period. Acta Obstet Gynecol Scand
1997;56:247-53
2. Garcia-Enguidanos A, Calle ME, Valero JA, Luna S, Domínguez-Rojas V.
Risk factors in miscarriage: a review. Eur J Obstet Gynecol Reprod Biol
2002;102:111-9
3. American College of Obstetricians and Gynecologists (ACOG). Management
of recurrent early pregnancy loss. Practice Bulletin no. 24. Washington, DC:
American College of Obstetricians and Gynecologists (ACOG); 2001
4. van den Boogaard E, Kaandorp SP, Franssen MT, Mol BW, Leschot NJ, et
al.
Consecutive or non-consecutive recurrent miscarriage: is there any
difference in carrier status? Hum Reprod 2010;25:1411-4
5. Stirrat GM. Recurrent miscarriage. Lancet 1990;336:673-5
6. Coulam CB. Epidemiology of recurrent spontaneous abortion. Am J Reprod
Immunol 1991;26:23-7
7. Vlaanderen W, Treffers PE Prognosis of subsequent pregnancies after
recurrent spontaneous abortion in first trimester. Br Med J (Clin Res Ed)
1987;295:92-3
8. Mevorach-Zussman N, Bolotin A, Shalev H, Bilenko N, Mazor M, Bashiri
A. Anxiety and deterioration of quality of life factors associated with
recurrent miscarriage in an observational study. J Perinat Med 2012;40:495501
9. Regan L. Recurrent miscarriage. BMJ 1991;302:543-4
10. Alberman E. The epidemiology of repeated abortion. In: Beard RW, Sharp F,
editors. Early Pregnancy Loss: Mechanisms and Treatment. London: RCOG
Press;1988 p. 9-17
100
11. Lee R, Silver R. Recurrent pregnancy loss: Summary and clinical
recommendations. Semin Reprod Med 2000;18;433-40
12. Ford HB, Schust DJ. Recurrent Pregnancy Loss: Etiology, Diagnosis, and
Therapy. Rev Obstet Gynecol 2009;2:76-83
13. Stray-Pedersen B, Stray-Pedersen S. Etiologic factors and subsequent
reproductive performance in 195 couples with a prior history of habitual
abortion. Am J Obstet Gynecol 1984;148:140-6
14. Carp HJ, Toder V, Mashiach S, Nebel L, Serr DM. Recurrent miscarriage: a
review of current concepts, immune mechanisms, and results of treatment.
Obstet Gynecol Surv 1990;45:657-69. Review.
15. Royal College of Obstetricians and Gynaecologists. The Investigation and
Treatment of Couples with Recurrent Miscarriage. Guideline No.17. London
Revised 2003 May
16. Diedrich U, Hansmann I, Janke D, Opitz O, Probeck HD. Chromosome
anomalies in 136 couples with a history of recurrent abortions. Hum Genet
1983;65:48-52
17. Carp H, Feldman B, Oelsner G, Schiff E. Parental karyotype and subsequent
live births in recurrent miscarriage. Fertil Steril 2004;81:1296-301
18. Abalovich M, Amino N, Barbour LA, Cobin RH, De Groot LJ, Glinoer D,
Mandel SJ, Stagnaro-Green A. Management of thyroid dysfunction during
pregnancy and postpartum: an Endocrine Society Clinical Practice Guideline.
J Clin Endocrinol Metab 2007;92:S1-47
19. Thatcher SS1, Jackson EM. Pregnancy outcome in infertile patients with
polycystic ovary syndrome who were treated with metformin. Fertil Steril
2006;85:1002-9
20. Jakubowicz DJ, Iuorno MJ, Jakubowicz S, Roberts KA, Nestler JE. Effects
of metformin on early pregnancy loss in the polycystic ovary syndrome. J
Clin Endocrinol Metab 2002;87:524-9
101
21. Legro RS, Barnhart HX, Schlaff WD, Carr BR, Diamond MP, Carson SA,
Steinkampf MP, Coutifaris C, McGovern PG, Cataldo NA, Gosman GG,
Nestler JE, Giudice LC, Leppert PC, Myers ER; Cooperative Multicenter
Reproductive Medicine Network. Clomiphene, metformin, or both for
infertility in the polycystic ovary syndrome. N Engl J Med 2007;356:551-66
22. Preston FE, Rosendaal FR, Walker ID, Briët E, Berntorp E, Conard J,
Fontcuberta J, Makris M, Mariani G, Noteboom W, Pabinger I, Legnani C,
Scharrer I, Schulman S, van der Meer FJ. Increased fetal loss in women with
heritable thrombophilia. Lancet. 1996;348:913-6
23. Rey E, Kahn SR, David M, Shrier I. Thrombophilic disorders and fetal loss:
a meta-analysis. Lancet 2003;361:901-8
24. Krabbendam I, Franx A, Bots ML, Fijnheer R, Bruinse HW. Thrombophilias
and recurrent pregnancy loss: a critical appraisal of the literature. Eur J
Obstet Gynecol Reprod Biol 2005;118:143-53
25. Wouters MG, Boers GH, Blom HJ, Trijbels FJ, Thomas CM, Borm GF,
Steegers-Theunissen RP, Eskes TK. Hyperhomocysteinemia: a risk factor in
women with unexplained recurrent early pregnancy loss. Fertil Steril
1993;60:820-5
26. Grandone E, Margaglione M, Colaizzo D, d'Addedda M, Cappucci G,
Vecchione G, Sciannamé N, Pavone G, Di Minno G. Factor V Leiden is
associated with repeated and recurrent unexplained fetal losses. Thromb
Haemost. 1997;77:822-4
27. Johnson PM, Christmas SE, Vince GS. Immunological aspects of
implantation and implantation failure. Hum Reprod1999;14:26-36
28. King A. Uterine leukocytes and decidualization. Hum Reprod Update
2000;6:28-36
29. Moffett A, Regan L, Braude P. Natural killer cells, miscarriage, and
infertility. BMJ 2004;329:1283-5
102
30. Wold AS, Arici A. Natural killer cells and reproductive failure. Curr Opin
Obstet Gynecol 2005;17:237-41
31. Rai R, Sacks G,Trew G. Natural killer cells and reproductive failure – theory,
practice and prejudice. Hum Reprod 2005;20:1123-6
32. Le Bouteiller P, Piccinni MP. Human NK cells in pregnant uterus: why
there? Am J Reprod Immunol 2008;59:401-6
33. Tuckerman E, Laird SM, Prakash A, Li TC. Prognostic value of the
measurement of uterine natural killer cells in the endometrium of women
with recurrent miscarriage. Hum Reprod 2007;22:2208-13
34. Bombell S, McGuire W. Cytokine polymorphisms in women with recurrent
pregnancy loss: meta-analysis. Aust N Z J Obstet Gynaecol 2008;48:147-54
35. Royal College of Obstetricians and Gynaecologists. The Investigation and
Treatment of Couples with Recurrent First- trimester and Second-trimester
Miscarriage. Green–top Guideline No. 17. April 2011
36. Cameron MJ, Kelvin DJ. Cytokines, Chemokines and their Receptors. 20002005 Landes Bioscience. Eurekah Bioscience Collection
37. Cohen S, Bigazzi PE, Yoshida T. Commentary. Similarities of T cell
function in cell-mediated immunity and antibody production. Cell Immunol
1974;12:150-9
38. Dumonde DC, Wolstencroft RA, Panayi GS. "Lymphokines": Non-antibody
mediators of cellular immunity generated by lymphocyte activation. Nature
1969;224:38-42
39. Paul WE, Seder RA. Lymphocyte responses and cytokines. Cell
1994;76:241-51
40. Paul WE, Seder RA. Acquisition of lymphokine-producing phenotype by
CD4+ T cells. Annu Rev Immunol 1994;12:635-73
41. Romagnani S. The Th1/Th2 paradigm. Immunol Today 1997;18:263-6
42. Luther SA, Cyster JG. Chemokines as regulators of T cell differentiation. Nat
Immunol 2001;2:102-7
103
43. Mosmann TR, Cherwinski H, Bond MW. Two types of murine helper T cell
clone. I. Definition according to profiles of lymphokine activities and
secreted proteins. J Immunol 1986;136:2348-57
44. Medawar PB. Some immunological and endocrinological problems raised by
the evolution of viviparity in vertebrates, Symp Soc Exp Biol Med
1953;7:320-38
45. Lim KJH, Odukoya OA, Li TC, Cooke ID. Cytokines and immuno-endocrine
factors in recurrent miscarriage. Hum Reprod Update 1996;2:469-81
46. Entrican G. Immune regulation during pregnancy and host–pathogen
interactions in infectious abortion. J Comp Pathol 2002;126:79-94
47. Ljunggren HG, Karre K. In search of the ‘missing self’: MHC molecules and
NK cell recognition. Immunol Today 1990;11:237-44
48. King A, Burrows TD, Hiby SE, Bowen JM, Joseph S, Verma S, Lim PB,
Gardner L, Le Bouteiller P, Ziegler A, Uchanska-Ziegler B, Loke YW.
Surface expression of HLA-C antigen by human extravillous trophoblast.
Placenta 2000;21:376-87
49. Hunt JS, Petroff MG, McIntire RH, Ober C. HLA-G and immune tolerance
in pregnancy. FASEB J 2005;19:681-93
50. Ellis SA, Palmer MS, McMichael AJ. Human trophoblast and the
choriocarcinoma cell line BeWo express a truncated HLA Class I molecule. J
Immunol 1990;144:731-5
51. Kovats S, Main EK, Librach C, Stubblebine M, Fisher SJ, DeMars R. A class
I antigen, HLA-G, expressed in human trophoblasts. Science 1990;248:220-3
52. Acar N, Ustunel I, Demir R. Uterine natural killer (uNK) cells and their
missions during pregnancy: a review. Acta Histochem 2011;113:82-91
53. Ghiringhelli F, Menard C, Martin F, Zitvogel L. The role of regulatory T
cells in the control of natural killer cells: relevance during tumor progression.
Immunol Rev 2006;214:229-38
104
54. Trowsdale J, Betz AG. Mother's little helpers: mechanisms of maternal–fetal
tolerance. Nat Immunol 2006;7:241-6
55. Heikkinen J, Mottonen M, Alanen A, Lassila O. Phenotypic characterization
of regulatory T cells in the human deciduas. Clin Exp Immunol
2004;136:373-8
56. Tilburgs T, Roelen DL, van der Mast BJ, de Groot-Swings GM, Kleijburg C,
Scherjon SA, Claas FH. Evidence for a selective migration of fetus-specific
CD4+CD25 bright regulatory T cells from the peripheral blood to the
decidua in human pregnancy. J Immunol 2008;180:5737-45
57. Arruvito L, Sanz M, Banham AH, Fainboim L. Expansion of CD4+CD25+
and FOXP3+ regulatory T cells during the follicular phase of the menstrual
cycle: implications for human reproduction. J Immunol 2007;178:2572-8
58. Sasaki Y, Sakai M, Miyazaki S, Higuma S, Shiozaki A, Saito S. Decidual
and peripheral blood CD4+CD25+ regulatory T cells in early pregnancy
subjects and spontaneous abortion cases. Mol Hum Reprod 2004;10:347-53
59. Arruvito L, Billordo A, Capucchio M, Prada ME, Fainboim L. IL-6 transsignaling and the frequency of CD4+FOXP3+ cells in women with
reproductive failure. J Reprod Immunol 2009;82:158-65
60. Hill JA. T-helper 1-type immunity to trophoblast: evidence for a new
immunological mechanism for recurrent abortion in women. Hum Reprod
1995;2:114-20. Review.
61. Raghupathy R, Makhseed M, Azizieh F, Omu A, Gupta M, Farhat R.
Cytokine production by maternal lymphocytes during normal human
pregnancy and in unexplained recurrent spontaneous abortion. Hum Reprod
2000;15:713-8
62. Makhseed M, Raghupathy R, Azizieh F, Omu A, Al-Shamali E, Ashkanani
L. Th1 and Th2 cytokine profiles in recurrent aborters with successful
pregnancy and with subsequent abortions. Hum Reprod 2001;16:2219-26
105
63. Lea RG, Underwood J, Flanders KC, Hirte H, Banwatt D, Finotto S, Ohno I,
Daya S, Harley C, Michel M, et al. A subset of patients with recurrent
spontaneous abortion is deficient in transforming growth factor beta-2producing "suppressor cells" in uterine tissue near the placental attachment
site. Am J Reprod Immunol 1995;34:52-64
64. Piccinni MP, Beloni L, Livi C, Maggi E, Scarselli G, Romagnani S.
Defective production of both leukemia inhibitory factor and type 2 T-helper
cytokines by decidual T cells in unexplained recurrent abortions. Nat Med
1998;4:1020-4
65. Vassiliadou N, Searle RF, Bulmer JN. Immunoregulatory activity of decidua
in spontaneous early pregnancy loss. Hum Reprod 1999;14:2252-6
66. Michimata T, Sakai M, Miyazaki S, Ogasawara MS, Suzumori K, Aoki K,
Nagata K, Saito S. Decrease of T-helper 2 and T-cytotoxic 2 cells at
implantation sites occurs in unexplained recurrent spontaneous abortion with
normal chromosomal content. Hum Reprod 2003;18:1523-8
67. Löhning M, Hegazy AN, Pinschewer DD, Busse D, Lang KS, Höfer T,
Radbruch A, Zinkernagel RM, Hengartner H. Long-lived virus-reactive
memory T cells generated from purified cytokine-secreting T helper type 1
and type 2 effectors. J Exp Med 2008;205:53-61
68. Mosmann TR, Sad S. Th1 and Th2 cells: different patterns of lymphokine
secretion lead to different functional properties. Annu Rev Immunol Today
1989;7:138-46
69. Mosmann TR, Coffman RL. Heterogeneity of cytokine secretion patterns and
functions of helper T cells. Adv Immunol 1989;46:111-47. Review.
70. Romagnani P, Annunziato F, Piccinni MP, Maggi E, Romagnani S. Th1/Th2
cells, their associated molecules and role in pathophysiology. Eur Cytokine
Netw 2000;11:510-1
106
71. Wegmann TG, Lin H, Guilbert L, Mosmann TR. Bidirectional cytokine
interactions in the maternal–fetal relationship: is successful pregnancy a Th2
phenomenon? Immunol Today 1993;14:353-7
72. Chaouat G, Zourbas S, Ostojic S, Lappree-Delage G, Dubanchet S, Ledee N,
Martal J. A brief review of recent data on some cytokine expressions at the
materno-foetal interface which might challenge the classical Th1/Th2
dichotomy. J Reprod Immunol 2002;53:241-56. Review
73. Raghupathy R. Th1-type immunity is incompatible with successful
pregnancy. Immunol Today 1997;18:478-82. Review.
74. Clark DE, Smith SK, Licence D, Evans AL, Charnock-Jones DS.
Comparison of expression patterns for placenta growth factor, vascular
endothelial growth factor (VEGF), VEGF-B and VEGF-C in the human
placenta throughout gestation. J Endocrinol 1998;159:459-67
75. Del Prete G, De Carli M, Lammel RM, D'Elios MM, Daniel KC, Giusti B,
Abbate R, Romagnani S. Th1 and Th2 T-helper cells exert opposite
regulatory effects on procoagulant activity and tissue factor production by
human monocytes. Blood 1995;86:250-7
76. Raghupathy R, Makhseed M, Azizieh F, Hassan N, Al-Azemi M, Al-Shamali
E. Maternal Th1- and Th2-type reactivity to placental antigens in normal
human pregnancy and unexplained recurrent spontaneous abortions. Cell
Immunol 1999;196:122-30
77. Rezaei A, Dabbagh A. T-helper (1) cytokines increase during early
pregnancy in women with a history of recurrent spontaneous abortion. Med
Sci Monit 2002;8:607-10
78. Hossein H, Mahroo M, Abbas A, Firouzeh A, Nadia H. Cytokine production
by peripheral blood mononuclear cells in recurrent miscarriage. Cytokine
2004;28:83-6
79. Makhseed M, Raghupathy R, Azizieh F, Al-Azemi MM, Hassan NA, Bandar
A. Mitogen-induced cytokine responses of maternal peripheral blood
107
lymphocytes indicate a differential Th-type bias in normal pregnancy and
pregnancy failure. Am J Reprod Immunol 1999;42:273-81
80. Kwak-Kim JY, Chung-Bang HS, Ng SC, Ntrivalas EI, Mangubat CP,
Beaman KD, Beer AE, Gilman-Sachs A. Increased T helper 1 cytokine
responses by circulating T cells are present in women with recurrent
pregnancy losses and in infertile women with multiple implantation failures
after IVF. Hum Reprod 2003;18:767-73
81. Bates MD, Quenby S, Takakuwa K, Johnson PM, Vince GS. Aberrant
cytokine production by peripheral blood mononuclear cells in recurrent
pregnancy loss? Hum Reprod 2002;17:2439-44
82. Daher S, de Arruda Geraldes Denardi K, Blotta MH, Mamoni RL, Reck AP,
Camano L, Mattar R. Cytokines in recurrent pregnancy loss. J Reprod
Immunol 2004;62:151-7
83. Giannubilo SR, Landi B, Pozzi V, Sartini D, Cecati M, Stortoni P, Corradetti
A, Saccucci F, Tranquilli AL, Emanuelli M. The involvement of
inflammatory cytokines in the pathogenesis of recurrent miscarriage.
Cytokine 2012;58:50-6
84. Bubanovic IV. Induction of thymic tolerance as possibility in prevention of
recurrent spontaneous abortion. Med Hypotheses 2003;60:520-4
85. Porter TF, LaCoursiere Y, Scott JR. Immunotherapy for recurrent
miscarriage. Cochrane Database Syst Rev CD2006;000112
86. Christiansen OB. Evidence-based investigations and treatments of recurrent
pregnancy loss. Curr Opin Obstet Gynecol 2006;18:304-12
87. Pandey MK, Thakur S, Agrawal S. Lymphocyte immunotherapy and its
probable mechanism in the maintenance of pregnancy in women with
recurrent spontaneous abortion. Arch Gynecol Obstet 2004;269:161-72
88. Ruiz-Alonso M, Blesa D, Simón C. The genomics of the human
endometrium. Biochim Biophys Acta 2012;1822:1931-42
108
89. Lessey BA. Assessment of endometrial receptivity. Fertil Steril 2011;96:5229
90. Hawkins SM, Matzuk MM. The menstrual cycle: basic biology. Ann N Y
Acad Sci 2008;1135:10-8
91. Simón C, Martín JC, Pellicer A. Paracrine regulators of implantation.
Baillieres Best Pract Res Clin Obstet Gynaecol 2000;14:815-26. Review.
92. Singh M, Chaudhry P, Asselin E. Bridging endometrial receptivity and
implantation: network of hormones, cytokines, and growth factors. J
Endocrinol 2011;210:5-14
93. Rashid NA, Lalitkumar S, Lalitkumar PG, Gemzell-Danielsson K.
Endometrial receptivity and human embryo implantation. Am J Reprod
Immunol 2011;66:23-30
94. Brosens JJ, Gellersen B. Something new about early pregnancy: decidual
biosensoring and natural embryo selection. Ultrasound Obstet Gynecol
2010;36:1-5
95. Lessey BA. Endometrial receptivity and the window of implantation.
Baillieres Best Pract Res Clin Obstet Gynaecol 2000;14:775-88. Review
96. Irizarry RA, Warren D, Spencer F, Kim IF, Biswal S, Frank BC, Gabrielson
E, Garcia JG, Geoghegan J, Germino G, Griffin C, Hilmer SC, Hoffman E,
Jedlicka AE, Kawasaki E, Martínez-Murillo F, Morsberger L, Lee H,
Petersen D, Quackenbush J, Scott A, Wilson M, Yang Y, Ye SQ, Yu W.
Multiple-laboratory comparison of microarray platforms. Nat Methods
2005;2:345-50
97. Bajpai AK, Davuluri S, Chandrashekar DS, Ilakya S, Dinakaran M, Acharya
KK. MGEx-Udb: a mammalian uterus database for expression-based
cataloguing of genes across conditions, including endometriosis and cervical
cancer. PLoS One 2012;7:e36776
109
98. Sherwin R, Catalano R, Sharkey A. Large-scale gene expression studies of
the endometrium: what have we learnt? Reproduction 2006;132:1-10.
Review.
99. Ponnampalam AP, Weston GC, Trajstman AC, Susil B, Rogers PA.
Molecular
classification
of
human
endometrial
cycle
stages
by
transcriptional profiling. Mol Hum Reprod 2004;10:879-93
100.
Kao LC, Tulac S, Lobo S, lmani B, Yang JP, Gemieyer A. Global
gene profiling in human endometrium during the window of implantation.
Endocrinology 2002;143:2119-38
101.
Carson DD, Lagow E, Thathiah A, Al-Shami R, Farach-Carson MC,
Vernon M. Changes in gene expression during the early to mid-luteal
(receptive phase) transition in human endometrium detected by high-density
microarray screening. Mol Hum Reprod 2002;8:871-9
102.
Borthwick JM. Charnoclc-Jones DS, Tom ML, Hull BD, Teimey R,
Phillips SC. Determination of the transcript profile of human cndometrium.
Mol Hum Reprod 2003;9:19-33
103.
Riesewijk A, Martin J, Van Os R, Horcajadas JA, Polman J, Pellicer
A. Gene expression profiling of human endometrial receptivity on days
LH+2 versus LH+7 by microarray technology. Mol Hum Reprod
2003;9:253-64
104.
Haouzi D, Mahmoud K, Fourar M, Bendhaou K, Dechaud H, De Vos
J. Identification of new biomarkers of human endometrial receptivity in the
natural cycle. Hum Reprod 2009;24:l98-205
105.
Punyadeera C, Dassen H, Klomp J, Dunselman G, Kamps R, Dijcks F.
Oestrogen-modulated gene expression in the human endometrium. Cell Mol
Life Sci 2005;62:239-50
106.
Revel A, Achache H, Stevens J, Smith Y, Reich R. MicroRNAs are
associated with human embryo implantation defects. Hum Reprod
2011;26:2830-40
110
107.
Van Vaerenbergh I, Mclntire R, Van Lommel L, Devroey P, Giudice
L, Bourgain C. Gene expression during successful implantation in a natural
cycle. Fertil Steril 2010;93:268.e15-268.el8
108.
Yanaihara A, Otsuka Y, lwasaki S, Aida T, Tachikawa T, lrie T.
Differences in gene expression in the proliferative human endometrium.
Fertil Steril 2005;83(Suppl. l): l206-15
109.
Horcajadas JA, Riesewijk A, Martin J, Cervero A, Mosselman S,
Pellicer A. Global gene expression profiling of human endometrial
receptivity. J Reprod Immunol 2004;63:41-9
110.
Mirkin S, Arslan M, Churikov D, Corica A, Diaz JI, Williams S. ln
search of candidate genes critically expressed in the human endometrium
during the window of implantation. Hum Reprod 2005;20:2104-17
111.
Talbi S, Hamilton AE, Vo KC, Tulac S, Overgaard MT, Dosiou C.
Molecular phenotyping of human endometrium distinguishes menstrual cycle
phases and underlying biological processes in normo-ovulatory women.
Endocrinology 2006;147:1097-121
112.
Critchley HO, Robertson KA, Forster T, Henderson TA, Williams
AR, Ghazal P. Gene expression profiling of mid to late secretory phase
endometrial biopsies from women with menstrual complaint. Am J Obstet
Gynecol 2006;195:406.el-406.l6
113.
Kuokkanen S, Chen B, Ojalvo L, Benard L, Santoro N, Pollard JW.
Genomic profiling of microRNAs and messenger RNAs reveals hormonal
regulation in microRNA expression in human endometrium. Biol Reprod
2010;82:791-801
114.
Tseng LH, Chen I, Chen MY, Yan H, Wang CN, Lee CL. Genome-
based expression profiling as a single standardized microarray platform for
the diagnosis of endometrial disorder: an array of 126-gene model. Fertil
Steril 2010;94:ll4-9
111
115.
Diaz-Gimeno P, Horcajadas JA, Martinez-Conejero JA, Esteban FJ,
Alama P, Pellicer A. A genomic diagnostic tool for human endometrial
receptivity based on the transcriptomic signature. Fertil Steril 2011;95:50-60
116.
Altmae S, Martinez-Conejero JA, Salumets A, Simon C, Horcajadas
JA, Stavreus-Evers A. Endometrial gene expression analysis at the time of
embryo implantation in women with unexplained infertility. Mol Hum
Reprod 2010;16:178-87
117.
Koler M, Achache H, Tsafrir A, Smith Y, Revel A, Reich R.
Disrupted gene pattem in patients with repeated in vitro fertilization (IVF)
failure. Hum Reprod 2009;24:2541-8
118.
Tapia A, Gangi LM, Zegers-Hochschild F, Balmaceda J, Pommer R,
Trejo L. Differences in the endometrial transcript profile during the receptive
period between women who were refractory to implantation and those who
achieved pregnancy. Hum Reprod 2008;23:340-51
119.
Matsuzald S. DNA microarray analysis in endometriosis for
development of more effective targeted therapies. Front Biosci (Elite Ed.)
2011;3:1139-53
120.
Habermann JK, Bundgen NK, Gemoll T, Hautaniemi S, Lundgren C,
Wangsa D. Genomic instability influences the transcriptome and proteome in
endometrial cancer subtypes. Mol Cancer 2011;10:132
121.
Horcajadas JA, Minguez P, Dopazo J, Esteban FJ, Dominguez F,
Giudice LC. Controlled ovarian stimulation induces a functional genomic
delay of the endometrium with potential clinical implications. J Clin
Endocrinol Metab 2008;93:4500-10
122.
Simon C, Oberye J, Bellver J, Vidal C, Bosch E, Horcajadas JA.
Similar endometrial development in oocyte donors treated with either highor standard-dose GnRH antagonist compared to treatment with a GnRH
agonist or in natural cycles. Hum Reprod 2005;20:3318-27
112
123.
Giudice LC. Application of functional genommics to primate
endometrium: insights into biological processes. Reprod Biol Endocrinol
2006;4:S4
124.
Simon C, Gimeno MJ, Mercader A, O'Connor JE, Remohi J, Polan
ML. Embryonic regulation of integrins beta 3, alpha 4, and alpha l in human
endometrial epithelial cells in vitro. J Clin Endocrinol Metab 1997;82:260716.
125.
Guzeloglu-Kayisli O, Kayisli UA, Taylor HS. The role of growth
factors and cytokine during implantation: endocrine and paracrine
interactions. Semin Reprod Med 2009;27:62-79
126.
Krieg SA, Fan X, Hong Y, Sang QX, Giaccia A, Westphal LM, Lathi
RB, Krieg AJ, Nayak NR. Global alteration in gene expression profiles of
deciduas from women with idiopathic recurrent pregnancy loss. Mol Hum
Reprod 2012;18:442-50
127.
Lédée N, Munaut C, Aubert J, Sérazin V, Rahmati M, Chaouat G,
Sandra O, Foidart JM. Specific and extensive endometrial deregulation is
present before conception in IVF/ICSI repeated implantation failures (IF) or
recurrent miscarriages. J Pathol 2011;225:554-64
128.
Othman R, Omar MH, Shan LP, Shafiee MN, Jamal R, Mokhtar NM
Microarray profiling of secretory-phase endometrium from patients with
recurrent miscarriage. Reprod Biol 2012;12:183-99
129.
von Wolff M, Thaler CJ, Strowitzki T, Broome J, Stolz W,
Tabibzadeh S. Regulated expression of cytokines in human endometrium
throughout the menstrual cycle: dysregulation in habitual abortion. Mol Hum
Reprod 2000;6:627-34
130.
K.
Banerjee P, Jana SK, Pasricha P, Ghosh S, Chakravarty B, Chaudhury
Proinflammatory
cytokines
induced
altered
expression
of
cyclooxygenase-2 gene results in unreceptive endometrium in women with
idiopathic recurrent spontaneous miscarriage. Fertil Steril 2013;99:179-87
113
131.
Jasper MJ, Tremellen KP, Robertson SA. Reduced expression of IL-6
and IL-1alpha mRNAs in secretory phase endometrium of women with
recurrent miscarriage. J Reprod Immunol 2007;73:74-84
132.
Li TC, Tuckerman EM, Laird SM. Endometrial factors in recurrent
miscarriage. Hum Reprod Update 2002;8:43-52. Review
133.
Banerjee P, Ghosh S, Dutta M, Subramani E, Khalpada J,
Roychoudhury S, Chakravarty B, Chaudhury K. Identification of key
contributory factors responsible for vascular dysfunction in idiopathic
recurrent spontaneous miscarriage. PLoS One 2013;8:e80940
134.
Suzuki D, Yamada A, Aizawa R, Funato S, Matsumoto T, Suzuki W,
Takami M, Miyamoto Y, Suzawa T, Yamamoto M, Baba K, Kamijo R.
BMP2 differentially regulates the expression of Gremlin1 and Gremlin2, the
negative regulators of BMP function, during osteoblast differentiation. Calcif
Tissue Int 2012;91:88-96
135.
Lories RJ, Derese I, Ceuppens JL, Luyten FP. Bone morphogenetic
proteins 2 and 6, expressed in arthritic synovium, are regulated by
proinflammatory cytokines and differentially modulate fibroblast-like
synoviocyte apoptosis. Arthritis Rheum. 2003;48:2807-18
136.
Erickson GF, Fuqua L, Shimasaki S.J Analysis of spatial and temporal
expression patterns of bone morphogenetic protein family members in the rat
uterus over the estrous cycle. Endocrinol. 2004;182:203-17
137.
Hromas R, Hufford M, Sutton J, Xu D, Li Y, Lu L. PLAB, a novel
placental bone morphogenetic protein. Biochim Biophys Acta 1997;1354:404
138.
Zhao GQ, Hogan BL. Evidence that mouse Bmp8a (Op2) and Bmp8b
are duplicated genes that play a role in spermatogenesis and placental
development. Mech Dev 1996;57:159-68
114
139.
Nakashima K, Yanagisawa M, Arakawa H, Kimura N, Hisatsune T,
Kawabata M, Miyazono K, Taga T. Synergistic signaling in fetal brain by
STAT3-Smad1 complex bridged by p300. Science 1999;284:479-82
140.
White PM, Morrison SJ, Orimoto K, Kubu CJ, Verdi JM, Anderson
DJ. Neural crest stem cells undergo cell-intrinsic developmental changes in
sensitivity to instructive differentiation signals. Neuron 2001;29:57-71
141.
Kléber M, Lee HY, Wurdak H, Buchstaller J, Riccomagno MM, Ittner
LM, Suter U, Epstein DJ, Sommer L. Neural crest stem cell maintenance by
combinatorial Wnt and BMP signaling. J Cell Biol 2005;169:309-20
142.
Cejalvo T, Sacedón R, Hernández-López C, Diez B, Gutierrez-Frías
C, Valencia J, Zapata AG, Varas A, Vicente A. Bone morphogenetic protein2/4 signalling pathway components are expressed in the human thymus and
inhibit early T-cell development. Immunology 2007;121:94-104
143.
Plikus MV, Mayer JA, de la Cruz D, Baker RE, Maini PK, Maxson R,
Chuong CM. Cyclic dermal BMP signalling regulates stem cell activation
during hair regeneration. Nature 2008;451:340-4
144.
Beck HN, Drahushuk K, Jacoby DB, Higgins D, Lein PJ. Bone
morphogenetic protein-5 (BMP-5) promotes dendritic growth in cultured
sympathetic neurons. BMC Neurosci 2001;2:12
145.
Segklia A, Seuntjens E, Elkouris M, Tsalavos S, Stappers E, Mitsiadis
TA, Huylebroeck D, Remboutsika E, Graf D. Bmp7 regulates the survival,
proliferation, and neurogenic properties of neural progenitor cells during
corticogenesis in the mouse. PLoS One 2012;7:e34088
146.
You L, Kruse FE. Differential effect of activin A and BMP-7 on
myofibroblast differentiation and the role of the Smad signaling pathway.
Invest Ophthalmol Vis Sci 2002;43:72-81
147.
Cheng H, Jiang W, Phillips FM, Haydon RC, Peng Y, Zhou L, Luu
HH, An N, Breyer B, Vanichakarn P, Szatkowski JP, Park JY, He TC.
115
Osteogenic activity of the fourteen types of human bone morphogenetic
proteins (BMPs). J Bone Joint Surg Am 2003;85-A:1544-52
148.
Zeisberg EM, Tarnavski O, Zeisberg M, Dorfman AL, McMullen JR,
Gustafsson E, Chandraker A, Yuan X, Pu WT, Roberts AB, Neilson EG,
Sayegh MH, Izumo S, Kalluri R. Endothelial-to-mesenchymal transition
contributes to cardiac fibrosis. Nat Med. 2007;13:952-61
149.
Tseng YH, Kokkotou E, Schulz TJ, Huang TL, Winnay JN, Taniguchi
CM, Tran TT, Suzuki R, Espinoza DO, Yamamoto Y, Ahrens MJ, Dudley
AT, Norris AW, Kulkarni RN, Kahn CR. New role of bone morphogenetic
protein 7 in brown adipogenesis and energy expenditure. Nature
2008;454:1000-4
150.
Li QS, Qi HB. Expression of bone morphogenetic protein-2 in fetal
membranes from pregnant women with premature rupture of membranes and
its clinic significante. Zhonghua Fu Chan Ke Za Zhi. 2009;44:405-8
151.
Kim GJ, Romero R, Kuivaniemi H, Tromp G, Haddad R, Kim YM,
Kim MR, Nien JK, Hong JS, Espinoza J, Santolaya J, Yoon BH, Mazor M,
Kim CJ. Expression of bone morphogenetic protein 2 in normal spontaneous
labor at term, preterm labor, and preterm premature rupture of membranes.
Am J Obstet Gynecol 2005;193:1137-43
152.
Kodama A, Yoshino O, Osuga Y, Harada M, Hasegawa A, Hamasaki
K, Takamura M, Koga K, Hirota Y, Hirata T, Takemura Y, Yano T, Taketani
Y. Progesterone decreases bone morphogenetic protein (BMP) 7 expression
and BMP7 inhibits decidualization and proliferation in endometrial stromal
cells. Hum Reprod 2010;25:751-6
153.
Stoikos CJ, Harrison CA, Salamonsen LA, Dimitriadis E. A distinct
cohort of the TGFbeta superfamily members expressed in human
endometrium regulate decidualization. Hum Reprod 2008;23:1447-56
154.
Förster R, Davalos-Misslitz AC, Rot A. CCR7 and its ligands:
balancing immunity and tolerance. Nat Rev Immunol 2008;8:362-71
116
155.
Badr G, Borhis G, Treton D, Richard Y. IFN{alpha} enhances human
B-cell chemotaxis by modulating ligand-induced chemokine receptor
signaling and internalization. Int Immunol 2005;17:459-67
156.
Reif K, Ekland EH, Ohl L, Nakano H, Lipp M, Förster R, Cyster JG.
Balanced responsiveness to chemoattractants from adjacent zones determines
B-cell position. Nature 2002;416:94-9
157.
Badr G, Borhis G, Treton D, Moog C, Garraud O, Richard Y. HIV
type 1 glycoprotein 120 inhibits human B cell chemotaxis to CXC
chemokine ligand (CXCL) 12, CC chemokine ligand (CCL)20, and CCL21.
J Immunol 2005;175:302-10
158.
Rangel-Moreno J, Moyron-Quiroz JE, Hartson L, Kusser K, Randall
TD. Pulmonary expression of CXC chemokine ligand 13, CC chemokine
ligand 19, and CC chemokine ligand 21 is essential for local immunity to
influenza. Proc Natl Acad Sci U S A 2007;104:10577-82
159.
Cassier PA, Treilleux I, Bachelot T, Ray-Coquard I, Bendriss-
Vermare N, Ménétrier-Caux C, Trédan O, Goddard-Léon S, Pin JJ, Mignotte
H, Bathélémy-Dubois C, Caux C, Lebecque S, Blay JY. Prognostic value of
the expression of C-Chemokine Receptor 6 and 7 and their ligands in nonmetastatic breast cancer. BMC Cancer 2011;11:213
160.
Shields JD, Kourtis IC, Tomei AA, Roberts JM, Swartz MA.
Induction of lymphoidlike stroma and immune escape by tumors that express
the chemokine CCL21. Science 2010;328:749-52
161.
Nancy P, Tagliani E, Tay CS, Asp P, Levy DE, Erlebacher A.
Chemokine gene silencing in decidual stromal cells limits T cell access to the
maternal-fetal interface. Science 2012;336:1317-21
162.
Kim MJ, Romero R, Kim CJ, Tarca AL, Chhauy S, LaJeunesse C, Lee
DC, Draghici S, Gotsch F, Kusanovic JP, Hassan SS, Kim JS. Villitis of
unknown etiology is associated with a distinct pattern of chemokine upregulation in the feto-maternal and placental compartments: implications for
117
conjoint maternal allograft rejection and maternal anti-fetal graft-versus-host
disease. J Immunol 2009;182:3919-27
163.
Castor CW, Miller JW, Walz DA. Structural and biological
characteristics of connective tissue activating peptide (CTAP-III), a major
human platelet-derived growth factor. Proc Natl Acad Sci U S A
1983;80:765-9
164.
Castor CW, Furlong AM, Carter-Su C. Connective tissue activation:
stimulation of glucose transport by connective tissue activating peptide III.
Biochemistry 1985;24:1762-7
165.
Dominguez F, Pellicer A, Simon C. The chemokine connection:
hormonal and embryonic regulation at the human maternal-embryonic
interface--a review. Placenta 2003;24:S48-55
166.
Fraccaroli L, Alfieri J, Leiros CP, Ramhorst R. Immunomodulatory
effects of chemokines during the early implantation window. Front Biosci
(Elite Ed). 2009;1:288-98
167.
Hannan NJ, Salamonsen LA. Role of chemokines in the endometrium
and in embryo implantation. Curr Opin Obstet Gynecol 2007;19:266-72
168.
Ramhorst R, Patel R, Corigliano A, Etchepareborda JJ, Fainboim L,
Schust D. Induction of maternal tolerance to fetal alloantigens by RANTES
production. Am J Reprod Immunol 2006;56:302-11
169.
Zhang Y1, Gu Y, Li JX, Li Y Correlations between chemokines
CXCL12, CCL2, RANTES and early abortion. Zhonghua Yi Xue Za Zhi
2012;92:9-11
170.
Robbiani DF, Finch RA, Jäger D, Muller WA, Sartorelli AC,
Randolph GJ. The leukotriene C(4) transporter MRP1 regulates CCL19
(MIP-3beta, ELC)-dependent mobilization of dendritic cells to lymph nodes.
Cell 2000;103:757-68
171.
Buonamici S, Trimarchi T, Ruocco MG, Reavie L, Cathelin S, Mar
BG, Klinakis A, Lukyanov Y, Tseng JC, Sen F, Gehrie E, Li M, Newcomb
118
E, Zavadil J, Meruelo D, Lipp M, Ibrahim S, Efstratiadis A, Zagzag D,
Bromberg JS, Dustin ML, Aifantis I. CCR7 signalling as an essential
regulator of CNS infiltration in T-cell leukaemia. Nature 2009;459:1000-4
172.
Leick M, Catusse J, Follo M, Nibbs RJ, Hartmann TN, Veelken H,
Burger M. CCL19 is a specific ligand of the constitutively recycling atypical
human chemokine receptor CRAM-B. Immunology 2010;129:536-46
173.
Patel VP, Kreider BL, Li Y, Li H, Leung K, Salcedo T, Nardelli B,
Pippalla V, Gentz S, Thotakura R, Parmelee D, Gentz R, Garotta G.
Molecular and functional characterization of two novel human C-C
chemokines as inhibitors of two distinct classes of myeloid progenitors. J
Exp Med 1997;185:1163-72
174.
Menzies-Gow A, Ying S, Sabroe I, Stubbs VL, Soler D, Williams TJ,
Kay AB. Eotaxin (CCL11) and eotaxin-2 (CCL24) induce recruitment of
eosinophils, basophils, neutrophils, and macrophages as well as features of
early- and late-phase allergic reactions following cutaneous injection in
human atopic and nonatopic volunteers. J Immunol 2002;169:2712-8
175.
Min JW, Lee JH, Park CS, Chang HS, Rhim TY, Park SW, Jang AS,
Shin HD. Association of eotaxin-2 gene polymorphisms with plasma
eotaxin-2 concentration. J Hum Genet 2005;50:118-23
176.
Homey B, Dieu-Nosjean MC, Wiesenborn A, Massacrier C, Pin JJ,
Oldham E, Catron D, Buchanan ME, Müller A, deWaal Malefyt R, Deng G,
Orozco R, Ruzicka T, Lehmann P, Lebecque S, Caux C, Zlotnik A. Upregulation of macrophage inflammatory protein-3 alpha/CCL20 and CC
chemokine receptor 6 in psoriasis. J Immunol 2000;164:6621-32
177.
Reboldi A, Coisne C, Baumjohann D, Benvenuto F, Bottinelli D, Lira
S, Uccelli A, Lanzavecchia A, Engelhardt B, Sallusto F. C-C chemokine
receptor 6-regulated entry of TH-17 cells into the CNS through the choroid
plexus is required for the initiation of EAE. Nat Immunol 2009;10:514-23
119
178.
Ogilvie P, Bardi G, Clark-Lewis I, Baggiolini M, Uguccioni M.
Eotaxin is a natural antagonist for CCR2 and an agonist for CCR5. Blood
2001;97:1920-4
179.
Salcedo R, Young HA, Ponce ML, Ward JM, Kleinman HK, Murphy
WJ, Oppenheim JJ. Eotaxin (CCL11) induces in vivo angiogenic responses
by human CCR3+ endothelial cells. J Immunol 2001;166:7571-8
180.
Villeda SA, Luo J, Mosher KI, Zou B, Britschgi M, Bieri G, Stan TM,
Fainberg N, Ding Z, Eggel A, Lucin KM, Czirr E, Park JS, CouillardDesprés S, Aigner L, Li G, Peskind ER, Kaye JA, Quinn JF, Galasko DR,
Xie XS, Rando TA, Wyss-Coray T. The ageing systemic milieu negatively
regulates neurogenesis and cognitive function. Nature 2011;477:90-4
181.
Godiska R, Chantry D, Raport CJ, Sozzani S, Allavena P, Leviten D,
Mantovani A, Gray PW. Human macrophage-derived chemokine (MDC), a
novel chemoattractant for monocytes, monocyte-derived dendritic cells, and
natural killer cells. J Exp Med 1997;185:1595-604
182.
Toulza F, Nosaka K, Tanaka Y, Schioppa T, Balkwill F, Taylor GP,
Bangham CR. Human T-lymphotropic virus type 1-induced CC chemokine
ligand 22 maintains a high frequency of functional FoxP3+ regulatory T
cells. J Immunol 2010;185:183-9
183.
Cocchi F, DeVico AL, Garzino-Demo A, Arya SK, Gallo RC, Lusso
P. Identification of RANTES, MIP-1 alpha, and MIP-1 beta as the major
HIV-suppressive factors produced by CD8+ T cells. Science 1995;270:18115
184.
Pavkova Goldbergova M, Lipkova J, Pavek N, Gatterova J, Vasku A,
Soucek M, Nemec P. RANTES, MCP-1 chemokines and factors describing
rheumatoid arthritis. Mol Immunol 2012;52:273-8
185.
Arenzana-Seisdedos F, Virelizier JL, Rousset D, Clark-Lewis I,
Loetscher P, Moser B, Baggiolini M. HIV blocked by chemokine antagonist.
Nature 1996;383:400
120
186.
Bakhiet M, Tjernlund A, Mousa A, Gad A, Strömblad S, Kuziel WA,
Seiger A, Andersson J. RANTES promotes growth and survival of human
first-trimester forebrain astrocytes. Nat Cell Biol 2001;3:150-7
187.
Pritts EA, Zhao D, Ricke E, Waite L, Taylor RN. PPAR-gamma
decreases endometrial stromal cell transcription and translation of RANTES
in vitro. J Clin Endocrinol Metab 2002;87:1841-4
188.
Apolinario A, Majano PL, Alvarez-Pérez E, Saez A, Lozano C,
Vargas J, García-Monzón C. Increased expression of T cell chemokines and
their receptors in chronic hepatitis C: relationship with the histological
activity of liver disease. Am J Gastroenterol 2002;97:2861-70
189.
Tyner JW, Uchida O, Kajiwara N, Kim EY, Patel AC, O'Sullivan MP,
Walter MJ, Schwendener RA, Cook DN, Danoff TM, Holtzman MJ. CCL5CCR5 interaction provides antiapoptotic signals for macrophage survival
during viral infection. Nat Med 2005;11:1180-7
190.
Karnoub AE, Dash AB, Vo AP, Sullivan A, Brooks MW, Bell GW,
Richardson AL, Polyak K, Tubo R, Weinberg RA. Mesenchymal stem cells
within
tumour
stroma
promote
breast
cancer
metastasis.
Nature
2007;449:557-63
191.
Van Damme J, Proost P, Lenaerts JP, Opdenakker G. Structural and
functional identification of two human, tumor-derived monocyte chemotactic
proteins (MCP-2 and MCP-3) belonging to the chemokine family. J Exp
Med 1992;176:59-65
192.
Blaszczyk J, Coillie EV, Proost P, Damme JV, Opdenakker G, Bujacz
GD, Wang JM, Ji X. Complete crystal structure of monocyte chemotactic
protein-2, a CC chemokine that interacts with multiple receptors.
Biochemistry 2000;39:14075-81
193.
Bazan JF, Bacon KB, Hardiman G, Wang W, Soo K, Rossi D,
Greaves DR, Zlotnik A, Schall TJ. A new class of membrane-bound
chemokine with a CX3C motif. Nature 1997;385:640-4
121
194.
Lucas AD, Bursill C, Guzik TJ, Sadowski J, Channon KM, Greaves
DR. Smooth muscle cells in human atherosclerotic plaques express the
fractalkine receptor CX3CR1 and undergo chemotaxis to the CX3C
chemokine fractalkine (CX3CL1). Circulation 2003;108:2498-504
195.
Hannan NJ, Jones RL, Critchley HO, Kovacs GJ, Rogers PA, Affandi
B, Salamonsen LA. Coexpression of fractalkine and its receptor in normal
human endometrium and in endometrium from users of progestin-only
contraception supports a role for fractalkine in leukocyte recruitment and
endometrial remodeling. J Clin Endocrinol Metab 2004;89:6119-29
196.
Sims NA, Jenkins BJ, Nakamura A, Quinn JM, Li R, Gillespie MT,
Ernst M, Robb L, Martin TJ. Interleukin-11 receptor signaling is required for
normal bone remodeling. J Bone Miner Res 2005;20:1093-102
197.
Paiva P, Salamonsen LA, Manuelpillai U, Walker C, Tapia A,
Wallace EM, Dimitriadis E. Interleukin-11 promotes migration, but not
proliferation, of human trophoblast cells, implying a role in placentation.
Endocrinology 2007;148:5566-72
198.
Chen HF, Lin CY, Chao KH, Wu MY, Yang YS, Ho HN. Defective
production of interleukin-11 by decidua and chorionic villi in human
anembryonic pregnancy. J Clin Endocrinol Metab 2002;87:2320-8
199.
Wynn TA. IL-13 effector functions. Annu Rev Immunol 2003;21:425-
56. Review
200.
Chegini N, Ma C, Roberts M, Williams RS, Ripps BA. Differential
expression of interleukins (IL) IL-13 and IL-15 throughout the menstrual
cycle in endometrium of normal fertile women and women with recurrent
spontaneous abortion. J Reprod Immunol 2002;56:93-110
201.
Cruikshank WW, Kornfeld H, Center DM. Interleukin-16. J Leukoc
Biol 2000;67:757-66. Review
202.
Miossec P, Korn T, Kuchroo VK. Interleukin-17 and type 17 helper T
cells. N Engl J Med 2009;361:888-98
122
203.
Lee SK, Kim JY, Hur SE, Kim CJ, Na BJ, Lee M, Gilman-Sachs A,
Kwak-Kim J. An imbalance in interleukin-17-producing T and Foxp3!
regulatory T cells in women with idiopathic recurrent pregnancy loss. Hum
Reprod 2011;26:2964-71
204.
Al-Khateeb GM, Sater MS, Finan RR, Mustafa FE, Al-Busaidi AS,
Al-Sulaiti MA, Almawi WY. Analysis of interleukin-18 promoter
polymorphisms and changes in interleukin-18 serum levels underscores the
involvement of interleukin-18 in recurrent spontaneous miscarriage. Fertil
Steril 2011;96:921-6
205.
Oppmann B, Lesley R, Blom B, Timans JC, Xu Y, Hunte B, Vega F,
Yu N, Wang J, Singh K, Zonin F, Vaisberg E, Churakova T, Liu M, Gorman
D, Wagner J, Zurawski S, Liu Y, Abrams JS, Moore KW, Rennick D, de
Waal-Malefyt R, Hannum C, Bazan JF, Kastelein RA. Novel p19 protein
engages IL-12p40 to form a cytokine, IL-23, with biological activities
similar as well as distinct from IL-12. Immunity 2000;13:715-25
206.
Wang M, Liang P. Interleukin-24 and its receptors. Immunology
2005;114:166-70
207.
Sokol CL, Barton GM, Farr AG, Medzhitov R. A mechanism for the
initiation of allergen-induced T helper type 2 responses. Nat Immunol
2008;9:310-8
208.
Sanderson CJ. Interleukin-5, eosinophils, and disease. Blood
1992;79:3101-9. Review
209.
Renauld JC, Houssiau F, Druez C, Uyttenhove C, Vink A, Van Snick
J. Interleukin-9. Int Rev Exp Pathol 1993;34:99-109. Review
210.
Gauchat JF, Aubry JP, Mazzei G, Life P, Jomotte T, Elson G,
Bonnefoy JY. Human CD40-ligand: molecular cloning, cellular distribution
and regulation of expression by factors controlling IgE production. FEBS
Lett 1993;315:259-66
123
211.
DiSanto JP, Bonnefoy JY, Gauchat JF, Fischer A, de Saint Basile G.
CD40 ligand mutations in x-linked immunodeficiency with hyper-IgM.
Nature 1993;361:541-3
212.
Stanley ER, Berg KL, Einstein DB, Lee PS, Pixley FJ, Wang Y,
Yeung YG. Biology and action of colony--stimulating factor-1. Mol Reprod
Dev 1997;46:4-10. Review
213.
Gout I, Dhand R, Panayotou G, Fry MJ, Hiles I, Otsu M, Waterfield
MD. Expression and characterization of the p85 subunit of the
phosphatidylinositol 3-kinase complex and a related p85 beta protein by
using the baculovirus expression system. Biochem J 1992;288:395-405
214.
Schoenborn JR, Wilson CB. Regulation of interferon-gamma during
innate and adaptive immune responses. Adv Immunol 2007;96:41-101.
Review
215.
Akirav E, Liao S, Ruddle N. “Chapter 2: Lymphoid Tissues and
Organs”. In Paul, William (Book). Fundamental Immunology (6th ed.).
Philadelphia, PA: Lippincott Williams & Wilkins. 2008; pp 27-55
216.
Hamada H, Meno C, Watanabe D, Saijoh Y. Establishment of
vertebrate left-right asymmetry. Nat Rev Genet 2002;3:103-13. Review
217.
Zhou X, Sasaki H, Lowe L, Hogan BL, Kuehn MR. Nodal is a novel
TGF-beta-like gene expressed in the mouse node during gastrulation. Nature
1993;361:543-7
218.
Saito S. Cytokine network at the feto-maternal interface. J Reprod
Immunol 2000;47:87-103. Review
219.
Mallmann P, Mallmann R, Krebs D. Determination of tumor necrosis
factor alpha (TNF alpha) and interleukin 2 (IL 2) in women with idiopathic
recurrent miscarriage. Arch Gynecol Obstet 1991;249:73-8
220.
Kilpatrick
DC.
Soluble
interleukin-2
receptors
in
recurrent
miscarriage and the effect of leukocyte immunotherapy. Immunol Lett
1992;34:201-6
124
221.
Wilson R, McInnes I, Leung B, McKillop JH, Walker JJ. Altered
interleukin 12 and nitric oxide levels in recurrent miscarriage. Eur J Obstet
Gynecol Reprod Biol 1997;75:211-4
222.
Vassiliadis S, Ranella A, Papadimitriou L, Makrygiannakis A,
Athanassakis I. Serum levels of pro- and anti-inflammatory cytokines in nonpregnant women, during pregnancy, labour and abortion. Mediators Inflamm
1998;7:69-72
223.
Koumantaki Y, Matalliotakis I, Sifakis S, Kyriakou D, Neonaki M,
Goymenou A, Koumantakis E. Detection of interleukin-6, interleukin-8, and
interleukin-11 in plasma from women with spontaneous abortion. Eur J
Obstet Gynecol Reprod Biol 2001;98:66-71
224.
MacLean MA, Wilson R, Thomson JA, Krishnamurthy S, Walker JJ
Am J. Changes in immunologic parameters in normal pregnancy and
spontaneous abortion. Obstet Gynecol 1991;165:890-5
225.
Hristov M, Zernecke A, Bidzhekov K, Liehn EA, Shagdarsuren E,
Ludwig A, Weber C. Importance of CXC chemokine receptor 2 in the
homing of human peripheral blood endothelial progenitor cells to sites of
arterial injury. Circ Res 2007;100:590-7
226.
Kiessling S, Muller-Newen G, Leeb SN, Hausmann M, Rath HC,
Strater J, Spottl T, Schlottmann K, Grossmann J, Montero-Julian FA,
Scholmerich J, Andus T, Buschauer A, Heinrich PC, Rogler G. Functional
expression of the interleukin-11 receptor alpha-chain and evidence of
antiapoptotic effects in human colonic epithelial cells. J Biol Chem
2004;279:10304-15
227.
Babensee JE, McIntire LV, Mikos AG. Growth factor delivery for
tissue engineering. Pharm Res 2000;17:497-504. Review
228.
Chen H, Shi S, Acosta L, Li W, Lu J, Bao S, Chen Z, Yang Z,
Schneider MD, Chien KR, Conway SJ, Yoder MC, Haneline LS, Franco D,
125
Shou W. BMP10 is essential for maintaining cardiac growth during murine
cardiogenesis. Development 2004;131:2219-31
229.
Suzuki A, Thies RS, Yamaji N, Song JJ, Wozney JM, Murakami K,
Ueno N. A truncated bone morphogenetic protein receptor affects dorsalventral patterning in the early Xenopus embryo. Proc Natl Acad Sci U S A
1994;91:10255-9
230.
Steinbeisser H, Fainsod A, Niehrs C, Sasai Y, De Robertis EM. The
role of gsc and BMP-4 in dorsal-ventral patterning of the marginal zone in
Xenopus: a loss-of-function study using antisense RNA. EMBO J
1995;14:5230-43
231.
Serra R, Johnson M, Filvaroff EH, LaBorde J, Sheehan DM, Derynck
R, Moses HL. Expression of a truncated, kinase-defective TGF-beta type II
receptor
in
mouse
skeletal
tissue
promotes
terminal
chondrocyte
differentiation and osteoarthritis. J Cell Biol 1997;139:541-52
232.
Paradisi R, Porcu E, Venturoli S, Maldini-Casadei M, Boni P.
Maternal serum levels of pro-inflammatory cytokines in missed and
threatened abortion. Am J Reprod Immunol 2003;50:302-8
233.
Schmitz J, Owyang A, Oldham E, Song Y, Murphy E, McClanahan
TK, Zurawski G, Moshrefi M, Qin J, Li X, Gorman DM, Bazan JF, Kastelein
RA. IL-33, an interleukin-1-like cytokine that signals via the IL-1 receptorrelated protein ST2 and induces T helper type 2-associated cytokines.
Immunity 2005;23:479-90
234.
Kapessidou P, Poulin L, Dumoutier L, Goldman M, Renauld JC,
Braun MY. Interleukin-22 deficiency accelerates the rejection of full major
histocompatibility complex-disparate heart allografts. Transplant Proc
2008;40:1593-7
235.
Hueng DY, Lin GJ, Huang SH, Liu LW, Ju DT, Chen YW, Sytwu
HK, Chang C, Huang SM, Yeh YS, Lee HM, Ma HI. Inhibition of Nodal
126
suppresses angiogenesis and growth of human gliomas. J Neurooncol
2011;104:21-31
236.
Brown S, Teo A, Pauklin S, Hannan N, Cho CH, Lim B, Vardy L,
Dunn NR, Trotter M, Pedersen R, Vallier L. Activin/Nodal signaling
controls divergent transcriptional networks in human embryonic stem cells
and in endoderm progenitors. Stem Cells 2011;29:1176-85
237.
Nadeem L, Munir S, Fu G, Dunk C, Baczyk D, Caniggia I, Lye S,
Peng C. Nodal signals through activin receptor-like kinase 7 to inhibit
trophoblast migration and invasion: implication in the pathogenesis of
preeclampsia. Am J Pathol. 2011;178:1177-89
238.
Dimitriadis E, Nie G, Hannan NJ, Paiva P, Salamonsen LA. Local
regulation of implantation at the human fetal-maternal interface. Int J Dev
Biol 2010;54:313-22. Review.
239.
Laird SM, Tuckerman EM, Cork BA, Linjawi S, Blakemore AI, Li
TC. A review of immune cells and molecules in women with recurrent
miscarriage. Hum Reprod Update 2003;9:163-74
127