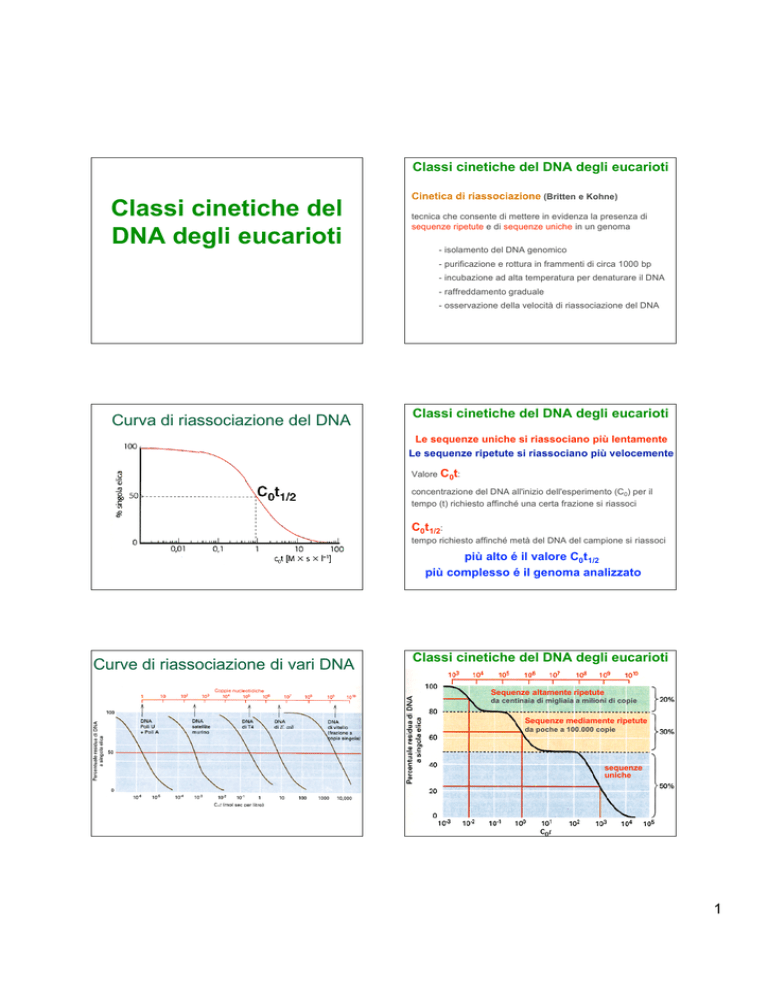
Classi cinetiche del DNA degli eucarioti
Classi cinetiche del
DNA degli eucarioti
Cinetica di riassociazione (Britten e Kohne)
tecnica che consente di mettere in evidenza la presenza di
sequenze ripetute e di sequenze uniche in un genoma
- isolamento del DNA genomico
- purificazione e rottura in frammenti di circa 1000 bp
- incubazione ad alta temperatura per denaturare il DNA
- raffreddamento graduale
- osservazione della velocità di riassociazione del DNA
Curva di riassociazione del DNA
Classi cinetiche del DNA degli eucarioti
Le sequenze uniche si riassociano più lentamente
Le sequenze ripetute si riassociano più velocemente
Valore C0t:
concentrazione del DNA all'inizio dell'esperimento (C0) per il
tempo (t) richiesto affinché una certa frazione si riassoci
C0t1/2:
tempo richiesto affinché metà del DNA del campione si riassoci
più alto é il valore C0t1/2
più complesso é il genoma analizzato
Curve di riassociazione di vari DNA
Classi cinetiche del DNA degli eucarioti
Sequenze altamente ripetute
da centinaia di migliaia a milioni di copie
Sequenze mediamente ripetute
da poche a 100.000 copie
sequenze
uniche
1
Sequenze uniche e sequenze ripetute
Le curve di C0t eucariotiche presentano tre fasi con diverso valore di C0t
Sequenze altamente ripetute
da centinaia di migliaia a milioni di copie
Tipicamente raggruppate in cluster in una o poche regioni cromosomiche
- prima curva
C0t = 10-2
I cluster possono:
- contenere ripetizioni in tandem di una singola sequenza
- avere combinazioni regolarmente alternate di molte sequenze
- alcuni cluster sono "puri"
- essere interrotti da una o più ripetizioni di elementi mediamente ripetuti
- seconda curva C0t = 1
- terza curva
C0t = 103
la differenza é dovuta alla presenza di DNA con caratteristiche
cinetiche diverse cioé con grado di ripetizione diverso
Sequenze altamente ripetute
Satellite alfa
l'unità ripetuta é di 171 bp
presente in tutti i centromeri
contiene una sequenza di 17 bp che lega
CENP-B detta CENP-B box
5' CTTCGTTGGAAACGGGA 3'
funzionalmente é il più importante
La cromatina corrispondente a sequenze altamente ripetute
é molto compatta nelle cellule interfasiche
é trascrizionalmente inattiva
Sequenze mediamente ripetute
Sequenze funzionali:
Geni per gli rRNA
geni per i tRNA
geni per gli istoni
Sequenza AAUAAA (poliadenilazione)
Sequenza telomeriche
Sequenze non funzionali
Sequenze disperse nel genoma o raggruppate in cluster in uno o più siti
Sono presenti in famiglie i cui singoli membri variano leggermente in
sequenza
Un alto numero di sequenze mediamente ripetute negli animali sono
fiancheggiate da brevi elementi ripetuti diretti
Indicazione che essi sono elementi mobili che possono spostarsi in altri punti del genoma
Sequenze mediamente ripetute
da poche a 100.000 copie
PSEUDOGENI
copie inattive di geni
SINE
Short INterspersed Elements
LINE
Long INterspersed Elements
Pseudogeni
Molte famiglie geniche comprendono sequenze attive e sequenze inattive
I componenti inattivi delle famiglie geniche sono detti: pseudogeni
A
*
A
A2
A1
A2
A3
A4
A3
Pseudogeni del tipo non processato o non maturato
si producono per duplicazione di un gene
il nuovo gene accumula mutazioni che lo rendono non funzionale
generalmente i geni di una famiglia sono raggruppati in cluster
ad esempio: globine, immunoglobuline
Pseudogeni del tipo processato o maturato
si producono per retrotrascrizione di un RNA messaggero
correlati agli mRNA maturi (senza introni)
mancano le sequenze 5' e 3' del gene principale
contengono una serie di basi A (derivate dalla coda di poli-A)
mancando le sequenze in 5' non vengono trascritti
i componenti di una famiglia sono dispersi in tutto il genoma
2
Tipo processato
Pseudogeni
possono comparire istantaneamente
il citocromo-c e la metallotionina possiedono pseudogeni che sono la copia
perfetta dei loro mRNA (inserimento nel genoma molto recente)
la beta-tubulina possiede tre pseudogeni molto diversi dal gene originario,
infatti sono comparsi 4, 11.5, 13 milioni di anni fa
sono ristretti quasi esclusivamente ai mammiferi
ca. il 20% del genoma dei mammiferi é rappresentato da pseudogeni maturati
nelle cellule dei mammiferi é elevata l'infezione da retrovirus
presenza di trascrittasi inversa
presenza di enzimi per l'inserimento del DNA nei genomi
Pseudogeni del tipo processato o maturato
si producono per retrotrascrizione di un RNA messaggero
correlati agli mRNA maturi (senza introni)
mancano le sequenze 5' e 3' del gene principale
contengono una serie di basi A (derivate dalla coda di poli-A)
mancando le sequenze in 5' non vengono trascritti
i componenti di una famiglia sono dispersi in tutto il genoma
Famiglie geniche con geni
raggruppati
Tipo non processato
sono comparsi molto tempo addietro: 1-2 milioni di anni
affinché un gene diventi non funzionale deve accumulare moltissime mutazioni
sono distribuiti in tutti gli eucarioti
Organizzazione dei geni omeotici (geni HOX)
AbdB
AbdA Ubx
Antp Scr Dfd zz
?
A13
10
A11 A10
9
8
A9
7
6
5
4
A7
A6 A5 A4
zi pb
3
2
A3 A2
lab
1
A1
Drosophila
Anfiosso
topo
HOXA
B9
B8 B7
B6 B5 B4
B3 B2
B1
HOXB
Famiglie geniche con geni
distribuiti nel genoma
C13
C12 C11 C10
C9
C8
C6 C5 C4
D13
D12 D11 D10
D9
D8
D4
HOXC
D3
D1
HOXD
Principali famiglie di DNA
ripetitivo intersperso
3
Sequenze LINE (Long INterspersed Elements)
Struttura di un elemento L1 di mammifero
6-7 Kb
Sequenze codificanti
Presenti nei Primati e in altri mammiferi
brevi ripetizioni dirette
(elementi mobili)
Elementi lunghi 6.000-7.000 bp
Ogni elemento completo contiene molte sequenze
che codificano proteine
Può essere presente una trascrittasi inversa attiva
Poche sequenze LINE sono complete
Come si origina una sequenza LINE?
p40
breve
segmento
di poli(A)
Trascrittasi inversa
La trascrittasi inversa sembra essere implicata
nella trasposizione dell’elemento L1 stesso
Sequenze LINE (Long INterspersed Elements)
Nell'uomo le ripetizioni LINE sono
50.000-100.000 (ca 5% del genoma)
# Disperse nei cromosomi
# Localizzate tra i geni e negli introni
# Osservate anche all'interno di
cluster di sequenze altamente ripetute
Sequenze LINE (Long INterspersed Elements)
Inserzioni recenti nel genoma umano
In due bambini emofilici é stata osservata una inserzione di
una sequenza LINE all'interno di un gene necessario per la
normale coagulazione del sangue: l'inserzione era assente
nei genitori
4
Sequenze SINE (Short INterspersed Elements)
Le SINE sono in numero più alto rispetto alla media
degli elementi mediamente ripetuti
100.000 - 500.000 copie in alcuni Primati
(3-8% del DNA totale)
Sequenze SINE (Short INterspersed Elements)
Famiglia Alu
presente nei Primati
identificata mediante l'enzima Alu I che taglia
all'interno dell’elemento ripetuto
un elemento Alu é lungo ca. 300 bp
composto da due ripetizioni non uguali di una sequenza simile
300.000 - 500.000 copie nel genoma umano
(6-8% del DNA totale)
alla regione codificante dell' scRNA
cioé una sequenza SINE ogni 5-9 kb
SRP 7SL
7SL-RNA
Elemento Alu
7SL-RNA
7SL-RNA
Come si sono originate le ripetizioni Alu?
Sequenze Alu
Sequenza
pseudogene
ripetuta
per l’scRNA 7SL
diretta six
130 bp
AAAAAA-TTTTTT--
pseudogene
per l’scRNA 7SL
160 bp
Inserzione di 31 bp
Sequenza
ripetuta
diretta dex
AAAAAA-TTTTTT--
molte Alu sono troncate e mancano di alcuni elementi
ca l'80% degli elementi Alu é fiancheggiata da brevi
sequenze ripetute dirette, elementi tipici delle sequenze
mobili
sono presenti anche negli introni in ca il 25% dei geni
la sequenza Alu più completa presenta un promotore interno
ma solo poche Alu sono trascritte
5
Trasposizione delle sequenze Alu
Trasposizione di sequenze Alu
Le sequenze Alu non sono autonome ma si osservano inserzioni
di Alu continuamente nel genoma umano
Sembra che la trasposizione delle sequenze Alu avvenga per
mezzo degli enzimi codificati dalle sequenze L1
velocità di circa una inserzione ogni 100 anni
in un caso di neurofibromatosi, il paziente
presentava l'inserzione di una sequenza Alu nel
gene NF1 che era assente nei genitori indicando
che essa era avvenuta nel paziente stesso
Distribuzione di elementi L1 e SINE
nel cluster della β-globina umana
Telomero (TTAGGG)n
Diverse Kb di DNA
Distribuzione
delle sequenze
ripetute più
comuni
Centromero: vari tipi di DNA satellite
Diverse Mb di DNA
LINE-1: preferenzialmente nelle bande G scure
Alu-1: preferenzialmente nelle bande G chiare
Telomero (TTAGGG)n
6










