1
UnasoluzionecontenenteunamisceladiNADPHeNAD+vieneanalizzata
spettrofotometricamente.L’assorbanzaditalemiscela,misuratainunacuvettada1cmdi
lunghezza,èparia:
A260nm=0,85
A340nm=0,11
CalcolarelaconcentrazionediNADPHeNAD+contenutinellamiscelae,sapendoilpeso
molecolaredelleduesostanze(PMnad+=663.4g/mol,PMnadph=744.4g/mol),laquantitàin
microgrammipresentinellasoluzionedipartenza,ilcuivolumetotaleeraparia58ml.I
coefficientidiestinzionedelNADPHedelNAD+allelunghezzed’ondasoprariportatesonoi
seguenti:
ε
(M-1cm-1)
Composto
260nm
340nm
NAD+
16900
~0
NADPH
16900
6220
2
Analizzareiseguentidatidicineticaenzimatica(veditabellasottostante),utilizzandoilsia
ilgraficodelleiperbolicheilgraficodeidoppireciproci.Le“velocitàiniziali2”sonostate
misurareinpresenzadiunsostanzaaggiuntanelsaggioenzimaticoallaconcentrazionedi50
µM.Qualefenomenostiamoosservandoinpresenzaditalesostanza?Dalgraficodeidoppi
reciprocimisuratesperimentalmentelecostanticineticheapparenti(KmeVmax)ediscutetei
risultati.
Concentrazione
disubstrato[S]
Velocitàiniziale1
(Vi)
Velocitàiniziale2
(Vi)
1/[S] 1/[Vi]1 1/[Vi]2
(?)
(?)
(?)
(M•10-3)
(mmoli•L-1•min-1)
(mmoli•L-1•min-1)
0,020
0,157
0,066
0,035
0,201
0,095
0,050
0,232
0,125
0,100
0,275
0,182
0,300
0,334
0,267
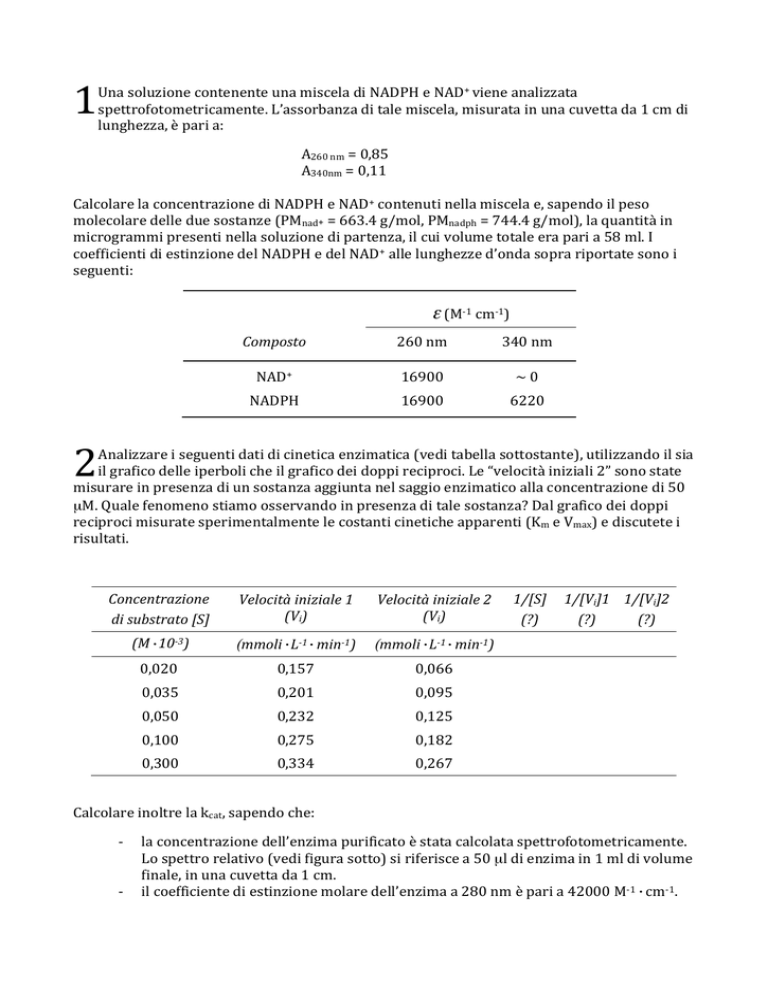
Calcolareinoltrelakcat,sapendoche:
-
laconcentrazionedell’enzimapurificatoèstatacalcolataspettrofotometricamente.
Lospettrorelativo(vedifigurasotto)siriferiscea50µldienzimain1mldivolume
finale,inunacuvettada1cm.
ilcoefficientediestinzionemolaredell’enzimaa280nmèparia42000M-1•cm-1.
inognisaggioenzimaticosonostatiutilizzati5µldiunasoluzionediluita250volte
dienzima,inunvolumefinaledi600µlincuvetta.
0.35
0.30
0.25
0.20
0.15
0.10
0.05
0.00
250
260
270
280
290
300
310
320
Lunghezze d'onda (nm)
3
InAllegato 1èfornitalamappadelcDNA(piùregionifiancheggianti)codificanteperla
treonina sintasidiEscherichiacoli,cheaveteadisposizioneinunaprovettadaqualche
parteinfondoalfreezer.Lastrutturaprimariadellaproteinaèlaseguente:
1
MKLYNLKDHNEQVSFAQAVTQGLGKNQGLFFPHDLPEFSLTEIDEMLKLDFVTRSAKILSAFIGDEIPQEILEERVRAAFA
FPAPVANVESDVGCLELFHGPTLAFKDFGGRFMAQMLTHIAGDKPVTILTATSGDTGAAVAHAFYGLPNVKVVILYPRGKI
SPLQEKLFCTLGGNIETVAIDGDFDACQALVKQAFDDEELKVALGLNSANSINISRLLAQICYYFEAVAQLPQEARNQLVV
SVPSGNFGDLTAGLLAKSLGLPVKRFIAATNVNDTVPRFLHDGQWSPKATQATLSNAMDVSQPNNWPRVEELFRRKIWQLK
ELGYAAVDDETTQQTMRELKELGYTSEPHAAVAYRALRDQLNPGEYGLFLGTAHPAKFKESVEAILGETLDLPKELAERAD
LPLLSHNLPADFAALRKLMMNHQ428
Evidenziatesullamappainallegatoilcodonediiniziodellaregionecodificanteeilcodone
distop.Verificateditrovarvinellaregionecodificantetraducendoiprimi5codoniegliultimi
5,utilizzandoilcodonusage(Allegato2).
Disegnatedeglioligonucleotidi(dellalunghezzadi25-30nucleotidi)cheservanodaprimer
perunareazionediPCRperl’amplificazionedelcDNAdellatreoninasintasi,inmododapoter
poi clonare tale cDNA dentro al plasmide pET22b(+) (mappa in Allegato 3), tra i siti di
restrizione NdeI e EcoRI. È possibile utilizzare questi due enzimi di restrizione per il
clonaggio?AltrimentiutilizzateXhoIcomesitodiclonaggioavalle.Clonatetuttalasequenza
dellatreoninasintasi,comprendendosialametioninainizialecheilcodonedistop.
Il punto isoelettrico della treonina sintasi è pari a 5.71. Se per la purificazione della
proteina voleste utilizzare una resina a scambio ionico, facendo in modo che la proteina si
attacchiallaresinainuntampone20mMpotassiofosfatopH7.2,cheresinaascambioionico
scegliereste: una CM-sephadex (carbossimetil-sephadex, scambio cationico) o una DEAEsepharosio (dietilamminetil-sepharosio, scambio anionico)? Che tampone utilizzereste per
eluirepoilaproteinadallaresina?
4
UnasoluzionecontieneunamisceladiNADHeATPvengonoanalizzati
spettrofotometricamente.L’assorbanzaditalemiscela,misuratainunacuvettada1cmdi
lunghezza,èparia:
A260nm=0,89
A340nm=0,12
CalcolarelaconcentrazionediNADPHeATPcontenutinellamiscela,esapendoilpeso
molecolaredelleduesostanze(PMatp=507.1g/mol,PMnadph=664.3g/mol),laquantitàin
microgrammipresentinellasoluzionedipartenza,ilcuivolumetotaleeraparia36ml.I
coefficientidiestinzionedelNADPHedell’ATPallelunghezzed’ondasoprariportatesonoi
seguenti:
εmol(M-1cm-1)
Composto
260nm
340nm
ATP
15400
~0
NADH
16900
6220
5
L’AspartatoaminotransferasidafegatoeluiscedaunacromatografiaBiogelP-300(gelfiltrazione)aunaposizionecorrispondentealpesomolecolaredi90kDa.Lastessa
proteina,sottopostaaelettroforesiinpresenzadiSDS,mostraunpesomolecolaredi45kDa.
Unsaggiosuuncampionediaspartatoaminotrasferasimostralapresenzadi5,43µgdi
piridossale5’-fosfato(PM=247,1)permilligrammodiproteina.
Checonclusionisipossonotrarresullastrutturadell’aspartatoaminotransferasi?
6
IltessutoepaticoembrionalecontieneunenzimachecatalizzalareazioneSèP.Ancheil
tessutoepaticoadultomostrataleattivitàenzimatica.Analizzareidatidiattività
enzimaticarelativiaidueenzimi(veditabellasottostante),utilizzandosiailgraficoVivs[S]
cheilgraficodeidoppireciproci.Determinare,dalgraficodeidoppireciproci,laKmelaVmax).
Chedifferenzac’èfral’enzimaembrionaleequelloadulto?
Concentrazioneiniziale
disubstrato[S]
Velocitàiniziale(Vi)
(µmoli•L-1•min-1)
(M•10-4)
Enzima adulto
Enzima embrionale
1,05
1,54
1,98
2,86
3,78
5,00
5,00
6,66
8,00
10,00
11,67
13,33
0,16
0,25
0,33
0,5
0,7
1
1,5
1,67
2,0
3,0
6,67
7,15
8,00
10,00
15,0
15,4
16,0
17,1
Inoltre,calcolarelakcat,sapendoche:
-
laconcentrazionedeglienzimipurificatièstatacalcolataspettrofotometricamente.
Glispettrirelativi(vedifiguresotto)siriferisconoa15µldienzimain1mldi
volumefinale,inunacuvettada1cm.
ilcoefficientediestinzionemolaredell’enzimaadultoa280nmèparia46000M-1•
cm-1,mentrequellodell’enzimaembrionaleèparia32000M-1•cm-1.
inognisaggioenzimaticosonostatiutilizzati3µldiunasoluzionediluita50volte
dienzima,inunvolumefinaledi600µlincuvetta.
Enzimaadulto
0.45
0.40
0.35
0.30
0.25
0.20
0.15
0.10
0.05
0.00
250
260
270
280
290
300
310
Lunghezza d'onda (nm)
0.14
Enzimaembrionale
0.12
0.10
0.08
0.06
0.04
0.02
0.00
250
270
280
290
Lunghezza d'onda (nm)
260
300
310
7
Diecigrammidiburrosonostatisaponificati;lafrazionenonsaponificabileèstata
estrattain50mldicloroformio.L’assorbanzaditalefrazione,misuratainunacuvettada1
cmdilunghezza,èparia:
A328nm=0,27
A458nm=0,24
CalcolarelaquantitàdicaroteneevitaminaAcontenutinelpanettooriginariodiburro(inµg
disostanzapergrammodiburro).IcoefficientidiestinzionedelcaroteneedellavitaminaA
allelunghezzed’ondasoprariportatesonoiseguenti:
a1%inCHCl3
Composto
328nm
458nm
Carotene
340
2200
VitaminaA
1550
~0
8
LaglicogenofosforilasidamuscoloeluiscedaunacromatografiaBiogelP-300
(cromatografiapergel-filtrazione)aunaposizionecorrispondentealpesomolecolaredi
360kDa.Lastessaproteina,sottopostaaelettroforesiinpresenzadiSDS,mostraunpeso
molecolaredi90kDa.Unsaggiosuuncampionedellaglicogenofosforilasimostralapresenza
di2,75µgdipiridossale(PM=247,1)permilligrammodiproteina.
Checonclusionisipossonotrarresullastrutturadellaglicogenofosforilasi?
9
Analizzareiseguentidatidicineticaenzimatica(veditabellasottostante),utilizzandoil
graficodeidoppireciproci.Qualeinaspettatofenomenostiamoosservando?Èpossibile
stimarelecostanticinetiche(KmeVmax)?
Concentrazione
iniziale
disubstrato[S]
Velocità
Iniziale(Vi)
1/[S]
(?)
1/[Vi]
(?)
(M 10-3)
•
0,6
1,3
2,5
5,0
10,0
20,0
(µmoli•L-1•min-1)
13,26
24,50
37,45
51,15
48,08
40,50
40,0
33,08
DopoaverstimatolaVmax,calcolarelakcat,sapendoche:
-
laconcentrazionedell’enzimapurificatoèstatacalcolataspettrofotometricamente.
Lospettrorelativo(vedifigurasotto)siriferiscea25µldienzimain800µldi
volumefinale,inunacuvettada1cm.
ilcoefficientediestinzionemolaredell’enzimaa280nmèparia31800M-1•cm-1.
inognisaggioenzimaticosonostatiutilizzati5µldiunasoluzionediluita150volte
dienzima,inunvolumefinaledi1000µlincuvetta.
10
InAllegato4èfornitalamappadelcDNA(piùregionifiancheggianti)codificanteper
lapiridossalkinasiumana(hPLK),cheaveteadisposizioneinunaprovettadaqualche
parteinfondoalfreezer.Lastrutturaprimariadellaproteinaèlaseguente:
1
MEEECRVLSIQSHVIRGYVGNRAATFPLQVLGFEIDAVNSVQFSNHTGYAHWKGQVLNSDELQELYEGLR
LNNMNKYDYVLTGYTRDKSFLAMVVDIVQELKQQNPRLVYVCDPVLGDKWDGEGSMYVPEDLLPVYKEKV
VPLADIITPNQFEAELLSGRKIHSQEEALRVMDMLHSMGPDTVVITSSDLPSPQGSNYLIVLGSQRRRNP
AGSVVMERIRMDIRKVDAVFVGTGDLFAAMLLAWTHKHPNNLKVACEKTVSTLHHVLQRTIQCAKAQAGE
GVRPSPMQLELRMVQSKRDIEDPEIVVQATVL312
Evidenziatesullamappainallegatoilcodonediiniziodellaregionecodificante(èilprimo
ATGcheincontrate)eilcodonedistop.Verificateditrovarvinellaregionecodificante
traducendoiprimi5codoniegliultimi5,utilizzandoilcodonusage(Allegato2).
VogliamoinserireilcDNAcodificanteperlahPLKnelvettorepET22(mappainAllegato3),
traisitidirestrizioneNdeIeEcoRI.Èunasceltaopportunaobisognasceglierealtrienzimidi
restrizione?PerprimacosabisognaamplificareilcDNA,inserendoalleestremitàisitidi
restrizioneadeguati.Disegnareglioligonucleotidi(20-30nucleotididilunghezza)da
utilizzarecomeprimerperlareazionediPCR.Disegnateduecoppiediprimer,unaper
eliminarelacodadipoli-HisalC-terminaledellaproteina,el’altrapermantenerelacodadi
istidine.Attenzioneanonandarefuoriframe.
11
Analizzareiseguentidatidicineticaenzimatica(veditabellasottostante),utilizzando
ilgraficoVivs[S].Determinaresel’enzimaobbedisceaunacineticaiperbolicao
sigmoidale,magariingrandendolaparteinizialedelgrafico.Stimarelecostanticinetiche(Km
o[S]0,5*eVmax).
*La[S]0.5equivaleallaKmperunenzimachemostracooperatività(andamentosigmoidedella
curva).
Concentrazioneiniziale
disubstrato[S]
Velocità
Iniziale(Vi)
(M•10-4)
(µmoli•L-1•min-1)
1,54
5,88
20,0
50,0
80,0
94,12
98,46
99,61
6,25
12,5
25,0
50,0
100,0
200,0
400,0
800,0
Inoltre,calcolarelakcat,sapendoche:
-
laconcentrazionedell’enzimapurificatoèstatacalcolataspettrofotometricamente.
Lospettrorelativo(vedifigurasotto)siriferiscea50µldienzimain800µldi
volumefinale,inunacuvettada1cm.
ilcoefficientediestinzionemolaredell’enzimaa280nmèparia53000M-1•cm-1.
inognisaggioenzimaticosonostatiutilizzati2µldiunasoluzionediluita25volte
dienzima,inunvolumefinaledi600µlincuvetta.
0.35
0.30
0.25
0.20
0.15
0.10
0.05
0.00
250
260
270
280
290
300
310
Lunghezze d'onda (nm)
320
12
Cercateinbancadati(PubMedNucleotide)lasequenzadelcDNAcodificanteperla
homoserinekinasediEscherichiacoli.Sitrattadell’enzimacon
“protein_id=AAN78504.1"inPubMedProtein.MappatelasequenzadicDNAutilizzando
Webcutter2econtrollatechecodifichieffettivamenteperlahomoserinekinase(utilizzandoil
codonusagediE.coli,Allegato2).
Chereazionecatalizzal’enzima?Scrivetelestrutturedisubstratieprodotti.Qualèl’“E.C.
number”dell’enizma?Dicheviabiosinteticafaparte?
DisegnatedeglioligonucleotidicheservanodaprimerperunareazionediPCRper
l’amplificazionedelcDNAdellahomoserinekinase,inmododapoterpoiclonaretalecDNA
dentroalplasmidepET22b(+)(Allegato3),traisitidirestrizioneNdeIeXhoI.Èpossibile
utilizzarequestidueenzimidirestrizioneperilclonaggio?Ipotizzateduediversiclonaggi.Nel
primodovreteaggiungereallahomoserinekinasi,inposizioneC-terminale,lacodadiistidine
presentenelplasmide.Nelsecondoclonaggioinvecedovetefareinmodochelacodadi
istidinenonvengatradotta.
13
LaserinaidrossimetiltransferasiumanaeluiscedaunacromatografiaBiogelP-300a
unaposizionecorrispondentealpesomolecolaredi200-220kDa.Lastessaproteina,
sottopostaaelettroforesiinpresenzadiSDS,mostraunpesomolecolaredicirca53kDa.Un
saggiosuuncampionediserinaidrossimetiltrasferasimostralapresenzadi3,85µgdi
piridossale5’-fosfato(PM=247,1)permilligrammodiproteina.
Checonclusionisipossonotrarresullastrutturadellaserinaidrossimetiltrasferasi?discutete
lastechiometriadelpiridossale5’-fosfato:checosapotrebbeesseresuccessodurante
l’analisi?