Mutazioni &
Mutagenesi
Importanza delle mutazioni
Le mutazioni costituiscono uno degli strumenti più importanti per
comprendere la funzione dei geni.
Per ottenere mutazioni si utilizzano:
- agenti mutageni (raggi X, EMS)
Mutazioni casuali, difficoltà di screening, no gene-specifico
- mutagenesi in vitro sequenza-specifica
Mutazioni sito-specifiche in modo da migliorare o alterare le caratteristiche
delle proteine (ingegneria proteica), diverse strategie di screening
- elementi inserzionali
Mutazioni di tipo KnockOut, recessive, casuali
Tipi di Mutazioni
- Puntiformi (silenti, missenso, nonsenso)
- Inserzioni/delezioni (frameshift?? Se non 3N)
Mutagenesi sito-specifica
per estensione del primer
Questo sistema utilizza uno
stampo di DNAss del gene da
mutare. Il primer si lega alla
sua sequenza complementare
introducendo una o più
mutazioni specifiche. L ibrido
viene poi trattato con DNApol.
e dNTP, per sintetizzare un
filamento complementare che
porta le basi mutate. Il
filamento ds così creato è
trasformato in cellule di E.coli.
In E.coli si avrà replicazione
sia di copie del plasmide wild
type che del plasmide
mutagenizzato.
Screening con oligonucletoide
ad alta stringenza
Mutagenesi sito-specifica
con il metodo dut- ungUn metodo più efficiente per effettuare mutagenesi sito-specifica è il
metodo dut- ung- in cui il filamento wild type è selettivamente
indirizzato verso la degradazione.
Si ottiene facendo crescere il fago filamentoso M13 in un ceppo di
E.coli che porta mutazioni nei geni dut e ung
Il gene DUT codifica per una UTPasi la cui funzione è di degradare il
dUTP nella cellula. La mutazione dut - provoca un elevata
concentrazione di dUTP (U) che si accumula nella cellula e tende a
sostituirsi al dTTP (T) durante la replicazione del DNA
Il gene UNG codifica per una uracil-N-glicosilasi che normalmente
rimuove l uracile dal DNA
Mutagenesi sito-specifica
con il metodo dut- ungDopo aver fatto crescere il costrutto in un
ceppo dut -, ung - si purifica il DNAss e si
procede alla mutagenesi in vitro
utilizzando il primer mutagenizzato. Si
otterrà un plasmide ds in cui il filamento
parentale sarà ricco in UTP mentre quello
mutato avrà un normale contenuto di TTP.
Trasformando in un ceppo di E.coli WT
per il gene UNG che codifica per una
uracil-N-glicosilasi i residui di uracile
contenuti nel filamento parentale saranno
degradati distruggendo selettivamente il
filamento stampo. In questo modo la
maggioranza dei trasformanti ottenuti
sarà di tipo mutante.
I plasmidi sono clonati inMutagenesi
un ceppo di E.coli
con attività dam metilasica così che i siti
sito-specifica
GATC siano metilati. I plasmidi sono isolati e denaturati. I primer mutagenizzati sono
per PCR
appaiati al DNA clonato. Viene effettuata
la PCR e come risultato si avranno molecole
mutagenizzate di neo sintesi non metilate e molecole stampo metilate. La miscela di
reazione viene digerita con MboI, enzima di restrizione metilazione-specifico che taglia
solo il filamento stampo parentale. Il DNA clonato mutato viene usato per trasformare
E.coli.
Mutagenesi random di un gene
e screening dei mutanti
Per creare nuove varianti della GFP
sono state introdotte mutazioni
mediante PCR. Per aumentare la
frequenza di mutazione, l MgCl 2 è
sostituito con MnCl2 e la concentrazione
di nucleotidi è sbilanciata così da avere
un nucleotide a concentrazione più
bassa in ogni reazione. I prodotti di PCR
sono poi clonati in un vettore
d espressione. I mutanti GFP sono
analizzati sotto luce UV così da
evidenziare le colonie che hanno
espressione aumentata o colori diversi.
Mutagenesi sito-specifica a cassetta
Si applica quando il sito da mutare è compreso tra due siti di restrizione
convenienti, ad una distanza massima di 80-100 bp
KpnI
EcoRI
HindIII
BamHI
Vogliamo, per esempio cambiare una A in una T in una zona compresa tra i siti
EcoRI e HindIII
GAATTCAACGTTGGAAATATGCCATTAAGCTT
CTTAAGTTGCAACCTTTATACGGTAATTCGAA
A questo scopo:
1) Rimuoviamo il frammento interno EcoRI/HindIII contenente la base da
cambiare
G
CTTAA
AGCTT
A
2) Sintetizziamo due oligonucleotidi (max 100 bp) complementari tra loro,
eccetto per le parti terminali e contenenti la base mutata
AATTCAACGTTGGAATTATGCCATTA
GTTGCAACCTTAATACGGTAATTCGA
3) Denaturando e rinaturando gli oligo, questi si appaieranno tra loro e
potranno essere ligati alla sequenza priva della cassetta EcoRI/HindIII
AATTCAACGTTGGAATTATGCCATTA
GTTGCAACCTTAATACGGTAATTCGA
G
CTTAA
AGCTT
A
Generando, così la mutazione sito-specifica desiderata
GAATTCAACGTTGGAATTATGCCATTAAGCTT
CTTAAGTTGCAACCTTAATACGGTAATTCGAA
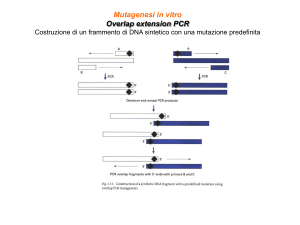
Mutagenesi
sito-specifica
per PCR
all interno di
un prodotto di
amplificazione
Mutagenesi Inserzionale
Un sistema alternativo di mutagenesi prevede l uso di DNA come agente
mutageno. Il genoma di piante e animali contiene elementi trasponibili,
sequenze di DNA che occasionalmente possono interrompere l integrità di un
gene causando una mutazione.
E importante sottolineare che la mutagenesi con elementi trasponibili puo
essere condotta a saturazione, cioe
inducendo un numero di trasposizioni
sufficientemente elevato da interrompere teoricamente, ciascun gene
dell organismo, almeno una volta in una delle linee mutagenizzate ottenute.
Oltre agli elementi trasponibili endogeni, anche i DNA esogeni possono
occasionalmente integrarsi in modo casuale interrompendo un gene.
Tra gli elementi trasponibili noti:
Elementi P di Drosophila
Tc1 in C.elegans
Ac-Ds in mais
Tam in Antirrhinium
Strategie genetico-molecolari
per studiare la funzione genica
Inattivazione di un gene
per identificare e caratterizzare un fenotipo
Non tutti i geni sono mutagenizzabili perché:
• geni funzionalmente ridondanti
(geni a funzione simile ma non omologhi)
(ridondanza strutturale v/s ridondanza funzionale)
• geni coinvolti in diverse fasi dello sviluppo, e mutazioni in
tali geni possono causare fenotipi letali o pleiotropici
mutagenesi sito-specifica
v/s
mutagenesi inserzionale random
Nelle piante ed in Drosophila No mutagenesi sito-specifica !!!!!
Mutagenesi Inserzionale random
Promoter Trapping
Utilizzando un gene reporter all interno del trasposone, è possibile
identificare promotori/enhancer mediante espressione del reporter.
Strategie di Mutagenesi Inserzionale
Nelle piante
a) T-DNA
• mutageno inserzionale che produce inserzioni stabili
• no hot spots di integrazione
b) Trasposoni
• il trasposone può essere exciso dal gene inattivato
(reversione del fenotipo = complementazione)
• eventi di trasposizione avvengono in zone limitrofe al sito di inserzione
Esempio di T-DNA ingegnerizzato
come elemento mutageno
RB
LB
GUS
nos3 ocs3
KanR
Pnos
35S
Erbic R
Gene tagging
Un vantaggio della mutagenesi inserzionale è rappresentato dal fatto che il
locus interrotto risulta anche marcato o tagged . L’uso di trasposoni non
endogeni o transgeni con caratteristiche equivalenti offre una sequenza tag
unica che permette l identificazione del gene inattivato senza ambiguità.
Tra le diverse strategie disponibili per isolare il gene taggato, o meglio le
flanking sequences del trasposone:
o Plasmic Rescue
o Inverse PCR
o Libreria genomica dell organismo taggato
Plasmid rescue
X
RB Kana
T-DNA
X
ori amp LB
Digestione
X
X
ori amp
T-DNA
Ligazione intramolecolare
T-DNA
X
X
X
Trasformazione E.coli
Selezione colonie AmpR, su LB+Amp
Inverse PCR
X
X
RB
LB
T-DNA
GUS
Kana
Digestione
X
X
T-DNA
Ligazione intramolecolare
T-DNA
X
PCR
X
Mutagenesi per
ricombinazione omologa
Qualsiasi regione del genoma di
lievito può essere sostituita da
sequenze di interesse, o un gene
endogeno può essere sostituito da una
sua copia mutagenizzata. Il DNA da
inserire viene fiancheggiato da
sequenze omologhe a quelle che
fiancheggiano la regione genomica da
sostituire. Le alte frequenze di
ricombinazione omologa di lievito,
garantiscono un alta percentuale di
ricombinazione con il cromosoma che
ha come risultato lo scambio genetico.
Il fenotipo mutante
dipende dall inserzione del T-DNA,
cioé dall inattivazione del gene X ???
DIMOSTRAZIONE FORMALE:
COMPLEMENTAZIONE!!!!!!!
Complementazione
Trasformazione dell organismo mutante con una copia del
gene WT, al fine di restaurare la funzione mancante