31/08/2009
Di
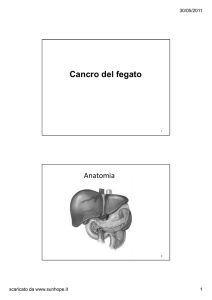
Diagnostica
ti virologica
i l i
Microscopia Elettronica
106 particelle virali per ml
I virus possono essere evidenziati in diversi campioni
Feci
Rotavirus, Adenovirus
Norwalk like viruses
Astrovirus, Calicivirus
Fluido da vescicole HSV
VZV
Cute (tamponi)
scaricato da www.sunhope.it
Papillomavirus,
Mollusco contagioso
1
31/08/2009
Problemi con il Microscopio
Elettronico
• Macchinari costosi
• Manutenzione costosa
• Necessità di personale specializzato
• A volte bassa sensibilità
MICROSCOPIO ELETTRONICO
Utilizza un fascio di elettroni al posto della luce. Poiché la lunghezza d'onda è
inversamente proporzionale alla velocità degli elettroni si può raggiungere un
limite di risoluzione di oltre 10000 volte rispetto al microscopio ottico e con un
ingrandimento di un milione x potendo vedere anche strutture di dimensioni pari
a 0,1 nm. L'immagine formata dagli elettroni non può essere vista direttamente,
viene quindi proiettata su uno schermo fluorescente o su una lastra fotografica.
Il campione, disperdendo o assorbendo gli elettroni che lo colpiscono,produce
un'immagine micrografica in funzione delle differenze di spessore, densità e
composizione chimica. Poiché anche le molecole di gas assorbono o riflettono
elettroni è necessario lavorare mantenendo la camera interna dello strumento
sotto vuoto.
scaricato da www.sunhope.it
2
31/08/2009
Microscopio elettronico a trasmissione (TEM).
Questo microscopio viene utilizzato per osservare
sezioni di cellule molto sottili per rilevare le strutture
interne. Il TEM viene anche utilizzato per studiare
i virus.
virus
Microscopio elettronico a scansione (SEM).
Questo microscopio serve per fornire immagini tridimensionali delle cellule.
scaricato da www.sunhope.it
3
31/08/2009
Electronmicrographs
Adenovirus
Rotavirus
Herpesvirus Particle
HSV-22 virus
HSV
i
particle.
ti l Note
N t that
th t all
ll
herpesviruses
have
identical
morphology and cannot be
distinguished from each other
under electron microscopy.
(Linda Stannard, University of Cape Town, S.A.)
scaricato da www.sunhope.it
4
31/08/2009
Adenovirus
• ds DNA virus
• non-enveloped
• At least 47 serotypes are
known
• classified into 6 subgenera:
A to F
(Linda Stannard, University of Cape Town, S.A.)
Influenza Virus
•
•
•
•
•
RNA virus, ggenome consists of 8
segments
enveloped
virus,
with
haemagglutinin and neuraminidase
spikes
3 types: A, B, and C
Type A undergoes antigenic shift
and drift.
Type B undergoes antigenic drift
only and type C is relatively stable
(Courtesy of Linda Stannard,
University of Cape Town, S.A.)
scaricato da www.sunhope.it
5
31/08/2009
Caso di encefalite virale.
Preparato di tessuto cerebrale
con presenza di particelle virali
del virus herpes simplex di tipo 1
(HSV-1) osservato al microscopio
Elettronico.
IMMUNOELETTROMICROSCOPIA
ELETTRONICA
Accoppia una reazione immunologia all’esame al microscopio
elettronico. Serve all’evidenziazione delle particelle virali
attraverso formazione di aggregati antigene-anticorpo o per
identificare sierologicamente i virus. Vengono utilizzati
anticorpi specifici, coniugati a oro colloidale (immunogold) in
quanto data l’elevata elettrondensità delle particelle di oro
colloidale
ll id l sii ha
h un contrasto
t t notevole
t l delle
d ll strutture
t tt
che
h hanno
h
reagito con tale coniugato, che diventano identificabili.
scaricato da www.sunhope.it
6
31/08/2009
Immunoelettronmicroscopia (IEM)
Tecnica dell’immunogold: anticorpi specifici anti-herpesvirus, coniugati a particelle d’oro colloidale, consentono
di identificare e di tipizzare le particelle virali presenti nel campione. Sono particolarmente visibili 3 virioni, con
envelope, ricoperti da granuli d’oro (punti neri, elettrondensi) che identificano l’avvenuto legame dell’anticorpo
con l’antigene specifico
scaricato da www.sunhope.it
7
31/08/2009
Isolamento di virus su animali
1) Provenienza
-SFP
2) Specie
-ospite naturale
-topi, cavie, criceti, ratti, gatti, conigli
3) Età
- neonati o animali molto giovani
g
4) Inoculazione
-intracerebrale -sottocutanea
-nasale
-intramuscolare
-intraperitoneale -endovenosa
-intradermica
Isolamento virale
a) Coltura in uova embrionate di pollo
b) Coltura su monostrati cellulari
scaricato da www.sunhope.it
8
31/08/2009
Coltura in uova
embrionate di pollo
Rappresentazione schematica dell’uovo embrionato di pollo, al decimo giorno di
incubazione.
scaricato da www.sunhope.it
9
31/08/2009
Lesioni (pocks) prodotte da poxvirus sulla membrana corion-allantoidea
di embrione di pollo
POCKS:
Vaccinia virus
Vaccinia virus
scaricato da www.sunhope.it
Pocks
HSV-1
Herpesvirus
10
31/08/2009
Isolamento Virale
Le colture cellulari sono il metodo maggiormente
utilizzato per isolare i virus e possono essere di tre
tipi:
1. Colture primarie - Monkey Kidney
2. Colture semi-continue - Human embryonic
kidney and skin
fibroblasts
3 C
3.
Colture
ltu continue
ntinu - HeLa,
H L V
Vero, H
Hep2,
p2 LLC-MK2,
LLC MK2
MDCK
Proceduta per l’allestimento di una
coltura
in vitro di cellule di rene.
scaricato da www.sunhope.it
11
31/08/2009
scaricato da www.sunhope.it
12
31/08/2009
Terreni di coltura
-Terreni liquidi
-Soluzione acquosa di: zuccheri
aminoacidi
sali minerali
vitamine
tampone (bicarbonato di sodio)
-CO2 (5%) (tappo aperto)
-Siero fetale (10%)
-Antibiotici: penicillina
streptomicina
BHK-21 (linea cellulare fibroblastica)
MCF-8 (linea cellulare epiteliale)
scaricato da www.sunhope.it
13
31/08/2009
scaricato da www.sunhope.it
14
31/08/2009
Modificazione di un monostrato cellulare in seguito ad infezione virale
T: h0
T: h10
T: h24
Effetto citopatico causato dalla crescita dei virus su colture cellulari
A) Herpes simplex virus in cellule di rene B) Citomegalovirus in fibroblasti umani; C) Virus respiratorio
sinciziale in cellule HEp-2; D) Cellule primarie di rene di coniglio non infette; E) Fibroblasti umani non infetti
F) Cellule HEp-2 non infette
scaricato da www.sunhope.it
15
31/08/2009
Effetto citopatico
Formazione di sincizi in colture
cellulari causate da RSV (top), and
virus del morbillo (bottom).
Sincizio indotto dalla replicazione di un retrovirus sinciziale in una coltura primaria
di fibroblasti di embrione di pollo
scaricato da www.sunhope.it
16
31/08/2009
Vacuolizzazione citoplasmatica indotta da un togavirus su cellule renali da feto bovino
scaricato da www.sunhope.it
17
31/08/2009
Corpi inclusi in cellule infettate da virus
a) poxvirus: inclusioni acidofile
intracitoplamatiche
(corpi del Guarnieri)
b)
herpesvirus: inclusioni acidofile
intranucleari
c)
reovirus: inclusioni acidofile
intracitoplasmatiche
perinucleari
d)
adenovirus: inclusioni basofile
intranucleari
e)
virus della rabbia: inclusioni acidofile
intracitoplasmatiche (corpi del Negri)
f)
morbillivirus: inclusioni acidofile
intracitoplasmatiche ed intranucleari
b ed f) fusione cellulare con produzione
di sincizi
Coltura cellulare di rene di scimmia infettata
con vaccinia virus: la freccia indica l’inclusione
eosinofilica sferoidale intracitoplamatica
(corpo del Guarnieri); la zona chiara che
circonda l’inclusione è particolarmente
caratteristica.
caratteristica
scaricato da www.sunhope.it
18
31/08/2009
Corpi inclusi intranucleari basofili da adenovirus
EMOADSORBIMENTO
Reazione di emoadsorbimento prodotta su cellule renali di scimmia, in coltura, infettate
con virus influenzale. I globuli rossi, aggiunti alla coltura, rimangono adesi alle
emagglutinine virali presenti sulla membrana della cellula infetta.
scaricato da www.sunhope.it
19
31/08/2009
ELVIS HSV, linea cellulare geneticamente ingegnerizzata per l’identificazione dell’Herpes simplex virus
Le cellule ELVIS sono cellule di rene di hamster che contengono il gene per la beta-galattosidasi sotto il controllo del
promoter UL39 dell’HSV. Se l’HSV è presente nel campione, le particelle virali penetrano nelle cellule, e le proteine
virali ICP0 e VP16 attivano il promoter UL39, portando alla sintesi della beta-galattosidasi. La presenza di betagalattosidasi viene rilevata mediante l’aggiunta di X-gal, un substrato per l’enzima. L’attività della beta-galattosidasi
sull’X-gal porta alla colorazione blu delle cellule ELVIS, visibile al microscopio.
Infezione del cervello di topo con un HSV incapace di replicarsi ingegnerizzato in modo da
E i
Esprimere
il gene batterico
b tt i per la
l beta-galattosidasi
b t
l tt id i come marcatore.
t
Un’aliquota di virus (alfa6) è stato iniettato nell’ippocampo di un ratto Sprague-Dawley.
Dopo 4 giorni, il ratto viene sacrificato, l’encefalo sezionato, fissato con paraformaldaide e
colorato con X-gal, che forma un precipitato blu in presenza dell’enzima.
In B: un ingrandimento dell’area indicata con la freccia nella figura A. Le cellule sono state
colorate con un colorante di contrasto. Il virus si trova concentrato nelle cellule piramidali e
nei loro processi dendritici.
scaricato da www.sunhope.it
20
31/08/2009
TITOLAZIONE
In molti casi è necessario conoscere oltre al tipo di virus
isolato anche la quantità di particelle virali. Le procedure
per la titolazione del virus si basano sul rilievo di alcune
caratteristiche fisiche, chimiche, biologiche di cui la più
importante è l’infettività cioè la capacità di un virus di
un virus di produrre effetti visibili nelle cellule ospiti.
Titolazione dei virus
Titolazione fisica
I virioni
i i i vengono contatii all microscopio
i
i elettronico
l
i mescolati
l i
con un numero noto di particelle di lattice
Titolazione per emagglutinazione
Questo metodo utilizza la proprietà di alcuni virus di agglutinare
i globuli rossi
Titolazione biologica
-Metodo delle placche che sfrutta il presupposto che una singola
particella virale sia in grado di iniziare un focolaio di infezione
-Metodo della diluizione limite – (Dose infettante il 50%)
scaricato da www.sunhope.it
21
31/08/2009
TITOLAZIONE FISICA
I virioni p
possono essere riconosciuti al ME e contati
mescolandoli ad un numero noto di particelle di
lattice ed eseguendo un conteggio proporzionale su
singole gocce di miscela. Però in questo modo non è
possibile differenziare le particelle infettive da quelle
che non lo sono. Questa tecnica è indaginosa e poco
usata.
TITOLAZIONE MEDIANTE
FENOMENO DI EMOAGGLUTINAZIONE
Un virione o una emoagglutinina isolata, si uniscono
contemporaneamente a due globuli rossi e funzionano da ponte
tra loro; con elevate concentrazioni virali si ottengono così degli
aggregati formati da un reticolo di eritrociti legati tra loro dalle
particelle virali. Utilizzando questo fenomeno è possibile titolare
i virus emoagglutinante, mediante il metodo della diluizione al
punto finale cioè: concentrazioni decrescenti di virus sono
mescolate ad una quantità standardizzata di globuli rossi e la più
alta diluizione di virus capace di provocare emoagglutinazione
evidente è detta unità emoagglutinante (UHA).
scaricato da www.sunhope.it
22
31/08/2009
Il riconoscimento dell’emoagglutinazione è basato
sull’esame
sull
esame dei globuli rossi dopo sedimentazione;
infatti mentre i globuli rossi normali si depositano sul
fondo del pozzetto raccogliendosi al centro e
formando un dischetto con margini ben definiti, i
globuli rossi agglutinati si dispongono in un sottile
strato, a margini irregolari, che ricopre tutto il fondo
del pozzetto.
Emoagglutinazione da virus
scaricato da www.sunhope.it
23
31/08/2009
scaricato da www.sunhope.it
24
31/08/2009
Placche da virus
METODO DELLE PLACCHE
Si basa sulla conta delle lesioni localizzate su un
monostrato di cellule sensibili o sulla membrana
corion allantoidea dell
corion-allantoidea
dell’uovo
uovo embrionato di pollo
prodotte da un virus.
Queste lesioni sono proporzionali al numero di unità
infettanti presenti nell’inoculum in quanto si assume
che una singola particella virale sia in grado di iniziare
l’infezione a carico di una cellula p
producendo lesioni,,
in successivi cicli di replicazione, alle cellule
adiacenti.
scaricato da www.sunhope.it
25
31/08/2009
Si utilizzano monostrati di cellule in piastre petri o in
piastre a pozzetti in cui viene allontanato il terreno e si
sostituiscono quantità costanti (0,1-0,2ml) di diverse
diluizioni (in base 10) della sospensione virale; si lascia
a contatto per 45 min (tempo di adsorbimento) per
consentire al virus di aderire alle cellule; si allontana poi
l’inoculum e si aggiunge sul monostrato terreno
semisolido (agar, carbossimetilcellulosa, composti
ggelatinosi).
) In q
questo modo,, il virus p
può diffondere dalla
cellula inizialmente infettata a quelle circostanti dando
luogo alle placche.
Metodo delle placche (secondo Dulbecco)
1))
2)
3)
4)
5)
Osservare le cellule al microscopio
p (piastra
(p
a 6 ppozzetti))
Lavare le cellule con PBS per 2 volte
Inoculare la diluizione virale in base 10 (da 0,2 a 0,5 ml)
Incubare a 37°C per 30 minuti
Aggiungere ¾ ml di terreno contenente metilcellulosa
(o altro agente gelatinoso)
6) Incubare a 37°C per il tempo necessario
7) Colorare e contare le placche
NUMERO PLACCHE X RECIPROCO DILUIZIONE X RECIPROCO VOLUME
Es.: 35 x 106 x 5 = 1,75 x 108 pfu/ml
scaricato da www.sunhope.it
26
31/08/2009
Risultato di una titolazione virale mediante il metodo delle placche
HERPESVIRUS PFUs: Diluted Virus
Mengo Virus Mutants: S, M, and L Plaques
scaricato da www.sunhope.it
27
31/08/2009
METODO DELLA DILUIZIONE LIMITE
E’ più rapido del metodo delle placche (entro 48 ore, mentre lo sviluppo di
placche richiede da 5g. a 3 sett.), e di più facile esecuzione.
Il virus è diluito serialmente (in base 10) e un volume costante è inoculato in un
numero stabilito di unità biologiche
g
((tessuto-colture,, embrioni di ppollo,, animali).
)
Si osserva poi per ogni diluizione, quante unità hanno risposto all’infezione, che
può provocare degenerazione delle tessuto-colture, comparsa di effetti citopatici,
morte degli embrioni, morte o malattia degli animali. Se il test è eseguito
correttamente, le concentrazioni più elevate del virus determinano infezione di
tutte le unità inoculate, mentre quelle meno elevate infettano un numero di unità
variabile finendo per non infettarne alcuna. Punto finale della titolazione è la
diluizione che infetta il 50% delle unità inoculate, e il titolo è espresso in DI50
(d
(dose
i f tt t all 50%).
infettante
50%)
scaricato da www.sunhope.it
28
31/08/2009
IDENTIFICAZIONE IN BASE A TECNICHE SIEROLOGICHE ED
IMMUNOLOGICHE
REAZIONI SIEROLOGICHE: sono utilizzate in clinica medica come supporto
diagnostico nei soggetti colpiti da malattie infettive,
infettive parassitarie,
parassitarie allergie,
allergie malattie
autoimmuni.
Il rilievo della presenza nel siero di sangue del paziente di elevate concentrazioni di
anticorpi che reagiscono con preparazioni allestite in laboratorio contenenti antigeni
o suoi prodotti derivanti dal microrganismo sospettato indica che il paziente è stato
esposto a quel patogeno e che ha reagito immunologicamente.
Si esaminano contemporaneamente due campioni di siero: il primo detto siero acuto
è prelevato appena inizia la sintomatologia clinica; il secondo detto siero
convalescente è prelevato dopo due o tre settimane.
.
Le reazioni sierologiche sono usate per identificare
microrganismi direttamente nei tessuti infetti o
dopo il loro isolamento in laboratorio. A tale scopo
d
devono
essere disponibili
di
ibili sieri
i i iperimmuni
i i
i o
anticorpi
monoclonali
che
reagiscono
specificamente solo con il patogeno che si ricerca.
Il tipo di reazione che si può osservare facendo
reagire un antigene con lo specifico anticorpo
dipende dallo stato fisico dell'antigene e dalle
condizioni sperimentali della prova che si vuole
eseguire
scaricato da www.sunhope.it
29
31/08/2009
Profilo sierologico tipico di un’infezione acuta
Durante una reinfezione le IgM possono essere assenti o con titolo molto basso
AGGLUTINAZIONE E PRECIPITAZIONE
E' una reazione antigene-anticorpo applicata a scopo sierodiagnostico o per
individuare la presenza di agglutinine (anticorpi agglutinanti) nel siero di pazienti.
E' importante sottolineare la sostanziale similitudine che sussiste tra reazione di
precipitazione (in cui si utilizzano antigeni solubili non legati a cellule) e
agglutinazione ( in cui sono necessarie cellule nella loro interezza per formare un
reticolo dato dall'unione dell'anticorpo a due cellule contigue).
L'agglutinazione permette di individuare anche antigeni solubili multivalenti
(antigeni capsulari) avvalendosi di matrici inerti al lattice ( di forma generalmente
sferica) coniugate con immunoglobuline specifiche note sensibilizzate (le
immunoglobuline sono legate sulla superficie delle sfere) dirette contro un certo
antigene. L'agglutinazione si visualizza con formazione di granuli sul fondo della
provetta o su particolari cartoncini a fondo scuro.
* A differenza dell'agglutinazione, la precipitazione è più rapida (15 min.) anche a
temperatura ambiente (20-25°C) ma risente in maggior misura del rapporto ottimale
antigene-anticorpo, inoltre non si manifesta quando i sieri campione sono diluiti
oltre 10-50 volte
scaricato da www.sunhope.it
30
31/08/2009
Grafico della formazione del precipitato. La
quantità di anticorpi è costante, mentre la
quantità di antigeni aumenta
progressivamente. Nel caso di eccesso di
antigeni o di anticorpi non si ha alcun
precipitato, mentre la precipitazione avviene
nella zona di equivalenza.
Esempio di una
reazione di
precipitazione
1
2
3
4
5
6
PRECIPITAZIONE IN GEL DI AGAR
U o entrambi
Uno
t
bi i reagenti
ti (antigene
( ti
ed
d anticorpo)
ti
)
diffondono in gel (agar, agarosio, amido) e danno
origine a precipitazione nel punto in cui si incontrano
a concentrazione ottimale. Si formano bande ben
visibili e separate per cui è possibile identificare i
singoli componenti antigenici presenti in una stessa
soluzione.
scaricato da www.sunhope.it
31
31/08/2009
Immunodiffusione doppia in gel di agarosio
Pozzetto centrale: Mix anticorpi contro HSV
Pozzetti laterali: Siero di pazienti
Risultati: la formazione di una linea
bianca
diprecipitato
indica
il
riconoscimento tra
antigene/anticorpo
(reazione positiva).
I pazienti 1, 2, 3, 4 e 5 risultano
infetti da HSV.
Il paziente 6 risulta negativo.
Immunodiffusione
radiale
HSV-1
Gli anticorpi vengono distribuiti uniformemente
nel gel di agarosio. In questo test, solo i campioni
contenente gli antigeni e caricati nei pozzetti,
possono diffondere. Nel momento in cui le
molecole antigeniche migrano nel gel, esse si
legano agli anticorpi presenti nel gel e nel
momento di equivalenza precipitano a formare
un anello. Il diametro del’anello permette di
risalire allaconcentrazione dell’antigene
Diametro di precipitazione
scaricato da www.sunhope.it
32
31/08/2009
REAZIONE DI DEVIAZIONE DEL
COMPLEMENTO
Il complemento, complesso di globuline presenti nel
siero, interviene aspecificamente nella risposta
immunitaria contro antigeni estranei legandosi al
complesso antigene-anticorpo e causando la lisi
dell'antigene.
Essendo un sistema cronolabile e termolabile, nella
utilizzazione diagnostica deve essere sempre
utilizzato
tili
t siero
i
f
fresco
(
(generalmente
l
t di cavia).
i )
Le tecniche diagnostiche che utilizzano il
complemento si avvalgono di due fasi:
Fissazione del complemento
1) Prima fase: siero del paziente da
esaminare (contenente anticorpi o antigeni
solubili) a cui si aggiunge antigene solubile
noto (per rivelare anticorpi) o anticorpo
noto
(per
rivelare
antigeni).
Dopo
incubazione si aggiunge siero fresco di cavia
in quantità nota e standardizzata.
standardizzata Il
complemento si lega al frammento Fc
dell'anticorpo soltanto se si forma
l'immunocomplesso.
scaricato da www.sunhope.it
33
31/08/2009
Fissazione del complemento
2) Seconda fase: ha funzione rivelatrice ed è
costituita dal complesso emazie ed anticorpi antiemazie
(emolisine). Se precedentemente non si è formato
l'immunocomplesso, il complemento è libero e si legherà
al complesso emazie-emolisine provocando la lisi delle
emazie (reazione negativa, assenza di antigene o
anticorpo nel siero del paziente), se invece si è formato
l'immunocomplesso,
p
il complemento
p
è legato
g
ad esso e
non sarà disponibile per il secondo sistema quindi non si
avrà lisi delle emazie (reazione positiva presenza di
antigene o anticorpi nel siero del paziente).
Fissazione del complemento
scaricato da www.sunhope.it
34
31/08/2009
Fissazione del complemento
Complement Fixation Test in Microtiter Plate. Rows 1 and 2 exhibit complement
fixation obtained with acute and convalescent phase serum specimens,
respectively. (2-fold serum dilutions were used) The observed 4-fold increase is
significant and indicates recent infection.
ELISA ( ENZYME LINKED IMMUNOASSORBENT
ASSAY)
Le tecniche immunoenzimatiche derivano da quelle radioimmunologiche,
ma utilizzano un enzima, al p
posto dell'isotopo
p radioattivo, come marker
dell'anticorpo specifico o dell'anti-gammaglobulina.
Permette di rivelare la presenza del microrganismo (antigene) o di
anticorpi, utilizzando anticorpi coniugati con enzima (fosfatasi alcalina o
perossidasi).
L'aggiunta di un substrato cromogeno per l'enzima mette in evidenza
l'eventuale presenza di antigene o anticorpo in base all'emissione di
colore dovuto all'azione dell'enzima, legato all'anticorpo, sul substrato;
la colorazione sarà tanto più marcata quanto maggiore è la quantità di
anticorpo coniugato con enzima che si lega al substrato.
Nella tecnica ELISA,generalmente gli antigeni sono adsorbiti alle pareti
di provette di plastica o pozzetti di piastre microtitre
scaricato da www.sunhope.it
35
31/08/2009
Enzyme-linked immunosorbent assay (ELISA)
a) Diretta
b) Indiretta
Enzyme-linked immunosorbent assay
Schema di base
Immunoassay competitivo
scaricato da www.sunhope.it
36
31/08/2009
ELISA for HIV antibody
Microplate ELISA for HIV antibody: coloured wells indicate reactivity
SIERONEUTRALIZZAZIONE VIRALE
Quando un virus è posto a contatto con un siero di un
soggetto convalescente
l
o immunizzato,
i
i
perde
d del
d l tutto
o in parte il potere infettante. Ciò è dovuto,
probabilmente, al fatto che gli anticorpi specifici
rivestono il virione impedendo l’adsorbimento di
questo alla cellula ospite e quindi il passaggio
dell’acido
dell
acido nucleico virale nella cellula.
cellula
scaricato da www.sunhope.it
37
31/08/2009
Il tasso di anticorpi neutralizzanti nel siero può essere
valutato allestendo diluizioni seriali, per raddoppio,
del siero mescolate a quantità standard di virus
infettante (100 DITC50) a T adatta per un certo tempo
(1-2 ore a T ambiente).
Le diverse miscele virus-siero sono poi saggiate per
rilevarne l’infettività residua mediante titolazione.
Titolo finale degli anticorpi neutralizzanti è
considerata la più alta diluizione del siero ancora
capace di prevenire i fenomeni legati alle proprietà
infettanti del virus.
scaricato da www.sunhope.it
38
31/08/2009
Immunofluorescenza
E' una metodica che consente
di identificare i microrganismi
presenti nel materiale biologico
opp re di individuare
oppure
indi id are antigeni
a localizzazione intracellulare o
anticorpi. La metodica si basa
sulla marcatura fluorescente
degli
anticorpi
rivelatori
osservati poi al microscopio a
fluorescenza.
Diretta
Indiretta
Immunofluorescenza
Cellule trasfettate con DNA plasmidico
Contenente il gene della glicoproteina gL
dell’HCMV. La proteina codificata dal gene
viene espressa in seguito alla sua trascrizione
in mRNA. Le cellule vengono permeabilizzate
ed incubate con un anticorpo che riconosce
la glicoproteina gL.
Tempo 0; Tempo 12h
Tempo 24h
scaricato da www.sunhope.it
39
31/08/2009
Visualizzazione al microscopio confocale
di due glicoproteine dell’HCMV.
(IE72 in rosso e pp65 in verde)
Cellule aortiche endoteliali primarie sono
state infettate con HCMV isolato da paziente
ed osservate al microscopio confocale dopo
8 giorni.
Associazione della glicoproteina E del VZV
((verde)
d ) con la
l membrana
b
di una cellula.
ll l
Espressione del recettore per la transferrina
nella stessa cellula (rosso).
Localizzazione di entrambe le proteine e
quindi della fluorescenza sugli stessi siti
cellulari (giallo).
Immunofluorescenza
Immunofluorescenza diretta su cellule infettate da rotavirus: l’anticorpo fluoresceinato è legato
specificamente ad antigeni virali presenti nel citoplasma.
scaricato da www.sunhope.it
40
31/08/2009
Metodi molecolari
• M
Metodi
t di basati
b
ti sulla
ll individuazione
i di id
i
d l genoma
del
virale.
• Rappresentano il futuro della diagnostica virologica.
Comunque, nonostante il continuo incremento dei
metodi molecolari, nella pratica clinica della
diagnostica di laboratorio il loro utilizzo è ancora
nettamente
inferiore
rispetto
ai
metodi
convenzionali.
convenzionali
Ibridazione molecolare
Quando un DNA a doppio filamento viene riscaldato a
temperatura superiore alla cosiddetta temperatura di fusione
(Tm) le due catene della molecola si separano in seguito alla
rottura dei legami ad idrogeno (deboli) che le mantengono
appaiate.
Se dopo la separazione, il DNA viene raffreddato lentamente in
condizioni ambientali favorevoli si ha una riassociazione
progressiva delle due eliche detta ibridazione.
La riassociazione avviene solo tra due sequenze strettamente
complementari che possono essere sia di RNA che di DNA (
DNA/DNA, DNA/RNA).
scaricato da www.sunhope.it
41
31/08/2009
Ibridazione molecolare
Sistemi per marcare sonde molecolari
a) Marcatura radioisotopica
b) Marcatura enzimatica
c) Marcatura mediante biotina-streptavidina
scaricato da www.sunhope.it
42
31/08/2009
Rilievo del genoma virale mediante l’uso di sonde marcate radioattivamente
DOT BLOT
Ibridizazzione con una sonda a DNA dal genoma di papillomavirus su quantità scalari (a, b, c, d, e) di DNA
estratto da un campioni patologici di tre pazienti.
Una serie di diluizioni di mRNA
campione, il cui titolo è noto,
sono allineate su un foglio di
nitrocellulosa o nylon. Su una
linea parallela sono depositate le
diluizioni della soluzione di RNA
da dosare. Gli RNA sono
immobilizzati ed ibridati con la
sonda radioattiva. Dopo
lavaggio viene praticata un
autoradiografia. L'intensità
p
delle macchie corrispondenti
alle diluizioni della soluzione in
cui si trova l'RNA da dosare
viene confrontata con quella dei
controlli.
Risultati: Il segnale radioattivo è proporzionale alla quantità di virus presente nel campione
I pazienti 1 e 2 sono positivi.
Il paziente 3 risulta negativo
Southern blotting:
tecnica di analisi del DNA messa a ppunto da
Southern nel 1975 per visualizzare i geni o
qualsiasi sequenza di DNA genomico, mediante
ibridazione con una sonda marcata e specifica,
con frammenti di restrizione del DNA separati
precedentemente con ll'elettroforesi
elettroforesi, denaturati e
trasferiti su membrana.
scaricato da www.sunhope.it
43
31/08/2009
Southern Blot
Il supporto viene poi pretrattato con la
soluzione salina priva di sonda
(preibridazione) in modo da saturare il
supporto e impedire il legame non
specifico della sonda marcata prima
dell'ibridazione. Si immerge poi per 1030 ore il filtro nella soluzione salina
contenente
la
sonda
radioattiva
(ibridazione ) a 65°C, si fanno infine una
serie di lavaggi per allontanare l'eccesso
di sonda che potrebbe dare falsi segnali
nell'immagine
autoradiografica.
La
sonda si andrà a legare solo a quella
ristretta regione del filtro (banda) dove è
fissato il frammento contenente sequenze
complementari Tale banda può essere
complementari.
messa
in
risalto
come
segnale
autoradiografico su una pellicola da
radiologia messa a contatto col filtro
oppure una reazione colorimetrica dopo
l'utilizzo del sistema avidina-cromogeno
per le sonde biotilinate.
Southern blot
Analisi di diversi ceppi di citomegalovirus
Soggetti positivi al CMV possono reinfettarsi
con un nuovo ceppo dopo trapianto d’organo
X1 ed Y1 = Prima del trapianto
X2 ed Y2 = Dopo il trapianto
scaricato da www.sunhope.it
44
31/08/2009
Polymerase Chain Reaction
• La PCR permette l’amplificazione in vitro (circa 106)di una
specifica sequenza bersaglio di DNA, rappresenta una
tecnica molto
l sensibile.
bl
• E’ basata su una reazione enzimatica che utilizza degli
oligonucleotidi sintetici che si ibridizzano alle estremità
della sequenza di acidi nucleici di interesse.
• Questi oligonucleotidi agiscono come primers per la Taq
polymerase termostabile. Cicli ripetuti (usualmente da 25 a
40) di denaturazione del DNA template (a 94oC), annealing
dei primers alla loro sequenza complementare (50oC),
C) e
l’estensione (72oC) risultano in una produzione esponenziale
dello specifico frammento bersaglio.
• Rilevamento ed identificazione del prodotto della PCR viene
effettuata mediante elettroforesi in gel di agarosio.
Polymerase Chain Reaction
• Vantaggi della PCR:
– Sensibilità estremamente elevata,
elevata può rilevare fino a una singola
copia di genoma virale.
– Facile
– Veloce
• Svantaggi della PCR:
– Facile da contaminare
– Necessità di operatori specializzati
– Difficolta di quantizzazione.
– Un risultato positivo può essere difficile da interpretare,
specialmente con I virus latenti (CMV), dove ciascun individuo
sieropositivo può avere nel sangue la presenza del virus,
presentando o meno sintomatologia specifica.
scaricato da www.sunhope.it
45
31/08/2009
Schematic of PCR
Each cycle doubles the copy number of the target
Polymerase Chain Reaction
scaricato da www.sunhope.it
46
31/08/2009
PCR (Primers)
La scelta dei primers è una tappa importante nella messa a
punto della PCR:
1) la sequenza dei primers non deve permettere la formazione
di stem-loop, né l'ibridazione tra primers;
2) la composizione in basi deve essere equilibrata
3) le Tm di due primers non devono essere troppo diverse
4) le sequenze scelte non devono corrispondere a sequenze
genomiche ripetute.
ripetute
5) la concentrazione degli ioni magnesio (necessari per
stabilizzare i nucleotidi) è compresa tra 0,5 e 2,5 mM
scaricato da www.sunhope.it
47
31/08/2009
NESTED PCR
Consiste nel realizzare due PCR successive utilizzando
coppie
i di primers
i
diff
differenti,
ti con la
l seconda
d coppia
i di
primers che contiene una sequenza inclusa in quella che
viene amplificata dalla prima coppia di primers.
Così, se la banda corrispondente alla prima amplificazione
è artefatta, durante la seconda PCR i primers della seconda
coppia non potranno ibridarsi e non avrà luogo
l amplificazione.
l'amplificazione
Questa tecnica permette di aumentare nello stesso tempo la
specificità e il tasso di amplificazione. E' particolarmente
adatta quando la quantità di bersaglio di partenza è bassa.
PCR e Multiplex PCR
scaricato da www.sunhope.it
48
31/08/2009
Tipizzazione di HSV mediante digestione con endonucleasi
di restrizione del prodotto di PCR
REAL TIME PCR (PCR Quantitativa)
L’analisi PCR quantitativa prevede, come la qualitativa, una prima fase
di estrazione del DNA dal campione in analisi. Il DNA, quindi,
sottoposto al processo di amplificazione con metodo Real Time PCR.
Tale tecnica usa una sonda complementare al bersaglio che
inizialmente non emette fluorescenza. Durante l’amplificazione del
DNA, l’enzima Taq polimerasi spezza questa sonda e la rende
fluorescente. Così è possibile rilevare in tempo reale l’andamento
dell’amplificazione del DNA, in quanto al progredire della stessa il
segnale fluorescente aumenta. La quantificazione del DNA è allora
possibile visto che esiste una diretta correlazione tra il contenuto di
DNA bersaglio presente inizialmente nel campione e tempo di inizio
emissione fluorescenza. In altre parole, prima parte l’emissione di
fluorescenza durante la PCR, tanto maggiore è il contenuto di DNA
scaricato da www.sunhope.it
49
31/08/2009
PCR quantitativa-competitiva
Un numere crescente di copie target di 144 bp
vengono distribuite in una serie di tubi, 250 copie
del competitore di 96 bp vengono aggiunte a
ciascun tubo. L’intensità delle bande viene
misurata mediante un sistema di video, e riportata
nel grafico.
AMPLICOR HIV-1 (ROCHE)
Nella reazione di PCR entrambi i primers sono biotinilati, per cui il prodotto della PCR viene trasferito
in piastra da microtitolazione sui cui pozzetti si trova una sonda con una sequenza specifica complementare
ad uno dei filamenti amplificati. Il prodotto di amplificazione viene denaturato ed il filamento complementare
viene catturato dalla sonda. Dopo lavaggio, viene aggiunto il substrato (perossidasi) e il cambiamento di colore
viene letto allo spettrofotometro.
scaricato da www.sunhope.it
50
31/08/2009
Quantiplex Branched-chain DNA (Bayer)
Il virus dal paziente viene precipitato mediante centrifugazione e trattato per rilasciare l’RNA. L’RNA target
viene ibridizzato a numerose sonde presenti nel pozzetto di una piastra per microtitolazione.
L’RNA immobilizzato viene in seguito ibridizzato a sequenze ramificate di DNA che vengono a loro volta
ibridizzate a sonde coniugate con l’enzima di rivelazione. Infine viene aggiunto un substrato luminescente e la
reazione viene letta mediante un luminometro. Questa tecnica non amplifica il target, ma il segnale.
NASBA
Permette di di amplificare
p
con la stessa efficacia sia ggli RNA
che i DNA. L'amplificazione è ottenuta senza cambiare la
temperatura, che viene mantenuta costantemente a 42°C.
Qualsiasi sia il tipo di acido nucleico di partenza, il prodotto di
amplificazione è insieme DNA e RNA.
Come nella tecnica della PCR sono necessari due
oligonucleotidi che inquadrano la sequenza da amplificare. Uno
dei due deve possedere comunque, nella porzione 5', una
sequenza che corrisponde ad un promotore per la T7 RNA
polimerasi (estratta da E. coli infettata dal fago T7).
scaricato da www.sunhope.it
51
31/08/2009
Nuclisens Nasba (Organon Teknika)
La prima tappa corrisponde ad una fase non ciclica che
produce un DNA a doppio filamento, di cui una estremità
possiede un promotore per la T7 RNA polimerasi.
La seconda tappa, che è ciclica, corrisponde alla fase di
amplificazione Essa si svolge in tre parti: nel corso della
amplificazione.
prima, la T7 polimerasi trascrive una serie di di RNA a
partire dal promotore T7; nella seconda, gli RNA che
sono stati trascritti vengono retro-trascritti in cDNA
dalla trascrittasi inversa; nell'ultima tappa del ciclo il
filamento di RNA è distrutto dalla Rnasi H, ed il secondo
filamento del cDNA viene sintetizzato immediatamente
dalla trascrittasi inversa che utilizza il secondo
oligonucleotide come primer.
Nel corso di questo ciclo vengono così ottenute più copie
del DNA di partenza. Esse servono da stampo al ciclo
seguente. Il tasso di amplificazione in ogni ciclo è molto
superiore a quello della PCR nella quale la quantità di
DNA viene semplicemente raddoppiata. Dopo 3 ore di
incubazione il tasso di amplificazione è da 106 a 107.
Nelle condizioni ottimali la quantità di RNA ottenuta è
100 volte superiore alla quantità di DNA.
scaricato da www.sunhope.it
52