La “chimica della vita” si basa sui composti del carbonio e
dipende da reazioni chimiche che avvengono in soluzione acquosa.
Le cellule contengono 4 famiglie principali di piccole molecole
organiche:
Amminoacidi
Subunità delle proteine
Nucleotidi
Subunità del DNA e del RNA
Zuccheri
Fonti di energia per la cellula
e subunità dei polisaccaridi
Acidi grassi
Fonti di energia (triacilgliceroli)
Componenti delle membrane cellulari
(fosfolipidi).
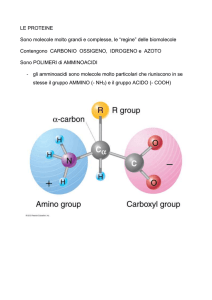
Le Proteine
La classe più versatile delle macromolecole biologiche
Alcune funzioni delle proteine:
Struttura generale di un amminoacido
Lisina
Stereoisomeria degli amminoacidi
I due isomeri L- e D- alanina sono immagini speculari non
sovrapponibili l’uno dell’altro
Per la classificazione e la
nomenclatura degli stereoisomeri il
composto di riferimento è lo zucchero
a 3 atomi di carbonio Gliceraldeide.
Con questo tipo di rappresentazione il gruppo R
dell’amminoacido va posto sotto il carbonio α. Gli Lamminoacidi hanno il gruppo amminico sulla sinistra, i Damminoacidi sulla destra
Gruppi R alifatici, non polari
Glicina
Alanina
Leucina
Metionina
Valina
Isoleucina
Gruppi R aromatici
Fenilalanina
Tirosina
Triptofano
Il triptofano e la tirosina, e in
misura minore la fenilalanina,
assorbono la luce ultravioletta.
Questo spiega perché la
maggior parte delle proteine
possiedono un caratteristico
assorbimento della luce a una
lunghezza d’onda di 280 nm.
Questa proprietà delle proteine
è utilizzata dai ricercatori per
idividuarle e quantificarle
Assorbimento della luce da parte delle molecole:
Un gran numero di biomolecole assorbono la luce a una
catteristica lunghezza d’onda.
La misura dell’assorbimento della luce con uno spettrofotometro è
utilizzata per identificare le molecole e per valutare la loro
concentrazione in soluzione.
La frazione della luce incidente che viene assorbita da una
soluzione a una data lunghezza d’onda è proporzionale allo
spessore della soluzione (cammino ottico) e alla concentrazione
della specie chimica che assorbe la luce.
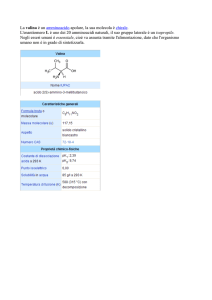
La legge di Lambert-Beer.
Io
log = εcl
I
Io = Intensità della luce incidente
I = Intensità della luce trasmessa
ε = coefficiente di estinsione molare (in unità di litro per moli-centimetro)
c = concentrazione della specie che assorbe la luce (in moli per litro)
l = lunghezza del cammino ottico (spessore del campione che assorbe la luce;
in centimetri)
L’espressione log (Io/I) viene detta assorbanza e indicata con A.
L’assorbanza A è direttamente proporzionale alla concentrazione
del soluto che sta assorbendo la luce.
I principali componenti di uno spettrofotometro
Io,
Intensità
della luce
incidente
Lampada:
Luce emessa
con una vasta
gamma di
lunghezze
d’onda
Monocromatore:
Sceglie e trasmette
una luce ad una
particolare
lunghezza d’onda
I,
Intensità
della luce
trasmessa
Cuvetta
Contenente c
moli/litro della
specie molecolare
che assorbe la luce
Registratore
Gruppi R polari, non carichi
Serina
Prolina
Treonina
Cisteina
Asparagina Glutammina
Prolina
Un amminoacido particolare
Struttura ciclica della Prolina
La catena laterale ( R ) è unita sia al carbonio α che al gruppo
amminico.
Cisteina
Due residui di cisteina possono essere uniti da un legame disolfuro
Gruppi R carichi positivamente
Lisina
Arginina
Istidina
Guanidino
(Arginina)
Imidazolo
(Istidina)
La ionizzazione dell’Istidina.
L’istidina può legare o rilasciare un protone a pH vicini
al pH fisiologico
Gruppi R carichi negativamente
Aspartato
Glutammato
Amminoacidi non standard
γ-Carbossiglutammato
4-Idrossiprolina
5-Idrossilisina
6-N-Metillisina
Desmosina
Selenocisteina
Le proteine possono contenere amminoacidi non standard che
derivano dalla modificazione chimica di uno dei 20 amminoacidi
standard dopo il suo inserimento nella proteina
La selenocisteina è un amminoacido raro che viene introdotto
nelle proteine durante la sintesi
L’ornitina e la citrullina non si trovano nelle proteine ma sono
intermedi della biosintesi dell’arginina e del ciclo dell’urea
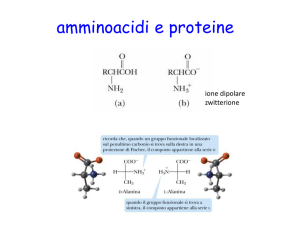
Struttura di un amminoacido
Forma non ionica
Forma ionica dipolare
(zwitterionica)
Gli stati di ionizzazione dipendono dal pH
Acido diprotico
NH3+
H - C - COOH
NH3+
- H+
H - C - COO-
NH2
H - C - COO-
+ H+
R
Forma predominante
a pH 1
R
Forma predominante
a pH 7
R
Forma predominante
a pH 11
La forma zwitterionica è quella predominante a valori di pH
vicini a quello fisiologico.
Gli amminoacidi hanno curve di titolazione caratteristiche
Titolazione di un amminoacido:
la glicina
pI = punto isoelettrico o pH
isoelettrico
Il valore di pH a cui la carica netta di un amminoacido è uguale a 0
viene detto punto isoelettrico o pH isoelettrico (pI).
Per un amminoacido come la glicina, che non ha gruppi ionizzabili
nella catena laterale, il punto isoelettrico è la media aritmetica dei
valori dei due pKa:
1
1
pI = (pK1 + pK 2 ) = (2,34 + 9,6) = 5,97
2
2
Un amminoacido ha una carica netta positiva a ogni valore di pH
inferiore rispetto al pI; se posto in un campo elettrico migra verso
l’elettrodo negativo (catodo),
ad ogni valore di pH superiore al pI ha carica netta negativa e
quindi migra verso l’elettrodo positivo (anodo)
Il valore di pKa di qualsiasi gruppo funzionale è influenzato dall’ambiente
chimico in cui si trova.
Tutti gli amminoacidi con un solo gruppo amminico α, un solo
gruppo carbossilico α e un gruppo R non ionizzabile hanno curve
di titolazione simili a quella della glicina.
Gli amminoacidi con un gruppo R ionizzabile hanno curve di
titolazione più complesse con tre fasi corrispondenti alle tre
possibili tappe di ionizzazione; in questo caso avremo tre valori di
pKa
Curva di titolazione del
Glutammato
(un amminoacido con R
contenente un gruppo acido).
Il pI = 3,22
(la media dei valori di pKa dei
due gruppi -COOH)
A questo valore di pH i due
gruppi -COOH contribuiscono
con una carica netta di -1 che
bilancia la carica netta +1 del
gruppo -NH3+
Curva di titolazione dell’
Istidina
(un amminoacido con R
contenente un gruppobasico).
Il pI = 7,59
(la media dei valori di pKa dei
gruppi amminico e imidazolico)
A questo valore di pH i due
gruppi basici contribuiscono
con una carica netta di +1 che
bilancia la carica netta -1 del
gruppo -COO-
Tipici valori di pKa dei gruppi ionizzabili presenti nelle proteine
L’istidina ha un gruppo R con pKa di 6,0 che può comportarsi da tampone a un pH vicino alla
neutralita
Reazione di
condensazione
Legame peptidico
Una catena polipeptidica è direzionale.
E’ possibile identificare un residuo ammino-terminale ed
un residuo carbossi-terminale
Estremità
ammino-terminale
Estremità
carbossi-terminale
Un tetrapeptide.
In rosa sono indicati i
gruppi ionizzabili
Come gli amminoacidi liberi, anche i
peptidi hanno curve di titolazione
caratteristiche e un pH isoelettrico (pI)
specifico, al cui valore non si muovono
in un campo elettrico.
I peptidi e i polipeptidi biologicamente attivi hanno dimensioni
molto variabili
(aspartame)
Dati molecolari di alcune proteine
E’ possibile calcolare in modo approssimativo il numero di residui
amminoacidici in una proteina semplice che non contiene altri gruppi
chimici dividendo la sua massa molecolare per 110.
La massa molecolare media dei 20 amminoacidi è 138, ma quelli più
piccoli sono più rappresentati nelle proteine.
Se si tien conto della proporzione con cui i vari amminoacidi entrano a
far parte delle proteine, la massa molecolare media si avvicina a 128
Per generare un legame peptidico viene eliminata una molecola di
acqua (Mw=18); la massa molecolare media di un residuo
amminoacidico in una proteina è 128 -18 = 110
Proteine che contengono solo amminoacidi = proteine semplici
Alcune proteine contengono gruppi chimici diversi dagli
amminoacidi= proteine coniugate
La parte non amminoacidica di una proteina coniugata è detto
gruppo prostetico
Il gruppo prostetico ha un ruolo determinante nella funzione delle proteine