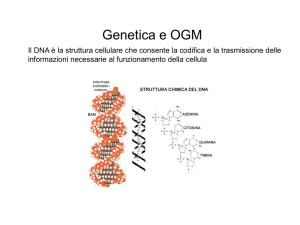
Metodi di analisi di OGM
Roberta Onori
Istituto Superiore di Sanità
Centro Nazionale per la Qualità degli Alimenti e per
i Rischi Alimentari
Quadro
no rmativo
Indag ini
s ulla
e s po s izio ne
Attività
Pre-marketing
me to di di analis i
Attività
Controllo
ufficiale
Postmarketing
Autocontrollo
Metodi di analisi degli OGM
¾ Ditte produttrici
¾ Per verificare le caratteristiche molecolari e la
segregazione dell’OGM
¾ Per monitorare la modificazione genetica mella
produzione di ibridi
¾ Idustrie alimentari, mangimistiche e sementiere
¾ Per garantire la tracciabilità dei prodotti
¾ Per garantire il rispetto delle normative vigenti
¾ Autorità Competenti
¾ Per l’attuazione del controllo ufficiale da parte dei
laboratori competenti
¾ Laboratori privati
¾ Per fornire servizio analitico ai richiedenti
Elementi che permettono di
individuare la presenza di OGM
Presenza del
“transgene”
Presenza della
proteina espressione
del transgene
inserito
Bt toxin crystals
METODI ANALITICI
per la DIAGNOSTICA degli OGM
DNA
PROTEINE
PCR
Tecniche
IMMUNOENZIMATICHE
(ELISA)
Metodi ELISA per la determinazione delle
proteine
Signal
development
proteine
Recognition
Sites
(Epitopes)
Enzima coniugato
Anticorpi
Anticorpo di cattura
Applicazione di metodi immunologici agli
OGM
I
Metodi rapidi qualitativi
I
Metodi quantitativi
Soia RR e colza
Mais Event 176
Mais Bt 11
Metodi rapidi
Reservoir Pad
Result Window
Labelled Filter
Cover
Reagent Flow
Lateral Flow Device Schematic
Reagent Flow
Reagent Flow
Single band
Negative Result
Two bands
Positive Result
Metodi quantitativi ELISA
Step
1.
Campione
Step 2.
Aliquota da analizzare
Step 5.
Data analysis
Step 4.
Estrazione delle
proteine
Step 3.
Dispensazione dell’estratto
nei pozzetti della piastra
Kit commerciali per l’analisi di OGM
BT Cry 1A(b)
Metodi ELISA
SVANTAGGI
VANTAGGI
• Materie prime o prodotti non
sottoposti a procedimenti tecnologici
in grado di denaturare le proteine
•Bassi costi rispetto PCR
• Impossibilità di discriminare OGM
diversi che esprimono la stessa
proteina
•Velocità del saggio
• Non esiste perfetta correlazione fra
proteina espressa e % di materiale
OGM
•Semplicità delle
operazioni (effettuabili anche
su campo TEST STRIP)
Metodi basati sul DNA
WT
mRNA
mRNA
Proteina - enzima
Proteina - enzima
metaboliti
OGM
P
E
Gene
T
P
E
Gene
T
metaboliti
mRNA
mRNA
mRNA
Proteina - enzima
Proteina - enzima
Proteina - enzima
metaboliti
metaboliti
metaboliti
Metodi basate sul DNA
Fasi analitiche di un metodo diagnostico basato sulla
PCR
Preparazione del campione
Estrazione del DNA
Valutazione quali-quantitativa del DNA
Preparazione dei reattivi della PCR
Amplificato
PCR
Preparazione Campione
“Laboratory Sample”
Materie prime
10.000 semi (CEN)
2500
2500
Omogeneizzazione/Macinazione
Aliquota 1 Aliquota 2
Aliquota 3
O
m
o
g
e
n
e
i
t
à
Aliquota 4
200-300 mg
Conservare a –20 qC
R
a
p
p
r
e
s
e
n
t
a
t
i
v
i
t
à
Alimenti processati
2 pacchetti
1 merendina
1 merendina
Omogeneizzazione/Macinazione
Aliquota 1
Aliquota 2
Aliquota 3
1-2 g
“Test Portion”
Estrazione
Estrazione DNA
DNA
Conservare a –20 qC
Aliquota 4
Estrazione del DNA
(ambiente dedicato)
Strumentazione
Bagno termostatato
Vortex mixer
Centrifuga
Reagenti e materiali monouso
Provette tipo eppendorf da 2 e 1,5 ml
Rack
Reattivi per l’estrazione (prodotti per
biologia molecolare)
Kit di purificazione
Acqua sterile
Puntali sterili con filtro
Pipette automatiche dedicate
La PCR Reazione a Catena della Polimerasi
DNA
Primers
dNTPs
PCR tampone + MgCl2
Taq DNA polymerase
Steps in a PCR Cycle
Denaturation (90-95 º C)
Primer Annealing (40-70 º C)
Extension (72 º C)
La PCR Reazione a Catena della Polimerasi
Curve di amplificazione
Numero di
molecole di
DNA
0
5
10
15
20
25
30
Numero di cicli
35
40
45
Metodi PCR per l’analisi di OGM
DNA della pianta
gene A
gene B
DNA della pianta
evento specifico
5
PCR specifica
pianta
4
PCR specifica
construtto
costrutto
gene
3
PCR specifica
del gene
2
PCR di
screening
OGM
O non OGM
1
PCR specifica
della specie
DNA
amplificabile
Al DNA si aggiunge una soluzione
colorata contenente un addensante che ne
facilita il caricamento su un gel di agarosio.
Analisi dell’amplificato
MWM
1a 1b 1c 2a 2b 2c 3a 3b 3c 4a 4b 4c 5a 5b 5c 6a 6b 6c MWM
Zeina
(specifico
del mais)
Lectina
(specifico
della soia)
MWM = Marker
Triplicati
1 = Mais 0% OGM
4 = Mais MON810 100% OGM
2 = Mais Bt-11 100% OGM
5 = Soia 0% OGM
3 = Mais Bt-176 100% OGM
6 = SoiaRR 100% GMO
Analisi dell’amplificato
MWM
1
2
3
4
5
6
7
C
MWM
437 bp (Bt-11)
194 bp (MON810)
120 bp (Bt-176)
MWM = Marker (Low Ladder 100 bp)
1 = MON810 0.5% OGM
2 = Bt-176 0.5% OGM
3 = Bt-11 0.5% OGM
4 = MON810 + Bt-11 0.25% OGM
C=
5=
6=
7=
No Template Control
MON810 + Bt-176 0.25% OGM
Bt-176 + Bt-11 0.25% OGM each
MON810 + Bt-11 + Bt-176 0.16% OGM each
GMO ?
Yes/No
*02GHWHFWLRQ
Positive
Negative
0.5% tolerance
*02LGHQWLILFDWLRQ
Yes
*02TXDQWLILFDWLRQ
No
Assay
individual
ingredients
Are they
Authorised ?
Yes/No
Illegal
Must be
Labelled ?
Yes/No
0.9% threshold
Less than 0.9%
No need for
labelling
More than 0.9%
Labelling
required
Metodi quantitativi per l’analisi di OGM
DNA
Monitoraggio in TEMPO REALE
dell’andamento della PCR
Analisi strumentale:
REAL TIME PCR
Proteine
Metodi
immunoenzimatici
ELISA
Raccomandazione EU 787/04 Percentuale di
DNA geneticamente modificato: percentuale
delle copie di DNA geneticamente modificato
rispetto alle copie di DNA specifico del taxon
bersaglio, calcolata in termini di genomi
aploidi
Sonde di ibridizzazione
SONDA ad IDROLISI TaqMan
Reporter (FAM,VIC, TET)
(Fluoresceina)
Quencher (TAMRA)
(Rodamina)
Trasferimento
di energia
5’
3’
Fam
Vic
Tet
Gruppo
Fosfato
Laser Excitation
TaqMan™ Fluorogenic
Probe
Reporter (FAM,VIC, TET)
(Fluoresceina)
5’
Quencher (TAMRA)
(Rodamina)
Distacco della sonda
3’
Fam
Vic
Tet
Gruppo
fosfato
Laser Excitation
CHIMICA TaqMan
Annealing
Denaturazione
• Polimerizzazione
R = Reporter (FAM,
VIC)
R
forward
primer
Q
Q
Q = Quencher
(TAMRA)
5’
3’
5’
5’
3’
5’
Taqman PCR Chemistry
Denaturation
Annealing
R = Reporter
Q = Quencher
• Polymerization
R
Q
Q
forward
primer
5’
3’
5’
5’
3’
5’
Taqman PCR Chemistry
R = Reporter
Q = Quencher
R
Q
Q
forward
primer
5’
5’
3’
3’
5’
5’
reverse
primer
Taqman PCR Chemistry
Denaturation
Annealing
R = Reporter
Q = Quencher
• Polymerization
R
Q
Q
forward
primer
5’
3’
5’
5’
3’
5’
. Taglio della sonda
R
Emissione di
fluorescenza
Q
Q
R = Reporter
forward
primer
5’
Q = Quencher
5’
3’
3’
5’
5’
8000
Campione
positivo
7000
1st cycle
5th cycle
10th cycle
Intensità di
Fluorescenza
15th cycle
20th cycle
6000
5000
25th cycle
30th cycle
35th cycle
Controllo
negativo
40th cycle
45th cycle
50th cycle
4000
3000
2000
1000
EMISSIONE del
EMISSIONE del
REPORTER
QUENCHER
0
480
500
520
540
560
1580
600
Lunghezza
wave length (nm) d’onda
620
640
660
REAL-TIME PCR
Permette di seguire la cinetica della
PCR mentre è in atto: DURANTE LA
FASE ESPONENZIALE
•Campioni vengono irradiati da
una sorgente luminosa
Fluorescenza
FLUORESCENZA
•viene rivelata da
una CCD camera
sotto il controllo
del computer
Cicli
Riduzione dei tempi di esecuzione dell’analisi
Elimina manipolazioni successive
VANTAGGI
all’amplificazione (minore possibilità di
contaminazione tra campioni)
Elimina l’impiego di reagenti radioattivi o tossici
(EtBr)
SISTEMA DI QUANTIFICAZIONE
Linea di base della
curva
Non è ancora misurabile
'Rn
Linea soglia
Taglia la curva del campione in
fase di crescita esponenziale
Ciclo soglia (Ct)
Ciclo in cui la curva di
amplificazione del campione
in fase esponenziale
taglia la linea soglia
0.5% OGM
1%OGM
Linea
threschold
5%OGM
0.1%OGM
Layout della piastra
S1
S1
S1
S2
S2
S2
S3
S3
S3
S4
S4
S4
EN
A
A
A
B
B
B
B
B
B
DO
1:10 1:10 1:10
1:10 1:10 1:10
GE
CCCNTC NTC NTC
NO
A
A
A
C+
C+
C+
S1
S1
S1
S2
A
A
A
A
1:10
C+
C+
C+
C1
S2
S2
A
A
1:10 1:10
C1
C1
S3
S3
S3
S4
S4
S4
B
B
B
B
1:10
B
1:10
B
1:10
NTC
NTC
NTC
C+ controllo positivo
C- controllo negativo
NTC Amplification reagent control
O
G
M
ANALISI QUANTITATIVA OGM
QUANTIFICAZIONE RELATIVA
DNA SPECIE
VEGETALE (lectina, invertasi,
DNA GM
(Soia RR, mais Bt 176-Bt 11 etc)
zeina, HMG)
% OGM
DNA soia RR
X 100
DNA soia
ANALISI QUANTITATIVA OGM
Calcolata
Calcolata sull’
sull’
INGREDIENTE
INGREDIENTE
Quantificazione relativa
DNA della SPECIE
DNA GM della Soia RR
VEGETALE SOIA (lectina)
(EPSPS)
DNA GM
%OGM =
DNA specie
CALCOLO DEL
CONTENUTO IN OGM
Curva di calibrazione costruita con
CRM (soia RR Fluka 0,1-0,5-1-2-5% RR)
1 Curva : mette in relazione la
differenza in cicli soglia tra target e
riferimento in funzione del log %OGM
20,00
18,00
16,00
14,00
12,00
10,00
8,00
6,00
4,00
2,00
0,00
¨Ct
Ct (OGM)-Ct(Lectina) = a log (% OGM) + b
-1,5
-1
-0,5
0
0,5
log (% OGM)
1
Metodo della “curva standard” copie genomiche
2 curve di calibrazione (transgene + gene di riferimento) basate sul
numero di copie genomiche
35,0
30,0
25,0
Ct
Raccomandazione EU 787/04
Percentuale di DNA geneticamente
modificato: percentuale delle copie di
DNA geneticamente modificato
rispetto alle copie di DNA specifico
del taxon bersaglio, calcolata in
termini di genomi aploidi
y = -3.3707x + 37.404
R 2 = 0.9991
20,0
y = -3.4481x + 41.303
15,0
R 2 = 0.9994
10,0
5,0
0,0
0,00
Copie genomiche soia RR
Copie genomiche soia
x100
1,00
2,00
3,00
4,00
lo g (10) c o pie g e no mic he
5,00
1 copia
•LOD valore al di sotto
del quale il metodo non
consente di
determinare la
presenza del parametro
analizzato
•LOQ indica il limite al
di sotto del quale il
metodo non consente di
determinare l’esatta
concentrazione del
parametro analizzato
20 copie
%OGM
0.001% (teorico)
0.01% (pratico)
%OGM
0.01% (teorico)
0.1% (pratico)
Principi di Base
Miniaturizzazione e automazione di saggi biologici
centinaia di migliaia di geni usati come sonde su una superficie
di 2-3 cm
Le sonde vengono attaccate su un supporto solido
Con l’aiuto della “Robotica”
L’informatica è una componente centrale in tutti gli
“step”
Tecniche Innovative
Extraction of
DNA
Selective
amplification
(marker sequences)
and labelling
Food sample
Activated
glass
surface with
fixed
specific
capture
probes
Hybridisation of PCR
products
Diagnosis : presence
of sequences specific
to the GMO
Latest GMOchips design
+ hy Ctl 3µM
+ hy Ctl 1.5µM
+ hy Ctl 750nM
+ hy Ctl 187.5nM
+ hy Ctl 93.75nM + hy Ctl 46.875nM
+ hy Ctl 11.72nM
+ hy Ctl 5.86nM
Bt11
Bt176
- hy Ctl
GA21
+ hy Ctl 375nM
+ hy Ctl 23.44nM
- hy Ctl
Mon810
RRS
Starlink a
Starlink d
Starlink e
Starlink f
OXY235-1
OXY235-2
OXY235-3
Mais
Soybean
Rapeseed
T25,T45,Topas
Tomato
nptII test
EF Bt11
Sugar beet
CaMV test (a)
EF Bt176
P35S test
Tnos test
CaMV test (b)
Pat gene
EF GA21
EF Mon810
EF RRS
EF Starlink
buffer
buffer
Fix Ctl 2.5nM
Fix Ctl 5nM
Fix Ctl 10nM
Fix Ctl 20nM
Fix Ctl 40nM
Fix Ctl 80nM
Fix Ctl 160nM
EF T25
GMOchips
T25 Mais
Pos Hyb CTL
Neg Hyb CTL
Specific capture probes for
Bt11
Capture probe for Pat
gene
Specific capture probes for
T25
Specific capture probes for
Mon810
Pos Fix CTL
Specific capture probes for
Bt176
Specific capture probes for GA21
Specific capture probe for
RRS
Capture
probe for P35S