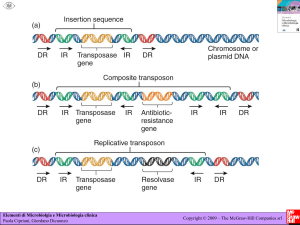
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
QUADRO 7.3 – BARBARA MCCLINTOCK E GLI ELEMENTI
GENETICI MOBILI (TRASPOSONI)
Figura 7.56
Cariossidi di mais
pigmentale (wild-type),
non pigmentale (colorless)
e pigmentate a settori (spotted)
risultanti dalla trasposizione
di elementi genetici mobili.
1
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
Figura 7.57a,b
Meccanismi di trasposizione:
(A) trasposizione conservativa;
(B) trasposizione replicativa
(da: R.J. Brooker 1999, modificata).
2
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
Figura 7.57c
Meccanismi di trasposizione: retrotrasposizione
(da: R.J. Brooker 1999, modificata)
3
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
Figura 7.58
Struttura e trasposizione
di un elemento trasponibile:
l'enzima trasposasi riconosce
le sequenze ripetute invertite
che fiancheggiano l'elemento
trasponibile, forma una struttura
ad anello inducendo l'escissione
dell'elemento stesso che in seguito
può reinserirsi in un diverso sito
genomico (da: B.B. Bunhanan
et al. 2000, modificata).
4
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
Figura 7.59
Struttura e trasposizione
di un retrotrasposone:
l'enzima trascrittasi inversa
produce una copia di DNA
usando come stampo un RNA
intermedio (da: B.B. Buchanan
et al. 2000, modificata).
5
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.60
Modello di rottura-fusione-ponte
in una coppia di cromosomi
(da: B.B. Buchanan et al.
2000, modificata).
6
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.61a
Sistema Ac/Ds di mais: effetti
della trasposizione sul colore
delle cariossidi.
7
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.61b
Trasposizione dell'elemento Ds
al locus C in presenza di Ac in mais
(l'endosperma di una cariosside
è triploide, in quanto deriva da due
nuclei aploidi materni e da un nucleo
aploide paterno)(Ds+ e Ac+ indicano
l'assenza di tali elementi).
8
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.62
Quadro sinottico
dei principali
effetti degli
elementi di
controllo,
con riferimento
al sistema Ac/Ds
(da: A.J.F.
Griffiths et al.
1999, modificato).
9
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.63
Rilevazione di una rottura
(fonte di instabilità)
cromosomica dovuta
all'azione dell'elemento Ds
in mais mediante analisi
cariologica e genetica
(da: A.J.F. Griffiths et al.
1999, modificata).
10
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Figura 7.64
Elementi del sistema Ac/Ds: struttura di Ac e dei membri Ds.
11
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Tabella 7.14
Trasposoni endogeni
caratterizzati nelle
specie vegetali.
12
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
ELEMENTI DI CONTROLLO Ac/Ds DI MAIS: PRIMO ESEMPIO
DI ELEMENTI TRASPONIBILI DESCRITTO PER GLI EUCARIOTI
Tabella 7.15
Trasposoni funzionanti
in sistemi eterologhi.
13
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
STRATEGIE DI MARCATURA TRASPOSONICA
Figura 7.65a
Strategie di marcatura
trasposonica: approccio mirato.
14
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
STRATEGIE DI MARCATURA TRASPOSONICA
Figura 7.65b
Strategie di marcatura
trasposonica: approccio casuale.
15
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
Figura 7.66
Meccanismo di funzionamento dei retrotrasposoni
(da: R.J. Brooker 1999, modificata).
16
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
Figura 7.67
Rappresentazione schematica di un retrotrasposone
o retroelemento virus-simile.
17
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
ELEMENTI Ty DI LIEVITO
Figura 7.68
Struttura di un elemento trasponibile di lievito (Ty1).
18
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
ELEMENTI COPIA-SIMILI, ELEMENTI GYPSY, ELEMENTI FB ED
ELEMENTI P DI DROSOPHILA
Figura 7.69a,b
Rappresentazione schematica
della struttura degli elementi
copia-simili (A), FB (B)
(da: A.J.F. Griffiths
et al. 1999, modificata).
19
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
ELEMENTI COPIA-SIMILI, ELEMENTI GYPSY, ELEMENTI FB ED
ELEMENTI P DI DROSOPHILA
Figura 7.69c
Rappresentazione schematica della struttura
degli elementi P (da: A.J.F. Griffiths et al. 1999, modificata).
20
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
RETROTRASPOSONI
RETROVIRUS, RETROGENI O RETROPOSONI
Figura 7.70
Ciclo vitale di un tipico
retrovirus (da: A.J.F.
Griffiths et al.
1999, modificata).
21
7.13 ELEMENTI GENETICI MOBILI
O ELEMENTI TRASPONIBILI
TRASPOSONI DI PROCARIOTI
Tabella 7.16
Principali tipi di trasposoni
procariotici ed aucariotici.
22
GENETICA FORWARD E REVERSE
Obiettivo: assegnare una funzione ad ogni gene
principali strategie sistematiche per risalire alla funzione di un
gene nelle piante. Sebbene molte delle tecniche descritte sono
utilizzate esclusivamente in ambito vegetale, le strategie generali
possono essere generalizzate anche ad altri sistemi.
Il metodo classico per determinare la funzione di un gene si basa sulla
ricerca e interpretazione di mutazioni in quel gene. Questo metodo si
basa sulla mutagenesi sistematica delle sequenze geniche e produce
collezioni di mutazioni (prevalentemente recessive) di tipo “loss of
function”.
La caratterizzazione genetica e funzionale di queste
mutazioni riesce, in molti casi, ad assegnare una funzione al gene in
analisi. A partire da questa informazione si risale alla sequenza genica,
utilizzando varie tecniche di clonaggio. Questo approccio che parte
dalla funzione per risalire al gene è noto come genetica diretta
(forward genetics).
La grande disponibilità di sequenze geniche conseguenti alla
diffusione delle tecniche di sequenziamento del DNA e,
specialmente, i grandi progetti di sequenziamento genomici, hanno
rivoluzionato l’approccio alla determinazione della funzione di un
gene. Frequentemente oggi si fa il percorso contrario; si parte da
una sequenza genica ben caratterizzata e si risale alla sua funzione.
Per esempio è possibile generare piante transgeniche ed esprimere
il gene in versione senso o antisenso, per studiarne il fenotipo e, da
questo, cercare di dedurre la funzione del gene. Formalmente questo
significa generare mutazioni (dominanti) di tipo “gain of function”
Allo stesso scopo, ma in maniera più sistematica, è possibile
analizzare apposite librerie di mutanti per identificare tutti i
mutanti di un gene d’interesse e conoscerne i fenotipi mutanti.
Questo approccio che parte dal gene per risalire alla funzione è
nota come genetica inversa (reverse genetics).
Fenotipo???
Fenotipo
FORWARD
Gene
Gene
mutante
T-DNA
REVERSE
PCR-screening
Funzione
Funzione???
Strategie
per studiare la
funzionemolecolari
di un gene nelle piante
Aumento espressione del gene endogeno:
• forte costitutiva
• ectopica
• inducibile
Riduzione/silenziamento del gene endogeno:
•• AntiSENSO
• RNAi
co-soppressione,
X Knock Out del gene.....
• genetica forward
• genetica reverse
Strategie
per studiare
la genetiche
funzione genica
Inattivazione
di un geneun fenotipo
per identificare
e caratterizzare
Non tutti i geni sono “mutagenizzabili” perché:
• (geni
geni funzionalmente
ridondanti
a
funzione
simile
ma non
omologhi)
(ridondanza
strutturale
v/s
ridondanza
funzionale)
•mutazioni
geni coivolti
in geni
diverse
fasi dello
sviluppo,
e
in
tali
possono
causare
fenotipi
letali o pleiotropici
Knock Out del gene.....
mutagenesiv/s
sito-specifica
mutagenesi inserzionale “random”
Nelle piante No mutagenesi sito-specifica !!!!!
Quindi............
mutagenesi inserzionale “random”
Strategie di Mutagenesi Inserzionale
a) T-DNA
• mutageno inserzionale che produce inserzioni stabili
• no “hot spots” di integrazione
b) Trasposoni
• il trasposone può essere exciso dal gene inattivato
(reversione del fenotipo = complementazione)
• eventi di trasposizione avvengono in zone limitrofe al sito di
inserzione
Transposon tagging
•Elementi
Il sistema
Ac/Ds (agente in trans)
Ac
: (4563 bp) codificano per una trasposasi
Elementi Ds: corrispondono ad elementi Ac deleti della trasposasi
Caratteristiche del sistema Ac/Ds in mais:
• contiene delle IR di 11-bp alle estremità, essenziali
• codifica per una proteina di 92kDa con attività di trasposasi
• 101 aa N-ter non necessari per la trasposizione
• circa 200 bp ad ogni estremità sono necessari per la trasposizione
• la trasposasi lega un esamero AAACGG
• dopo trasposizione vengono generati dei DR di 8-bp
• altri residui di DNA vengono lasciati a seguito della trasposizione
• gli elementi DS sono deleti o della trasposasi o delle IRs
Transposon tagging
• Barbara Mc Clintock è stato il 1°scienziato a predire che i trasposoni,
elementi genetici mobili, erano presenti nei genomi eucariotici. Lei ha
isolato il sistema Ac/Ds da mais.
• Il sistema Ac/Ds è stato utilizzato in Arabidopsis x generare
collezioni di mutanti. La bassa attività dell’elemento Ac, è stata
aumentata usando la trasposasi sotto il 35S. Cmq la frequenza di
reinserzione dell’elemento Ac è relativamente bassa. Ciò puo’ anche
dipendere da successive trasposizioni dell’elemento, dovuto a mancato
controllo dell’attività di trasposizione. Un’alternativa a ciò è l’uso della
trasposasi sotto controllo di un promotore HS.
• Elementi trasponibili isolati da mais e Antirrhinum sono utilizzati in
altre specie nelle quali risultano assolutamente attivi.
Vantaggi del Transposon Tagging
• Evitare mutazioni indesiderate (dovute all’evento di trasformazione).Cosi la
mutagenesi è separata dalla trasformazione perchè la trasposasi ed il
trasposone/T-DNA (Ds)sono trasformati in 2 diverse piante. La trasposizione
avviene solo in seguito all’incrocio tra le due linee.
• Ottenere mutazioni stabili che possono revertire. Una mutazione causata da un
trasposone inerte è stabile finchè il trasposone non è mobilizzato da una
trasposasi, in seguito a reincrocio. Quando il trasposone salta lascia il
“footprint”, ma il fenotipo PUO’ comunque revertire.
• Ottenere nuove mutazioni. Quando il trasposone salta e lascia il “footprint”
questo può causare una nuova mutazione (frameshift, inserzioni aminoacidiche)
•“Taggare” il gene d’interesse x inserzione. I trasposoni di tipo Ac fanno
trasposizioni “short-range”. Identificando una linea con trasposone inerte che
mappa vicino al gene d’interesse, si mobilizza il trasposone x incrocio con la
trasposasi e si seleziona il “salto” nel gene.
T-DNA tagging
• Inserzione del T-DNA nel genoma vegetale mediante una “illegitimate
recombination”
• No hot spots di integrazione
• Distribuzione random sui 5 cromosomi di Arabidopsis
• Produzione di >18000 FSTs (Flanking Sequence Tags) x analisi dei TDNA IS
RISULTATI
• 40% delle integrazioni sono in geni
• Nessuna categoria di geni è particolare bersaglio di integrazione
• FSTs più frequenti negli introni che negli esoni (43FSTs/Mb v/s
33FSTs/Mb)
• LB spesso integrato full-length, mentre RB risulta spesso troncata
• Coinvolgimento di “microsimilarità” tra LB e la sequenza vegetale preIS
• Integrazione influenzata anche dalla composizione nucleotidica
• Preferenza per regioni T-rich (i.e. promotori e introni)
Mutagenesi inserzionale “genomica”
Quante linee bisogna produrre per saturare il gen
Calcolo
teorico:
Il T-DNA si inserisce nel genoma seguendo una distribuzione di Poisson,
quindi……
F (geni con almeno 1 inserzione= saturazione) = 1 - e-m
dove
m rappresenta
il numero
medio di eventi per
gene, i.e.
m
=
numero
di
inserzioni
indipendenti/numero
di
geni
Se:
1.5
T-DNA
locus per line
75/85%
saturazione
con 25.000 T-DNA lines
Ma noi evidenziamo
solo
inserzioni in regioni codificanti,
quindi
….CORREZIONE!
95% saturazione
con circa 50.000 linee
indipendenti
Strategia di Isolamento Mutanti
In Planta
Vacuum
Infiltration
Raccolta semi T1
T0
Autoimpollinazione
T2
Analisi
in vitro
delle
plantule
Kr
Ks
Selezione
in serra
dei
trasformanti
Basta resistentiT1
Raccolta semi T2
Kr
•• Analisi
dei mutanti
Kana omo
resistenti
Propoagazione
di piante
ed eterozigoti
Knockology
Ma
il T-DNA è legato
(responsabile
del)
al fenotipo mutato???
•Analisi edidel
segregazione
del
marcatore
(inserzione T-DNAfenotipo
in 1 o piùmutante
loci??)
•Caratterizzazione
molecolare
del sito dipiù
inserzione
(T-DNA
singolo, a tandem
oppure inserzione
complessa??)
•Analisi
PCR sucon
moooooolti
mutanti
(es.
mutante
fenotipo
letale,
non è possibile ctrl la resistenza
a Kana)
•Clonaggio delle “Flanking Sequences”
Analisi di segregazione
T2 Plants aa(Kr/Kr)Aa(Kr/Ks)
AA(Ks/Ks)
96
193
94
383
1
2
1
autoimpollinazione
T3 Plants xy1(Kr) WT(Kr/Ks) Ks Total
a
24
47 26
97
b
c
e
25
27
121
46
55
00
24
28
121
95
110
Total
Ma(responsabile
il T-DNA è del)
legato
al fenotipo mutato???
• Analisiedidel
segregazione
del
marcatore
(inserzione T-DNA fenotipo
in 1 o piùmutante
loci??)
• Caratterizzazione
molecolare
del sito di
(T-DNA
singolo, a tandem
oppure inserzione
piùinserzione
complessa??)
• (es.
Analisi PCR su moooooolti mutanti
mutante con fenotipo letale,
non è possibile ctrl la resistenza a Kana)
• Clonaggio delle “Flanking Sequences”
x
0,23
4,4
E.RV E.RV
RB
GUS
E.RV
nos3’ ocs3’
KanR
LB
Pnos 35S
Erbic R
Digestione EcoRV:
e 4,4
Kb interne
T-DNA
x Kb 0.23
fino alKbsito
EcoRV
sul DNAalvegetale
NT
Caratterizzazione
Molecolare
del sito di inserzione
1,8 Kb RB-LB IR
0,23 Kb
4,4 Kb
1,2 Kb RB-RB DR
0,23 Kb
Ma(responsabile
il T-DNA è del)
legato
al fenotipo mutato???
• Analisiedidel
segregazione
del
marcatore
(inserzione T-DNAfenotipo
in 1 o piùmutante
loci??)
• Caratterizzazione
molecolare
del sito dipiù
inserzione
(T-DNA
singolo, a tandem
oppure inserzione
complessa??)
• Analisi
PCR sucon
moooooolti
mutanti
(es.
mutante
fenotipo
letale,
non è possibile ctrl la resistenza
a Kana)
• Clonaggio delle “Flanking Sequences”
Analisi per PCR
•seme/embrione
Estrazione DNA
dai singoli
se il fenotipo
è letale)mutanti
(da
•LB,
PCR
con oligo specifici per il T-DNA: Kana, RB,
Basta
•blotting
Analisi dei prodotti di PCR su gel/Southern
Ma(responsabile
il T-DNA è del)
legato
al fenotipo mutato???
• Analisiedidel
segregazione
del
marcatore
(inserzione T-DNAfenotipo
in 1 o piùmutante
loci??)
• Caratterizzazione
molecolare
del sito dipiù
inserzione
(T-DNA
singolo, a tandem
oppure inserzione
complessa??)
• Analisi
PCR sucon
moooooolti
mutanti
(es.
mutante
fenotipo
letale,
non è possibile ctrl la resistenza
a Kana)
• Clonaggio delle “Flanking Sequences”
Strategie
di clonaggio
delle
“Flanking
Sequences”
- Plasmid Rescue
- Inverse PCR (IPCR)
-con
Libreria
sonda genomica
T-DNA da DNA mutante, con screening
X
X
T-DNA
GUS Ori Amp Kana
Digestione
X
X
T-DNA
Ligazione intramolecolare
T-DNA
X
X
X
Trasformazione E.coli
Selezione colonie AmpR, su LB+Am
Strategie
di clonaggio
delle
“Flanking
Sequences”
- Plasmid Rescue
- Inverse PCR (IPCR)
-screening
Libreriacongenomica
da DNA
sonda T-DNA
mutante,
con
X
GUS
X
T-DNA
Kana
Digestione
X
T-DNA
X
Ligazione intramolecolare
T-DNA
X
PCR
CC
CC
Strategie
di clonaggio
delle
“Flanking
Sequences”
- Plasmid Rescue
- Inverse PCR (IPCR)
- Libreria genomica da DNA mutante
Libreria genomica da DNA mutante
•Kana
Digestione
del DNA
mutante
o di plantula
R (eterozigote)
con genomico
enzimi adeguati
x il(omozigote)
clonaggio
•genomiche
Clonaggio (l,
delcosmidi)
DNA genomico così digerito, in vettori x librerie
•E.coli
Trasformazione (per elettroporazione) di cellule competenti di
•Basta)
Screening della libreria genomica con sonda T-DNA (Kana, RB, LB,
• Isolamento dei cloni positivi, sequenza delle FS
•genomica/cDNA
Amplificazione WT
delle FS x utilizzarle come sonda su una libreria
•responsabile
Isolamento del
della
copia WT del gene/cDNA che, inattivato, è
fenotipo
Ildall’inserzione
fenotipo mutante
dipende
T-DNA,
cioé dall’inattivazione deldel
gene
X ???
DIMOSTRAZIONE
FORMALE:
COMPLEMENTAZIONE!!!!!!!
Complementazione:
Trasformazione
della
pianta
mutante con
una
copia
del
gene
WT,
al
fine
di
restaurare
la
funzione
mancante
•• p35S::cDNAX
• pX::cDNAX
pX::geneX
Un
esempio:
I mutanti pasticcino
pas1
B
E
N
E
N X S
X
pas1
WT
WT
V
RB
S N
GUS
E
T-DNA
V
N
LB
N
X