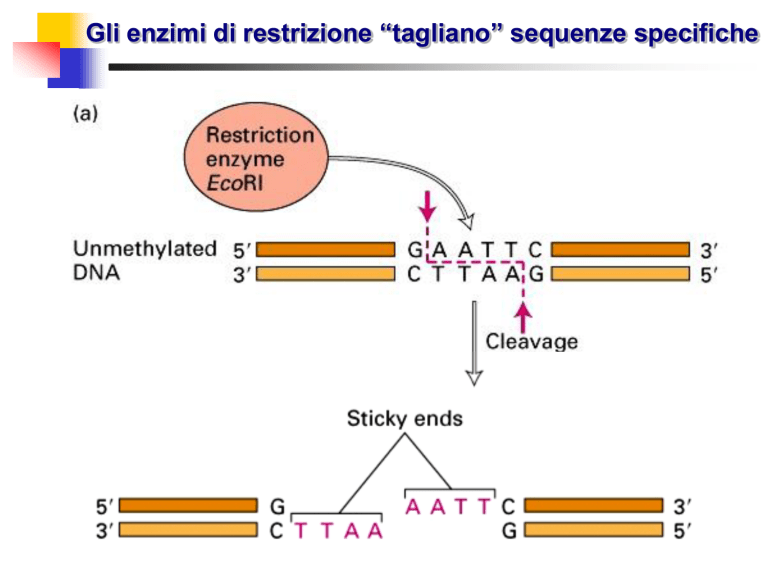
Gli enzimi di restrizione “tagliano” sequenze specifiche
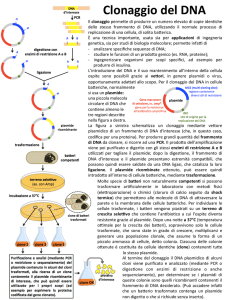
“Legatura” delle estremità coesive del DNA
DNA del gene da inserire
5’-C-G-G-T-A-C-T-A-G-OH
3’-G-C-C-A-T-G-A-T-C-T-T-A-A-PO4
DNA del plasmide
+
PO4-A-A-T-T-C-A-G-C-T-A-C-G-3’
HO-G-T-C-G-A-T-G-C-5’
Appaiamento delle estremità
complementari
5’-A-C-G-G-T-A-C-T-A-G A-A-T-T-C-A-G-C-T-A-C-G-3’
3’-T-G-C-C-A-T-G-A-T-C-T-T-A-A G-T-C-G-A-T-G-C-5’
+ DNA Ligasi, + ATP
5’-A-C-G-G-T-A-C-T-A-G-A-A-T-T-C-A-G-C-T-A-C-G-3’
3’-T-G-C-C-A-T-G-A-T-C-T-T-A-A-G-T-C-G-A-T-G-C-5’
Molecola di DNA ricombinante
Le “estremità coesive” facilitano la ricombinazione in vitro
Un vettore di clonaggio (plasmide)
circa 3000
coppie di
basi
ori
poli-linker
R
Amp
EcoR1
Pst1
Cla1
BamH1
Sst1
Not1
EcoRV
HindIII
Il Plasmide che useremo
origine della replicazione
Ampr
Gene LacZ
(colorazione)
Sito di “clonaggio”
pBSC (~3 kb)
Inseriremo un frammento di DNA di ~ 1000 basi
Da dove prendiamo il gene GFP…
1069
3323
2613
Da dove prendiamo il gene GFP…
1450
3323
2234
…e dove lo inseriamo !
45
Amplificazione e purificazione di un plasmide
"amplificazione" di un
"clone" batterico che
contiene il plasmide
di partenza
estrazione e
purificazione
del DNA
plasmidico
La “digestione” con enzimi di restrizione
Preparare 2 campioni:
1) PBSC (vettore); 2) P3-GFP (inserto)
DNA (pBSC ovvero P3-GFP)
Miscela di reazione (pH 7.5, forza ionica media)
Acqua (per portare a “volume”)
Enzima (Kpn1)
Enzima (BamH1)
Incubare a 37°C per 1-2 ore
5µl (5µg)
5µl (10x)
38µl (50 tot)
1µl (10U.E.)
1µl (10U.E.)
Miscela di caricamento (glicerolo, coloranti)
10µl (6x)
Caricare su gel di agarosio (30µl per “pozzetto”)
Separazione di frammenti di DNA mediante
elettroforesi su gel - I
Elettroforesi su Gel di agarosio
La purificazione dei frammenti dal gel
pBSC Kpn1+BamH1
P3-GFP Kpn1+BamH1
Inserimento del DNA nel plasmide
taglio con
enzima di
restrizione
DNA del
plasmide
(circolare)
frammento di DNA
da clonare (GFP)
legame
covalente
DNA ligasi
La reazione di ligasi
Preparare 3 campioni :
V) Vettore da solo (C+);
I) Inserto da solo (C-);
V+I) Vettore + Inserto
Vettore (pBSC Kpn1/Bamh1) purificato
Inserto (frammento di 1035 basi di P3-GFP)
Miscela di reazione (pH 7.5, forza ionica media)
ATP (fonte di energia)
Acqua (per portare a “volume”)
Enzima (DNA ligasi, estratta dal fago T7)
Incubare a 15°C “overnight”
2µl (100 ng)
5µl (250 ng)
2µl (10x)
2µl (10x)
8µl
1µl (1 U.E.)
“Trasformazione” dei batteri con DNA plasmidico
E. Coli (Amps )
Selezione per crescita in
presenza di ampicillina
membrana
permeabilizzata
con Ca2+
Ampr
batteri “competenti”
E. Coli + plasmide (Ampr )
Colonie positive (bianche) e negative (blu)
Purificazione di un plasmide ricombinante
"trasformazione" dei
batteri con il plasmide
ricombinante
"piastramento" dei
batteri trasformati
solo i batteri nei quali
é entrato il plasmide
formano le "colonie"
estrazione e
purificazione
del DNA
plasmidico
"amplificazione" di un
"clone" batterico che
contiene il plasmide
"ricombinante"
Analisi delle minipreparazioni di DNA
Estrazione del DNA da due colonie BIANCHE
Digestione con gli enzimi Kpn1 + BamH1
Miniprep. 1
M1 ND DIG
4268
3530
2027
1904
1584
1375
947
831
Miniprep. 2
ND DIG
M2
DNA tagliato da uno solo dei due enzimi
(digestione parziale) = plasmide linearizzato
2915 + 1035 = 3950 basi
pBluescript
2915 basi
1400
Gene GFP
1035 basi
517
396



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)