09/11/2011
Ritornando all’esempio precedente, io a questo punto posso ricercare le mutazioni della connessina
26 che è la più frequente: se la trovo mutata in tutti e due, allora il 100% dei figli avrà la sordità
congenita, se solo uno dei due è mutato, in questo caso la malattia è dovuta a due loci diversi e
quindi, anche se non conosciamo il secondo locus, sappiamo che i figli saranno sempre eterozigoti
per entrambi i loci e quindi mai ammalati.
Se nessuno dei due presenta mutazioni della connessina 26, allora probabilmente hanno mutazioni
in loci minori e quindi è assai improbabile che abbiano tutti e due la stessa mutazione, a meno che
non siano imparentati tra di loro.
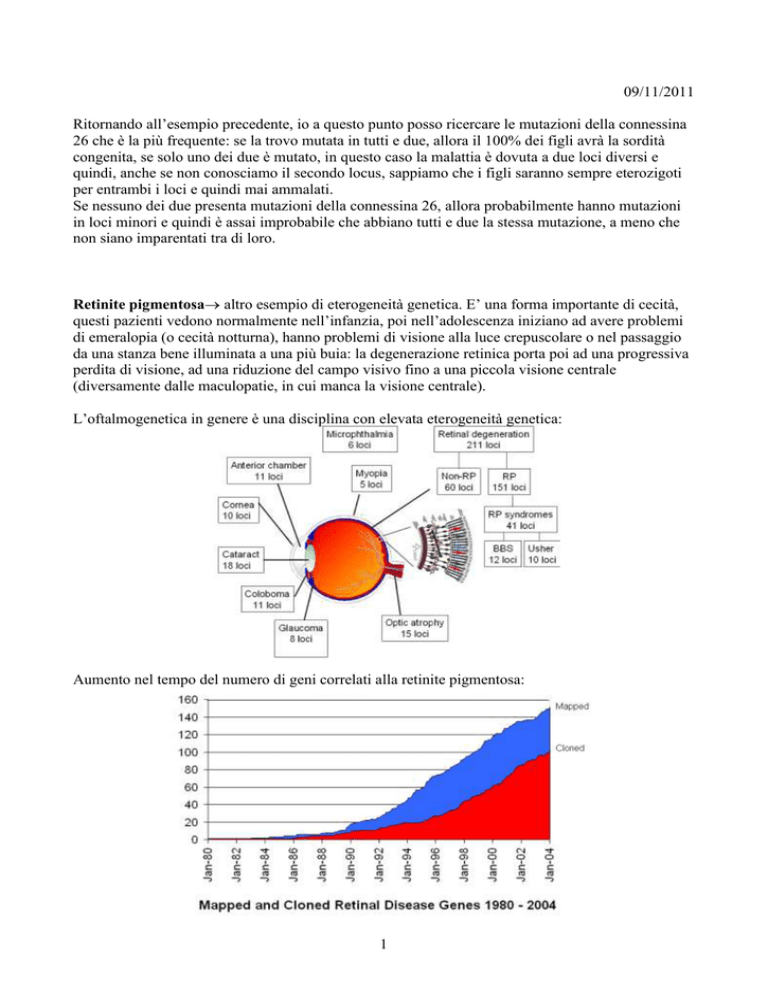
Retinite pigmentosa altro esempio di eterogeneità genetica. E’ una forma importante di cecità,
questi pazienti vedono normalmente nell’infanzia, poi nell’adolescenza iniziano ad avere problemi
di emeralopia (o cecità notturna), hanno problemi di visione alla luce crepuscolare o nel passaggio
da una stanza bene illuminata a una più buia: la degenerazione retinica porta poi ad una progressiva
perdita di visione, ad una riduzione del campo visivo fino a una piccola visione centrale
(diversamente dalle maculopatie, in cui manca la visione centrale).
L’oftalmogenetica in genere è una disciplina con elevata eterogeneità genetica:
Aumento nel tempo del numero di geni correlati alla retinite pigmentosa:
1
In questi pazienti si faceva uno studio di segregazione per valutare se la forma di retinite era
autosomica dominante, recessiva o X-linked, quando era possibile capirlo (mutazioni ex-novo).
Nei casi dominanti, vado a studiare per primi i geni della rodopsina e della periferina, i quali
coprono una percentuale rilevante di queste forme: se ci sono altri sintomi associati, come la sordità
nella sindrome di Usher (ipoacusia e retinite pigmentosa) si vanno a studiare le mutazioni
responsabili della sindrome.
Ci sono forme legate al cromosoma X con un gene ricorrente, e forme recessive in cui normalmente
si va a studiare il gene RTE65, che può causare l’amaurosi congenita di Leber (raro disturbo visivo
che comporta la cecità nei nascituri), forme di retinite pigmentosa, etc. per questa forma
specifica di retinite c’è la possibilità di sostituire il gene-malattia, ci sono trial clinici in corso.
Un’altra patologia con elevata eterogeneità genetica è la Sindrome di Noonan
ha una prevalenza di 1/2000 individui, condizione monogenica autosomica dominante ma con
un’ampia eterogeneità sia genetica che fenotipica (espressività variabile).
Il primo gene responsabile ad essere identificato è stato PTPN11, che copre circa il 50% dei
pazienti affetti da questa sindrome.
PTPN11 entra in questo pathway molecolare che è la via delle RAS-kinasi andando a studiare gli
altri geni che fanno parte di questo pathway, sono state trovate mutazioni che coprivano l’altro 50%
di pazienti che non avevano la mutazione di PTPN11 (patologie dette complessivamente
“rassopatie”).
Analisi mutazionale:
PTPN11 50%
SOS1
10-13%
K-RAS < 5%
RAF1
3%
Le caratteristiche cliniche di questa sindrome comprendono dismorfismi facciali come:
- fronte molto alta;
- ipertelorismo (distanza maggiore degli occhi tra loro);
- fessure palpebrali rivolte verso il basso;
- orecchie a impianto basso, retroruotate;
- attaccatura bassa dei capelli;
- bordi del labbro superiore accentuati;
- collo corto con cute nucale ridondante;
2
Altre caratteristiche tipiche sono:
- cardiopatia congenita (50-80%): nella maggior parte dei casi rappresentata da una stenosi
valvolare polmonare (20-50%) e cardiomiopatia ipertrofica (20-30%);
- bassa statura (40-50%): ritardo nella crescita post-natale;
- ritardo mentale, difficoltà di apprendimento (25-35%);
- diatesi emorragica (30%);
- Anomalie linfatiche pre-natali: igroma cistico, poliidramnios, idrope fetale (rara); postnatali;
- Anomalie renali (11%): spt. Idronefrosi; più rare: doppio distretto, agenesia/ipoplasia renale,
ectopia, anomalie di rotazione
- Anomalie cutanee: spt. cheratosi follicolare
- Anomalie oculari (95%): strabismo, ambliopia, nistagmo
Nelle forme conclamate, la diagnosi della sindrome di Noolan è abbastanza semplice: talvolta il
quadro clinico non è così chiaro e la diagnosi arriva molto in ritardo. A volte, questa viene
diagnosticata in un neonato affetto e tramite analisi genetica si scopre che gliel’ha trasmessa un
genitore che però mostra sintomi minimi concetto di ESPRESSIVITA’ VARIABILE
Ci possono essere individui che presentano la mutazione, ma hanno sintomi e segni minimi: questi
vanno identificati perché hanno il 50% di probabilità di trasmettere la mutazione ai figli.
Molti affetti hanno una mutazione ex novo.
rischio di ricorrenza per i fratelli trascurabile (<1%): mosaicismo germinale teoricamente
possibile ma mai riportato in letteratura
Il 30-75% degli affetti ha un genitore affetto da una forma molto lieve che viene riconosciuta solo a
posteriori: rischio per i fratelli: 50%. Vanno identificati perché potrebbe essere scambiata per
mutazione ex novo.
Esempio di penetranza incompleta ed espressività variabile:
Oloprosencefalia difetto della linea mediana. Raro, colpisce 1/16.000 nati vivi, ma si pensa che
negli aborti precoci questo difetto abbia una frequenza molto elevata (circa 1 su 500). In questa
patologia, non si ha la separazione dei due emisferi cerebrali, che può essere:
- completa, cioè oloprosencefalia alobare, i due emisferi non sono separati
- semilobare, separati in parte
- lobare, quasi del tutto separati.
E’ molto eterogenea, sia genotipicamente che fenotipicamente.
Ci sono forme cliniche molto gravi, che possono arrivare fino alla ciclopia; ci sono difetti della
piramide nasale (radice unica centrale) e difetti di palatoschisi, microcefalia, ipotelorismo.
Coppia ha una figlia malata che muore pochi giorni dopo la nascita: viene effettuata la diagnosi e ai
genitori viene detto che spesso si tratta di anomalie casuali.
Hanno un altro figlio, sano.
Alla terza gravidanza, il ginecologo all’ecografia della 20esima settimana vede un difetto di
oloprosencefalia: la gravidanza viene interrotta.
Com’è possibile che da due genitori “sani” siano nati due bambini con la stessa mutazione?
3
È molto improbabile che entrambi abbiano sviluppato la stessa mutazione ex novo.
La madre presentava un incisivo unico mediano e un lieve ipotelorismo unici segni nella madre
Aveva il difetto genetico.
Estrema variabilità dell’espressività
la madre presenta solo un unico incisivo, mentre i figli hanno i
segni conclamati della patologia.
Si fa lo studio genetico di Sonic Hedgehog (coinvolto nel 20% delle forme familiari) e si trova la
mutazione responsabile del difetto nella famiglia; inoltre, viene effettuata anche l’analisi indiretta
Quarta gravidanza valutata con analisi dei villi all’11esima settimana: gravidanza interrotta.
Non si può determinare quale sarà l’espressività clinica della malattia perché dipende da molti altri
cofattori.
L’espressività variabile rappresenta proprio la presenza di spettri fenotipici differenti all’interno
della stessa famiglia dove sono presenti le stesse mutazioni.
4
Noi possiamo avere che alcuni individui affetti da una patologia hanno un’espressività conclamata,
mentre altri hanno minimi segni.
L’espressività variabile correla con la penetranza incompleta
PENETRANZA INCOMPLETA: percentuale di individui che, a parità di genotipo, mostrano
il fenotipo.
Se, per esempio, in una famiglia abbiamo dieci individui con una mutazione, ma solo sette
manifestano i segni clinici della malattia, significa che quella patologia ha una penetranza del 70%.
Quindi, un individuo con patologia autosomica dominante ha effettivamente il 50% di possibilità di
trasmetterla ai figli solo se quella malattia ha una penetranza del 100%: ma se una malattia ha una
penetranza ridotta, ci possono essere individui che non manifestano la malattia ma che possono
trasmetterla alla loro prole si parla di SALTO DI GENERAZIONE, è l’unico caso per le
patologie autosomiche dominanti (per le recessive, è comune il salto), caratteristico delle patologie
autosomiche dominanti a penetranza incompleta.
La penetranza incompleta non va confusa con l’espressività variabile individuo può avere
minimi segni che non sono stati rilevati e che sono la chiave di lettura per capire che
quell’individuo ha un rischio di ricorrenza elevato.
Sia la penetranza incompleta che l’espressività variabile sono dovute al fatto che una mutazione non
è isolata, ma si trova in un background genetico diverso per ogni individuo: tutte le varianti presenti
negli altri geni possono in qualche modo favorire o meno l’espressività clinica di un gene
responsabile di una malattia monogenica detti GENI MODIFICATORI
È facile capire che questo si verifica più comunemente per le patologie autosomiche dominanti,
dove l’espressione del gene-malattia è controbilanciata dall’allele normale e da fattori genetici o
ambientali che possono indirizzare la malattia verso una forma più o meno grave.
Quindi, tornando all’esempio dell’oloprosencefalia
sono stati identificati numerosi geni responsabili:
SHH (Sonic hedgehog)
3,7% sporadic cases
18 % familial cases
ZIC2
SIX3
TGIF
PATCHED1
5% sporadic and familial cases
1% sporadic and familial cases
1% sporadic and familial cases
TDGF1
Nodal /TGF-b pathway
SHH signaling
GLI2
FAST1
Fanno tutti parte di pathway molecolari specifici.
5
Proteine segnate in nero sono quelle responsabili dell’oloprosencefalia: a qualsiasi altezza io
interrompa la cascata, avrò sempre oloprosencefalia.
L’eterogeneità genetica deriva da una base molecolare, cioè geni diversi causano la stessa patologia
perché intervengono nella stessa cascata molecolare.
Esempio:
La prima paziente ha una mutazione in SHH ereditata dalla madre e in più una delezione di TGIF,
probabilmente di nuova insorgenza.
Nel secondo caso, il padre non era disponibile, la bimba ha una mutazione in SHH ereditata dalla
madre e una mutazione in TGIF ex novo o ereditata dal padre.
Nel terzo caso, non erano disponibili né la madre né il padre, la bambina presenta mutazione di
SHH e ZIC2.
6
Generazioni di individui con la mutazione, ma che presentavano difetti trascurabili, come l’incisivo
unico o l’ipotelorismo: nasce un figlio con oloprosencefalia conclamata.
sono intervenute ulteriori mutazioni in altri geni che fanno parte dello stesso pathway
concetto di ETEROGENEITA’ DIGENICA: per alcune patologie occorre avere mutazioni
in due geni diversi perché la patologia si manifesti.
L’effetto digenico può essere di due tipi:
1. la prima mutazione è sufficiente a dare un fenotipo, magari lieve: poi interviene la seconda
mutazione che modifica il fenotipo e lo aggrava (gene modificatore), come nell’esempio;
2. l’effetto di genico è sinergico, cioè servono entrambe le mutazioni perché la malattia si
manifesti.
eccezioni all’ereditarietà mendeliana
Ancora più complesso è il caso della retinite pigmentosa: ci sono forme a trasmissione digenica e ci
sono forme sindromiche la cui ereditarietà è ancora più complessa.
Sindrome di Bardet-Biedl: malattia autosomica recessiva caratterizzata da retinite pigmentosa;
polidattilia, obesità, ritardo mentale, ipogonadismo, displasia renale e bassa statura.
Sono stati identificati 12 geni (ad oggi 14) responsabili della medesima forma sindromica:
Type 1 (BBS1)
Type 2 (BBS2)
Type 3 (BBS3)
Type 4 (BBS4)
Type 5 (BBS5)
Type 6 (BBS6)
Type 7 (BBS7)
Type 8 (BBS8)
Type 9 (BBS9)
Type 10 (BBS10)
Type 11 (BBS11)
Type 12 (BBS12)
BBS1 (209901)
BBS2 (606151)
ARL6 (608845)
BBS4 (600374)
BBS5 (603650)
MKKS (604896)
BBS7 (607590)
TTC8 (608132)
PTHB1(607968)
C12ORF58 (610148)
TRIM32 (602290)
C4ORF24 (610683)
7
11q13
16q21
3p13-p12.
15q22.3-q23
2q31
20p12*
4q27
14q32.11
7p14
12q
9q33.1
4q27
La sindrome di Bardet-Biedl è normalmente omozigote recessiva nell’esempio c’è la stessa
famiglia analizzata prima per il gene BBS1 e poi per il gene BBS6
L’individuo 02 è eterozigote composto per il gene BBS1, ha due mutazioni in punti diversi della
sequenza dei due alleli, cioè non è omozigote recessivo (malattia recessiva può essere causata da
omozigosi recessiva, cioè stessa mutazione su entrambi gli alleli, oppure da eterogizosi composta,
cioè due mutazioni diverse sui due alleli che però inattivano ugualmente il prodotto genico
determinando la malattia): la prima mutazione è una mutazione missense, mentre la seconda
determina un codone di stop prematuro e quindi una proteina tronca.
Nello stesso individuo, è mutato anche un allele del gene BBS6, quindi ha tre alleli alterati a livello
dei geni che causano la sindrome.
Stessa cosa
Si parla quindi di EREDITARIETA’ TRIALLELICA
nell’ultimo esempio, il padre è omozigote per una mutazione su BBS1, ma non è malato, mentre
la madre molto probabilmente è portatrice di un’altra mutazione: i figli sono malati perché ereditano
tutte e tre le mutazioni. In alcune famiglie, sono necessari tre alleli mutati per causare la
manifestazione clinica della patologia, e si parla quindi di ereditarietà triallelica, mentre altre sono
normalmente omozigoti recessive per la stessa mutazione.
Per esempio, ci sono un 70% di individui che con 2 mutazioni al
locus BBS1 che presentano la malattia, mentre un 10% ha due
mutazioni a BBS1 più una mutazione in un altro locus e una serie di
individui che hanno una mutazione al locus BBS1 e probabilmente
altre due mutazioni non identificate.
La malattia quindi è complessa non solo per l’elevata eterogeneità
genetica, ma anche perché ha una porzione di pazienti che si
comportano come omozigoti recessivi e altri che necessitano di tre
alleli mutati.
8
10/11/2011
Ci possono essere differenti mutazioni all’interno dello stesso gene che causano fenotipi diversi tra
di loro SERIE ALLELICA
Si tratta di diversità fenotipiche basate su serie alleliche
Esempio
Location
Disease
Disease
Gene
Elliptocytosis (130600)
SPTA
1q21
Pyropoikilocytosis (266140);
spherocytosis, recessive (270970)
Hemorrhagic diathesis due
to factor V deficiency
(AR) (227400)
F5
1q23
Thrombotic diathesis due to
deficiency of cofactor for activated
protein C (AD) (188055)
Achondroplasia (100800)
FGFR3
4p16.3
Hypochondroplasia (14600)
Hurler syndrome (607014)
IDUA
4p16.3
Scheie syndrome (607016)
Cystic fibrosis (219700)
CFTR
7p31.2
Bilateral absence of vas
deferens (277180)
Myotonia congenita,
dominant (160800)
CLCN1
7q35
Myotonia congenita,
recessive (255700)
Wolman disease,
infantile form (278000)
LIPA
10q24-q25
Cholesteryl ester
storage disease (278000)
Sickle cell anemia
(141900.0243)
HBB
11p11.5
Erythrocytosis due to Hb Osler
(141900.0211)
…etc.
Alcune mutazioni danno due diverse patologie a seconda della trasmissione
una forma di paraparesi spastica ereditaria a trasmissione dominante, in casi di forme recessive
da una lipodistrofia generalizzata congenita.
Ci sono anche alcuni oncogeni che, oltre a dare forme tumorali, possono dare anche patologie
congenite
Esempio
gene RET: primo gene coinvolto nel Morbo di Hirschprung, detto anche Megacolon congenito,
caratterizzato dall’assenza di gangli parasimpatici intrinseci della sottomucosa e del plesso
mioenterico.
Ha un’incidenza di 1/5000 nati vivi; si presenta con una severa costipazione, distensione
addominale, i bambini alla nascita non riescono ad espellere il mecomio e devono essere operati
urgentemente perché vanno incontro a ostruzione intestinale.
La causa molecolare è l’arresto prematuro della migrazione delle cellule gangliari provenienti dalla
cresta neurale.
C’è un’ampia variabilità dell’espressività clinica, con forme:
- ultracorte
- corte
- limitate al sigma retto (sono la maggior parte)
- estese al colon discendente, traverso, ascendente.
9
In alcuni casi, l’agangliosi può interessare tutto il colon e la parte terminale del tenue: questi
pazienti vengono chiamati TCA = total colon agangliosis
L’analisi del cariotipo della prima bimba su cui si è studiato il Morbo d Hirschprung presentava una
delezione del braccio lungo del cromosoma 10.
Avendo trovato altri pazienti con delezioni simili sul cromosoma 10, si sono analizzati i geni
presenti nella regione minima di delezione comune: tra questi geni, c’era il proto-oncogene RET.
RET è un recettore tirosin-kinasico: il legame con i suoi ligandi lo porta a dimerizzare e ad
autofosforilarsi, attivando una serie di pathway, tra cui quello delle MAP kinasi.
Mutazione di RET sono associate alle sindromi MEN2A e MEN2B,
cioè neoplasie endocrine multiple, e queste sono localizzate
soprattutto nel dominio ricco in cisteine, mentre mutazioni
localizzate un po’ lungo tutto il gene sono responsabili del Morbo di
Hirschprung, soprattutto delle forme familiari dominanti che sono anche
quelle più estese.
chiaro esempio di serie allelica
= missense or nonsense mutation
= deletion
= insertion
= splice mutation
= the same mutation associated with both MEN2A or FMTC and HSCR
promoter
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
c-RET protein
1114 aa isoform
Signal
peptide (1-28)
extra-cellular domain
TMD
(636-657)
Tyrosine Kinase domain
(726-999)
10
20
21
Rosso mutazioni responsabili del Morbo di Hirschprung
Verde mutazioni responsabili di MEN2A
Giallo mutazioni responsabili di MEN2B
Quindi questo è un esempio perfetto di serie allelica: mutazioni sullo stesso gene determinano
malattie molto diverse, come un’anomalia congenita e delle sindromi cancerose.
Il Morbo di Hirschprung è anche un esempio di eterogeneità genetica, poiché dopo l’identificazione
del primo gene, se ne sono individuati molti altri:
GDN
F
NTN
ART
PSP
ECE1
PRE PRO EDN
EDN3
H2
N
GFRa1
GFRa2
RET
RET
SOX10
GFRa3
GFRa4
RET
COOH
EDNRB
ZFHX1B
KIAA1279
-
mutazioni in alcuni dei ligandi di RET
mutazioni nel sistema endotelina-3/recettore dell’endotelina
mutazioni nell’enzima che attiva l’endotelina
mutazioni in alcuni fattori di trascrizione (SOX10; ZFHX1B responsabile di una
particolare forma di Morbo di Hirschprung, la sindrome di Mowat-Wilson, rara condizione
genetica caratterizzata da un fenotipo cranio-facciale tipico -lobo dell’orecchio a “globulo
rosso” e un sopracciglio diradato nel terzo medio- , deficit cognitivo da moderato a severo,
epilessia e anomalie congenite multiple di vario tipo compresi anomalie genitourinarie, in
particolare ipospadia nei maschi, cardiopatie congenite, agenesia del corpo calloso e
anomalie oculari, oltre che da megacolon congenito; KIAA1279)
Quindi, da una parte il gene RET è un esempio di serie allelica, dall’altra il Morbo di Hirschprung è
caratterizzato da un’ampia eterogeneità genetica.
Un caso particolarmente importante di penetranza ridotta sono le MALATTIE AD
INSORGENZA TARDIVA serie di malattie non congenite, ma che si presentano più avanti,
nell’adolescenza o nell’età adulta, sebbene l’alterazione genetica che le causa sia già presente alla
nascita.
I motivi per cui una malattia ritardi così a lungo la sua insorgenza possono essere diversi:
- lento accumulo di sostanze nocive: quando raggiungono una certa quantità, si avrà il
danno dovuto alla morte contemporanea delle cellule colpite
11
-
-
morte rallentata di certi tessuti
incapacità di riparare certe forme di danno ambientale: es. mutazioni del DNA che si
accumulano
geni oncosoppressori: modello del “doppio colpo”, un allele alterato viene ereditato, ma
la patologia si manifesta quando l’altro allele viene messo fuori uso da un’altra
mutazione
cause sconosciute
A
probabilità di un individuo che ha il gene malattia di sviluppare i sintomi ad una certa età.
B
rischio di un individuo figlio di un affetto di portare il gene malattia ad una determinata età.
In caso di una patologia dominante a insorgenza tardiva, un individuo alla nascita ha il 50% di
probabilità di essere affetto se uno dei due genitori era ammalato, ma alla nascita non si vede.
La probabilità di un individuo figlio di un affetto di essere portatore per un gene-malattia alla
nascita è del 50%, ma con gli anni, visto che non si sviluppano sintomi, la probabilità diminuisce
fino a sfiorare lo zero intorno ai settant’anni; un individuo con il gene-malattia ha probabilità
sempre crescente, man mano che passano gli anni, di sviluppare la malattia, tanto che a settant’anni
ha sicuramente manifestato i segni della patologia.
Questi grafici venivano utilizzati per valutare il rischio di un individuo ad una certa età di
sviluppare la malattia: oggi si fa la ricerca diretta del gene-malattia responsabile.
Legato alle malattie ad insorgenza tardiva, c’è il fenomeno dell’ANTICIPAZIONE, che potrebbe
essere considerato un caso particolare di espressività variabile: l’anticipazione indica la tendenza da
parte di alcune malattie genetiche a trasmissione autosomica dominante di divenire più gravi nelle
generazioni successive e talvolta anche di anticipare l’epoca di insorgenza.
Non va però confusa con l’espressività variabile: ci possono essere situazioni in cui nell’ambito
della stessa famiglia ci sono individui con la medesima patologia in forme più o meno gravi, ma
non per questo significa che c’è anticipazione, che deve per forza essere da una generazione
all’altra. In alcuni casi, può essere riferita ad errori nella diagnosi: per esempio, in un genitore si
arriva alla diagnosi tardivamente, se il figlio ha ereditato la malattia, ai primi segni si fa subito la
diagnosi, ma non per questo significa che la malattia sia insorta prima, l’abbiamo diagnosticata più
precocemente perché era già stata riscontrata nel genitore, il quale magari alla stessa età del figlio
presentava gli stessi sintomi che non erano stati presi in considerazione all’epoca.
Il fenomeno dell’anticipazione è un fenomeno particolare, spiegato da un meccanismo molecolare
12
l’instabilità di certi trinucleotidi repeat che possono espandersi in certe malattie (X - Fragile,
Distrofia Miotonica, Malattia di Huntington). La severità e l’età di insorgenza in queste malattie
correla con la lunghezza del repeat e questa tende ad espandersi attraverso le generazioni.
Esempio Malattia di Huntington
Patologia autosomica dominante a penetranza completa e insorgenza tardiva che presenta il
fenomeno dell’anticipazione
Per ogni individuo affetto è segnata l’età della diagnosi che è sempre più precoce.
Descritta per la prima volta nel 1873 da Huntington, è una forma di coreoatetosi, i pazienti hanno
difficoltà a camminare, hanno tremori e vanno incontro a demenza.
Il gene è stato identificato sul cromosoma 4: codifica per una proteina, detta Huntingtina, che
nell’esone 2 contiene un trinucleotide repeat (CAG) che codifica per la glutammina.
Nei soggetti normali, questo trinucleotide è ripetuto da 9 a 32 volte nel gene; quando questo
trinucleotide super le 37-39 ripetizioni, si ha la malattia.
Tra 32 e 37 il paziente non è ammalato, ma è a rischio di avere figli con la malattia, poiché si trova
nella fascia grigia di pre-mutazione in cui di per sé quel numero di ripetizioni non determina nel
paziente la malattia, ma fa sì che nella meiosi (soprattutto quella maschile) questa regione sia
instabile e possa espandersi e se trasmette un gamete con un numero di triplette superiore a 37
l’individuo che nascerà svilupperà la malattia.
La correlazione tra numero di ripetizioni e gravità/età d’insorgenza della malattia è diretta:
- numero di ripetizioni < 50 esordio tardivo della malattia e forma meno grave
- numero di ripetizioni > 60 esordio giovanile e forma più grave
L’anticipazione non è altro che un’insorgenza più precoce e più grave della malattia ed è dovuta
all’espansione di queste triplette durante le meiosi di individui portatori della pre-mutazione o della
mutazione vera e propria.
Oggi è possibile fare la diagnosi diretta ma con molti problemi etici.
13
11/11/2011
Acondroplasia malattia a trasmissione autosomica dominante, penetranza completa: nella
maggior parte dei casi i bimbi affetti nascono da genitori sani, come in questo caso
Genitori vanno in consulenza e chiedono che probabilità hanno di avere un altro figlio
acondroplasico escludendo la non paternità, si tratta di una mutazione “de novo”, la cui
probabilità di ricomparsa è molto bassa, a meno che non si tratti di MOSAICISMO
presenza in un individuo di due o più linee cellulari tra loro geneticamente diverse causate da
mutazioni insorte post-zigote, perciò le mutazioni interesseranno solo alcuni tessuti o addirittura
uno solo, come ad esempio le cellule germinali: in questo caso, l’individuo non è malato, ma
avendo la mutazione nelle cellule germinali, c’è rischio di ricorrenza della malattia nella famiglia
(rischio è meno dell’1%, ma può capitare, mentre per una coppia con figlio con osteogenesi
imperfetta, il rischio di ricorrenza è del 5/6%). In certe patologie quindi, questo fenomeno può
essere consistente.
Più gameti con la
stessa mutazione
Potrebbe sembrare la trasmissione di una patologia recessiva, ma io so che l’acondroplasia è
dominante, quindi devo subito pensare che si tratti di mosaicismo.
L’unica cosa che si può fare è, una volta individuata la mutazione del primo figlio, escludere che il
feto presenti la stessa mutazione nelle successive gravidanze.
Il gene responsabile è il recettore FGFR3, espresso ad alti livelli sulla membrana cellulare dei
condrociti negli abbozzi cartilaginei delle ossa. Normalmente il legame con il ligando stimola la
maturazione della cellula, facendone cessare la proliferazione.
Le mutazioni di FGFR3 responsabili di acondroplasia fanno sì che il recettore sia sempre attivato,
anche quando non c’è il suo ligando, cosa che compromette la normale maturazione delle ossa.
ECCEZIONI RIGUARDANTI L’EREDITARIETÀ’ X-LINKED
Una donna può essere affetta da una patologia recessiva legata all’X solo quando è figlia di un
uomo malato e di una donna portatrice (deve essere omozigote).
14
xy
xx
xy
xx
xy
xx
xy
xo
xy
xy
xy
xx
xx
xy
xy
Lyonizzazione sbilanciata
Nella prima famiglia c’è un maschio ammalato perché ha ricevuto l’allele malattia, un maschio sano
e una figlia malata perché ha ricevuto due alleli alterati; nel secondo caso, il padre è sano e la madre
portatrice, quindi il maschio che riceve l’allele mutato sviluppa la patologia, ma anche la femmina è
malata, perché? sindrome di Turner: ha una sola X, in particolare solo quella malata ereditata
dalla madre. Questa è una concomitanza di eventi rara; in questo caso sviluppa la malattia anche se
ha un solo X malato.
Nella terza famiglia, la figlia malata è eterozigote come la madre, ma manifesta la malattia
diversamente dal genitore: ciò è dovuto all’inattivazione dell’X fenomeno della
LYONIZZAZIONE SBILANCIATA: negli individui normali in seguito a Lyonizzazione sia gli
individui maschi che le femmine hanno un solo X attivo per cellula. Questo processo di
compensazione di dose tende a compensare gli effetti di copie multiple di geni legati alla X
(“compensazione di dose”). Responsabile dell’inattivazione della X è il gene XIST che viene
espresso esclusivamente dal cromosoma X inattivo. Tutte le donne quindi sono un mosaico
composto da circa il 50% di cellule in cui è inattivato il cromosoma X paterno e un 50% in cui è
inattivato il cromosoma X materno: questo fenomeno fu scoperta da una ricercatrice di nome Lyon.
Talvolta si può andare incontro a una lyonizzazione sbilanciata donne che inattivano per esempio
il 90% di un cromosoma X e solo il 10% dell’altro: se il cromosoma X attivo nella maggior parte
delle cellule porta un gene-malattia la donna può manifestare i segni clinici di quella patologia.
Se si osservano nuclei di cellule femminili normali XX si osserva una massa di cromatina
fortemente condensata che non è presente nei nuclei delle cellule maschili normali XY.
Si tratta del corpo di Barr e rappresenta un cromosoma X altamente condensato e quindi inattivo.
Questo fenomeno è stato studiato da Mary Lyon e pertanto è detto “LYONIZZAZIONE”.
I postulati sono:
a) Il corpo di Barr è un cromosoma geneticamente inattivo.
b) L’inattivazione avviene circa al 16° giorno dopo la fecondazione.
c) Il cromosoma X che viene inattivato è scelto a caso tra i cromosomi X materno e paterno,
secondo un processo indipendente da cellula a cellula (una volta che un cromosoma X è inattivato
in una cellula, tutta la progenie cellulare eredita lo stesso tipo di inattivazione).
d) Il numero di corpi di Barr, e perciò di cromosomi X inattivati, è uguale al totale dei cromosomi X
– 1 (una volta il conteggio dei corpi di Barr veniva utilizzato per diagnosticare le aneuploidie che
interessano i cromosomi sessuali).
Test di Humara test che si usa per valutare la lyonizzazione: viene utilizzato il promoter del gene
Humara (Human Androgen-Receptor) che contiene una regione polimorfica (CA-repeat).
Normalmente nella regione promotrice ci sono isole CpG che vengono metilate per inattivare
l’espressione di quel gene: ci sono degli enzimi di restrizione che sono sensibili alle metilasi (se c’è
15
una metilazione, non tagliano più) ed enzimi insensibili alle metilasi. Quindi, se digeriamo il DNA
di una femmina con l’enzima HPA2, questo non taglierà le regioni metilate, mentre taglierà solo le
regioni non metilate; mentre, se si utilizza l’enzima MSP1, questo taglierà tutte le regioni in cui
riconosce la sua specifica sequenza, che siano metilate o meno.
A monte del polimorfismo c’è una sequenza specifica riconosciuta da entrambi gli enzimi: nel
riquadro ci sono due donne, una con inattivazione sbilanciata e l’altra con inattivazione bilanciata
random. Quando faccio il genomico, non rilevo nulla, perché in entrambe ho due picchi, uno un po’
più grande e uno un po’ più piccolo. Se digerisco il DNA con MSP1 verrà digerito tutto e non vedrò
nulla; ma se digerisco con HPA2, verrà digerito solo il cromosoma X in cui il gene non è metilato,
cioè la copia attiva, mentre non riuscirà a digerire la copia inattiva. Nel primo caso, è evidente lo
sbilanciamento a favore di una delle due copie dell’X.
Talvolta, quando c’è un difetto genetico importante sul cromosoma X, si ha un’inattivazione
sbilanciata che tutela la donna dall’insorgenza di quella malattia: questo è ciò che accade
normalmente nei casi di TRASLOCAZIONI X-AUTOSOMICHE.
X
der
21
2
der X
21
der 2
X
der
X
Le traslocazioni possono avvenire anche tra un cromosoma X e un autosoma.
Quando non c’è perdita di materiale genetico, si parla di “traslocazione bilanciata”: donne portatrici
di traslocazioni X bilanciate, al momento dell’inattivazione dell’X inattiveranno porzioni
dell’autosoma traslocato, mentre presenteranno doppia copia di alcuni geni dell’X (di quelli
traslocati sull’autosoma). Queste cellule sono sfavorite rispetto alle altre cellule e muoiono: nelle
donne adulte troveremo un’inattivazione sbilanciata a favore delle cellule che hanno attivo il
cromosoma X traslocato, poiché in questo modo non si andrà ad inattivare una parte dell’autosoma
e i geni dell’X saranno in singola copia.
16
In questi casi, se il punto di rottura della traslocazione interessa un gene, quella donna manifesterà
una malattia X-linked recessiva per un’inattivazione sbilanciata primo esempio sopra: la rottura
a livello del cromosoma X rompeva il gene per la distrofia di Duchenne (cromosoma attivo in tutte
le cellule era appunto quello traslocato, come sempre, che presentava il gene interrotto e quindi non
funzionante in tutte le cellule).
Per certi caratteri, l’assenza di un allele normale è letale prima della nascita.
Per caratteri X-Linked con questa caratteristica i maschi non nascono e solo le femmine sono
affette: tra queste vi sono
- incontinentia pigmenti
- Goltz Syndrome o Focal Dermal Hypoplasia
- Sindrome di Rett
- Sindrome craniofrontonasale
Sindrome craniofrontonasale patologia particolare, con alterato sviluppo del massiccio
craniofacciale con marcato ipertelorismo, rime palpebrali rivolte verso l’alto, solco sulla punta del
naso, anomalie del corpo calloso, anomalie ungueali, possono mostrare un lieve ritardo mentale.
Questa malattia si manifesta nelle donne portatrici e non nei maschi malati.
L’albero mostra questo tipo di segregazione: la nonna ammalata ha trasmesso l’X malato sia al
figlio (sano) che alla figlia (malata) e ovviamente tutte le figlie del maschio portatore saranno
malate. Bisogna prestare attenzione perché la trasmissione avviene completamente al contrario delle
patologie X-linked classiche.
Ancora non si sa bene perché le femmine siano malate e i maschi poco affetti, ma l’ipotesi è che la
proteina non funzionante crei più danni in presenza di un 50% di proteine sane, da una sorta
d’interazione tra le due.
17
17-25/11/2011
Sindrome di Angelman grave ritardo mentale, non parlano, hanno microbrachicefalia, bocca
larga con lingua protrudente, denti spaziati, capelli biondi (65%).
Hanno difficoltà a camminare (atassia) e compiono movimenti stereotipati e ripetitivi, hanno crisi
epilettiche e alterazioni specifiche all’EEG.
Sindrome di Prader-Willi ampia variabilità nell’espressione e nella severità della malattia: alla
nascita questi bimbi hanno come caratteristica principale un’ipotonia importante che diminuisce col
tempo. L’altezza alla nascita è normale e diminuisce a partire dai primi due mesi di vita; presentano
obesità, rima palpebrale rivolta verso il basso, strabismo, ridotto diametro bifrontale, mani e piedi
piccoli, ipogonadismo, ritardo mentale variabile (da medio a grave), problemi comportamentali
soprattutto riguardo al cibo (non sono mai sazi) caratteristici.
Si tratta quindi di due malattie completamente diverse: ma da un punto di vista molecolare, con la
metodica FISH si vede un’identica delezione sul braccio lungo del cromosoma 15
mancanza della stessa porzione di DNA provoca due sindromi diverse.
Dipende dall’IMPRINTING GENOMICO: dipende dall’origine parentale, cioè se la delezione
avviene sul cromosoma 15 paterno, si avrà la sindrome di Prader-Willi, mentre se avviene sul
cromosoma 15 materno, si avrà la sindrome di Angelman.
Per un certo numero di geni autosomici l’espressione è monoallelica: i esprime solo la copia
ereditata dal padre o solo quella ereditata dalla madre.
L’imprinting descrive differenze nell’espressione di alleli paterni e materni di certi geni autosomici
nei mammiferi; per la stragrande maggioranza dei geni, l’espressione di un allele non dipende
dall’origine materna o paterna dello stesso (espressione biallelica).
Tuttavia per alcuni geni l’espressione dipende dalla loro origine materna o paterna, e tutto questo va
sotto il nome di imprinting genomico.
Talvolta l’imprinting può essere legato alla trasmissione di alcune patologie e quindi costituisce
un’eccezione all’ereditarietà mendeliana (modalità di trasmissione molto più complessa).
Quando l’imprinting può creare un problema?
quando alcuni geni sottoposti a imprinting genomico vengono deleti
Sapevamo che l’espressione di un dato gene può dipendere dal background genetico o
dall’influenza dell’ambiente, ma non si era mai pensato che essa potesse anche dipendere
dall’origine parentale.
Alcune osservazioni hanno fatto pensare a tutto questo:
Embrioni di topo manipolati in modo da possedere una copia del genoma materno o
paterno non si sviluppano sebbene posseggano un numero diploide di cromosomi.
Aborti umani triploidi sono fenotipicamente differenti e queste differenze dipende
dall’origine materna o paterna del genoma in più.
Certi caratteri autosomici dominanti si manifestano solo quando ereditati dal padre o
dalla madre.
L’imprinting sembra agire a livello trascrizionale: il meccanismo sembra coinvolgere la metilazione
del DNA, ma i dettagli sono complessi e non ancora compresi fino in fondo
18
Il cromosoma rappresentato nel disegno presenta due loci: A sul braccio corto e B sul braccio
lungo: nel gamete maschile, il locus B è sottoposto a imprinting genomico paterno (spento) e A no,
mentre nel gamete femminile, il locus A è sottoposto a imprinting genomico materno e B no.
Quindi è fondamentale che i geni sottoposti a imprinting paterno vengano ricevuti in copia intatta
dalla madre e viceversa.
Nella progenie maschile, durante la spermatogenesi, in qualche modo, un meccanismo riorganizza
l’imprinting in modo da trasmettere sempre il locus B spento e il locus A attivo: viceversa accade
nella progenie femminile.
Perciò ci sono meccanismi complessi che riconoscono i geni sottoposti a imprinting.
Le patologie legate all’imprinting vengono trasmesse nel seguente modo:
IMPRINTING MATERNO malattia trasmessa attraverso il padre
IMPRINTING PATERNO malattia trasmessa attraverso la madre
Esempio (imprinting paterno):
Legend
unaffected individual
2
4
I
1
2
Angelman Syndrome
fetus
mutation carrier
miscarriage
II
III
1
1
2
2
3
3
4
4
Una signora incinta di un feto di sesso maschile va in consulenza perché ha una sorella con tre figli,
di cui i due maschi affetti da un grave ritardo mentale pensava a un difetto legato all’X
I figli della sorella erano affetti da sindrome di Angelman: patologia di origine soprattutto sporadica
per delezione di parte del cromosoma 15 UBE3A è il gene sottoposto a imprinting paterno che,
se manca la copia materna o è alterata, può dare la sindrome.
I due individui hanno ricevuto dalla madre una copia di UBE3A con una mutazione: pertanto,
siccome il gene è sottoposto a imprinting paterno, tutte le copie ricevute dal padre sono inattive, la
19
copia della madre è alterata e quindi presentano la malattia. La figlia non è malata perché ha
ricevuto la copia sana: la madre non è ammalata perché la copia mutata era quella paterna, che le
era stata trasmessa con l’imprinting, e quindi spenta.
Anche il nonno aveva la copia mutata silenziata perché ricevuta dal padre.
La signora aveva ricevuto dalla madre la copia sana e quindi stava bene, ma dal padre aveva
ricevuto la copia mutata silenziata, ed era quindi a rischio di trasmissione.
DISOMIA UNIPARENTALE talvolta le cellule possono mostrare un corredo cromosomico
diploide normale (46 XX; 46 XY), ma in realtà possono mascherare un ineguale contributo paterno
e/o materno: il caso estremo è rappresentato dalla “diploidia uniparentale” dove tutti i cromosomi
derivano da un singolo genitore.
Una diploidia uniparentale comporta un mancato sviluppo embrionale nell’uomo.
La “mola idatiforme” rappresenta uno zigote con apparente corredo cromosomico 46 XX che non
sviluppa l’embrione. L’epitelio trofoblastico può trasformarsi in coriocarcinoma. Dipende da una
diploidia parentale paterna.
Il “teratoma ovarico”, al contrario, è rappresentato da una massa disorganizzata di tessuti
embrionali senza presenza di annessi extra-embrionali. Dipende da una diploidia uniparentale
materna.
Più spesso i casi di disomia riguardano un singolo cromosoma (uniparentale), che può essere:
ETERODISOMIA: se si hanno due copie dei cromosomi paterni o materni
ISODISOMIA: se si hanno due copie dello stesso cromosoma paterno o materno
Com’è possibile?
può essere dovuta ad un embrione trisomico (mancata separazione in meiosi) per un certo
cromosoma che perde una copia di tale cromosoma per restaurare un assetto cromosomico diploide
normale compatibile con la vita.
Potrebbe essere dovuta ad una pressione selettiva su di un embrione monosomico, determinando
così la duplicazione del cromosoma in singola copia per avere un normale embrione diploide.
Può causare malattie legate all’imprinting (esempio: riceve due copie materne di un gene con
imprinting materno, anche se non sono mutate, esprimerà la patologia perché non ha copie attive) e
patologie autosomiche recessive.
20
Ciascuno di noi ha due cromosomi 15, uno di origine paterna e uno di origine materna: il pallino
segna il centro che stabilisce l’imprinting, il quadrato rappresenta i geni responsabili della PraderWilli, mentre il triangolo rappresenta UBE3A.
I geni anneriti sono quelli sottoposti a imprinting e quindi spenti.
Entrambe le sindromi sono dovute a delezioni su quel tratto di cromosoma 15 o per disomia
uniparentale: ciò che determina una o l’altra sindrome è qual è il cromosoma alterato.
DNA MITOCONDRIALE
Ciò che noi vediamo è il fenotipo: nell’albero 1 a un certo punto compaiono due individui
ammalati, apparentemente senza spiegazione.
Ma se si analizza il genotipo, si vede che c’è una lunga serie di individui che mostrano mutazioni a
livello dell’mtDNA (M=muscolo, B=sangue), ma il fenotipo si esplicita solo negli ultimi due
individui dove le quote di mtDNA mutato superano una cera SOGLIA
le malattie dovute al DNA mitocondriale sono tutte malattie a soglia, devono superare una
determinata quantità di mtDNA mutato per manifestarsi.
21
Può creare confusione, perché può essere scambiato per una patologia recessiva: in realtà è una
trasmissione matrilineare, dove vengono trasmesse molecole di mtDNA mutato che sviluppano la
patologia solo se superano una certa quantità.
22
23
CARATTERISTICHE DELL’mtDNA:
POLIPLASMIA: in ogni cellula sono presenti molti mitocondri ed ogni mitocondrio
contiene multiple copie del suo genoma (eccetto piastrine e ovulo non fertilizzato)
migliaia di copie mtDNA per cellula. Durante la divisione cellulare i mitocondri
vengono distribuiti casualmente alle cellule figlie e quindi la genetica mitocondriale è
più simile alla genetica di popolazione che alla genetica mendeliana.
ETEROPLASMIA: in tessuti normali tutte le copie di mtDNA sono identiche
omoplasmia. Nel caso di una mutazione del mtDNA questa può colpire tutte le copie
oppure essere presente solo in una percentuale di genomi eteroplasmia.
Generalmente i polimorfismi neutrali sono omoplasmici mentre la maggior parte delle
mutazioni-malattia sono eteroplasmiche eteroplasmia cellulare o mitocondriale?
EFFETTO SOGLIA: l’espressione clinica delle mutazioni del mtDNA è determinata
dalla relativa proporzione wild type/mutato in un determinato tessuto; è necessario un
numero minimo di copie per danneggiare il metabolismo energetico di un determinato
organo o tessuto (valore relativo e non assoluto) (SNC, cuore, muscolo, rene e ghiandole
esocrine) (bilancio energetico).
SEGREGAZIONE MITOTICA: durante la divisione cellulare la proporzione di
genomi mutati può variare per deriva nelle cellule figlie, con conseguente cambiamento
fenotipico.
EREDITA’ MATERNA: virtualmente tutti i mitocondri dello zigote derivano
dall’oocita e perciò la modalità di trasmissione delle mutazioni mitocondriali differisce
dalla trasmissione mendeliana classica:
madre portatrice trasmissione a tutta la progenie, ma solo le figlie femmine possono
trasmettere la mutazione ai loro figli.
Eteroplasmia + effetto dose eccezioni fenotipiche all’eredità matrilineare.
CODICE GENETICO “PRIVATO”: una particolarità del codice genetico
mitocondriale sta nel fatto che esso è leggermente diverso da quello comunemente noto.
Il codone UGA, normalmente codone di stop, codifica per il triptofano. I vertebrati,
inoltre, usano la sequenza AUA (e l'uomo anche AUU) per codificare la metionina (e
non l'isoleucina) mentre AGA ed AGG funzionano come codoni di stop. Si è visto,
inoltre, che tra specie diverse vi possono essere differenze nel codice mitocondriale che,
di conseguenza, non è uguale per tutti.
Difetti dell’mtDNA possono essere dovuti a:
sostituzioni nucleotidiche
delezioni
inserzioni
24
Sostituzioni nucleotidiche generalmente associate a patologie neurologiche ed oftalmologiche
Leber’s Hereditary Optic Neuropathy (LHON)
Cecità ad insorgenza tardiva dovuta alla morte del nervo ottico è generalmente determinata da un
cambio arg-his (np 11778) nel gene ND4 presente in omoplasmia nella maggioranza dei pazienti,
ma può essere determinata da numerose altra mutazioni a carico dei complessi per il trasporto degli
elettroni, talora anche in combinazione: la probabilità di cecità aumenta negli individui con
mutazioni più gravi o nella combinazione di mutazioni diverse.
Neurogenic muscle weakness, Ataxia and Retinite Pigmentosa (NARP)
Retinite pigmentosa, atassia, convulsioni, demenza, debolezza muscoli prossimali di origine
neurogena, neuropatia sensitiva e ritardo nello sviluppo è determinata da un cambio leu-arg (np
8993) nell’ATPasi6; tale mutazione è sempre presente in eteroplasmia e la gravità dei sintomi è
correlata alla percentuale del DNA mutante.
Fosforilazione ossidativa (GENI OXPHOS): la via metabolica mitocondriale per la produzione
dell’energia necessaria alla cellula è composta da 5 complessi enzimatici le cui subunità sono
codificate sia da geni mitocondriali sia da geni nucleari.
I complessi I-IV permettono il trasporto di elettroni dal NADH + H+ e dal FADH2 fino
all’ossigeno, accettore finale della catena. Durante questo trasporto degli elettroni si ottiene un
flusso di protoni dalla matrice mitocondriale verso la membrana mitocondriale interna: questo
passaggio crea un gradiente elettrochimico che viene utilizzato dalla ATP sintetasi (complesso V)
per sintetizzare ATP.
La localizzazione nucleare di questi geni permette una regolazione tessuto-specifica del
metabolismo energetico:
isoforme tessuto-specifiche
espressione variabile dei geni nei diversi tessuti e durante lo sviluppo.
25
26
30/11/2011
PATOLOGIE CROMOSOMICHE
Normalmente, le patologie cromosomiche sono situazioni sporadiche, ma in alcune famiglie si
possono ripetere anomalie cromosomiche dovute ad alterazioni presenti nel DNA dei genitori.
Lo studio dei cromosomi è detto CITOGENETICA.
Un cromosoma è una molecola di DNA compattata: fa due giri e mezzo attorno al nucleosoma,
formando la cosiddetta “collana di perle”, poi si spiralizza ulteriormente fino all’immagine del
cromosoma in metafase.
27
Le malattie cromosomiche sono più complesse delle malattie geniche, poiché la perdita o la
presenza in triplice copia di una porzione molto ampia di DNA causa l’alterazione di più geni o
porzioni codificanti e quindi un quadro sindromico molto più complesso.
L’esame del cariotipo è l’esame più semplice per l’indagine del genoma: si prendono poche gocce
di sangue periferico e si mettono in terreno di coltura, dove viene stimolata la crescita dei linfociti:
successivamente si aggiunge colchicina che inibisce le cellule in metafase. Si tratta la coltura con
una sostanza ipotonica (KCl), poi si fissa il tutto su vetrino con una soluzione di 3 a 1 di metanolo e
acido acetico il tutto richiede solo 3-4 giorni
In seguito si bandeggiano: ci sono varie metodiche di bandeggio, quello con tripsina e Giemsa è
quello più usato (detto bandeggio G, mentre il bandeggio R in pratica è il negativo del G).
In passato, a questo punto si fotografavano al microscopio ottico i cromosomi in metafase, che
venivano poi ritagliati e appaiati col proprio omologo seguendo le dimensioni e il bandeggio.
Oggi, la metafase viene acquisita dalla fotocamera e ci sono programmi che riallineano i cromosomi
tra di loro: l’operatore deve controllare che l’allineamento sia corretto e che non vi siano anomalie
nel numero o nella struttura dei cromosomi.
Classificazione delle anomalie cromosomiche:
COSTITUZIONALI: presenti in tutte le cellule dell’organismo, presenti fin dal
concepimento in uno dei due gameti o nelle prime fasi di sviluppo
SOMATICHE: sono mutazioni post-zigotiche, a seconda del momento in cui
avvengono possono interessare molti tessuti o anche uno solo (mosaicismo genetico)
DI NUMERO: aneuploidie (monosomie, trisomie), poliploidie (numero di cromosomi è
pari a un multiplo del numero n aploide: triploidia, tetraploidia)
DI STRUTTURA: traslocazioni, inversioni, delezioni, duplicazioni.
28
Le cause di aneuploidia possono essere:
mancata separazione dei cromosomi alla prima o alla seconda divisione meiotica:
incapacità di cromosomi separati di appaiarsi durante la prima divisione meiotica, o dei
cromatidi fratelli appaiati di separarsi nella seconda divisione meiotica. I due cromosomi
o cromatidi congiunti migrano ad un polo e vengono inclusi in una sola cellula figlia,
mentre l’altra avrà materiale genetico in meno
ritardo nell’anafase: ritardata migrazione del cromosoma durante l’anafase, conseguente
perdita del cromosoma. Mancata incorporazione di un cromosoma nel nucleo di una
delle cellule figlie
Le anomalie di struttura dei cromosomi possono essere bilanciate o sbilanciate:
BILANCIATE: nella maggioranza dei casi non sono correlate a un fenotipo anomalo perché non c’è
acquisizione o perdita di materiale genetica.
SBILANCIATE: correlate a fenotipo anomalo (malformazioni o ritardo mentale) e tanto più è grave
lo sbilanciamento tanto più grave sarà il fenotipo. Se uno sbilanciamento è consistente, può causare
aborto precoce.
Esempio:
Si contano 45 cromosomi: le monosomie costituzionali sono incompatibili con la vita, solo la
monosomia X, Sindrome di Turner, è compatibile con la vita.
In realtà, è un cariotipo bilanciato: il cromosoma 21 è traslocato sul cromosoma 14, quindi non c’è
stata realmente perdita di materiale genetico
si tratta di una traslocazione Robertsoniana: si tratta di traslocazioni che interessano i
cromosomi acrocentrici (13, 14, 15, 21, 22) sono cromosomi che hanno il centromero spostato ad
una estremità. In questo caso, le braccia corte di questi cromosomi non sono altro che DNA
ripetitivo, DNA satellitare, che contiene porzioni non importanti del nostro genoma: può accadere
che due cromosomi acrocentrici perdano le braccia corte, dove non c’è materiale genetico rilevante,
e si fondano per il centromero, a dare quello che appare come un unico cromosoma (come in questo
caso).
29
Esempio:
In questo caso, contiamo 46 cromosomi: in realtà è sbilanciato, con una trisomia 21 in cui il terzo
cromosoma 21 si trova sul cromosoma 14.
Quindi, il bilanciamento o lo sbilanciamento non si riferiscono tanto al numero di cromosomi, ma
alla perdita o all’acquisizione di materiale genetico.
30
Nel caso di inversione, può accadere che:
1. la rottura sia sullo stesso braccio del cromosoma: se quel tratto si inverte, di parla di
INVERSIONE PARACENTRICA, inversione che non interessa il centromero. Altre volte
succede che viene perso il frammento di DNA e avviene una DELEZIONE
INTERSTIZIALE (il frammento non avendo centromero viene perso durante la meiosi)
2. la rottura comprenda il centromero (un punto di rottura sul braccio corto e uno sul braccio
lungo): in questo caso, si parla di INVERSIONE PERICENTRICA. Può accadere che
vengano perse le pozioni terminali del cromosoma: la porzione centrale, che non ha i
telomeri, si chiude a formare il CROMOSOMA AD ANELLO (spesso correlato a patologie
gravi perché si perdono pezzi importanti di genoma).
La gravità delle patologie cromosomiche è correlata al tipo di cromosoma coinvolto e alla quantità
di geni interessati: quindi più grave è lo sbilanciamento, tanto più precoce sarà l’interruzione di
gravidanza.
Nei casi di anomalie bilanciate, il problema non si pone per il soggetto portatore, ma per la sua
discendenza.
Un’anomalia bilanciata può causare un problema in colui che la porta quando per esempio avviene
una traslocazione reciproca: quell’individuo può presentare un quadro fenotipo quando i punti di
rottura interessano una sequenza genica, un gene che determina una patologia dominante (gene
sensibile all’aploinsufficienza), oppure quando la traslocazione determina un “effetto di posizione”,
cioè porta il gene in una regione trascrizionalmente inattiva, oppure quando si forma un gene
chimerico. Nella maggior parte dei casi la traslocazione cade in porzioni di genoma non codificanti
e quindi non si manifesta.
Se troviamo in un feto la stessa traslocazione presente nel genitore (che presenta un fenotipo
normale) il rischio di ricorrenza è molto basso, ci aspettiamo che anche nel figlio non ci siano
manifestazioni fenotipiche: se invece la traslocazione è ex novo, c’è il rischio di un quadro clinico
(il rischio di un’anomalia è quantificato in un 3,5 per punto di rottura in più).
Esempio:
31
L’anomalia nel genitore non si presenta perché è tutto bilanciato: alla meiosi, i cromosomi si
dividono e formano gameti con:
entrambi i cromosomi sani
entrambi i cromosomi con la traslocazione
In questi due casi, il genoma sarà bilanciato (un individuo sarà completamente sano e l’altro sarà
portatore della medesima anomalia del genitore).
Quando tuttavia si forma un gamete con un cromosoma integro e uno dei due traslocati, si avrà
sempre una porzione in più di un cromosoma e una di meno dell’altro (parziale trisomia e parziale
monosomia): queste sono situazioni di sbilanciamento. Le due situazioni di sbilanciamento sono
genotipicamente e fenotipicamente diverse e di conseguenza possono dare patologie diverse e
ricorrenti all’interno della stessa famiglia.
Esempio:
Anche per quanto riguarda le traslocazioni Robertsoniane è importante dare il rischio di ricorrenza
alla famiglia: se per esempio nasce un figlio con sindrome di Down, è importante verificare il
cariotipo di questo bimbo, perché nella maggior parte dei casi si tratta di una trisomia libera e
omogenea dovuta a una non disgiunzione (più frequentemente nella prima meiosi materna, per
fattori ancora non ben conosciuti), ma dobbiamo essere sicuri che uno dei due genitori non sia in
realtà portatore di una traslocazione Robertsoniana, come nel caso dell’esempio (14-21).
L’individuo portatore è sano perché è bilanciato: come prima, il problema si ha alla meiosi.
La frequenza delle anomalie cromosomiche è direttamente correlata con l’età materna e
inversamente proporzionale all’epoca gestazionale (cioè, se una gravidanza arriva a termine è più
improbabile che vi sia un’anomalia cromosomica, perché normalmente un’anomalia determina
un’interruzione precoce della gravidanza). A 30 anni, il rischio di avere un figlio con Sindrome di
Down è 1/1000: a 40 anni, 1/100.
Il numero di anomalie cromosomiche alla nascita è un po’ inferiore all’1%: se facciamo il cariotipo
32
fetale, escludiamo uno 0,65% circa, cioè si escludono le anomalie cromosomiche più frequenti e
che si possono vedere.
Bimbi con Sindrome di Down nati da donne con età diversa: i figli di donne oltre i 40 anni, per il
97% sono affetti da trisomia 21 omogenea, dovuta a non disgiunzione, quindi correlati all’età
materna, il 2% da una situazione a mosaico (germinali o somatici: ci sono persone che hanno il
2/3% di cellule con trisomia e non manifestano nessun sintomo) e l’1% da traslocazione.
Viceversa, i bimbi Down che nascono da ragazze di età inferiore a 20 anni, il 15% ha rischio di
ricorrenza (mosaicismi + traslocazioni): questo è importante per calcolare il rischio di ricorrenza di
Sindrome di Down in una famiglia, poiché se c’è una traslocazione o un mosaicismo in uno dei
genitori c’è un rischio concreto che l’anomalia si ripresenti.
Anche se la diagnosi della Sindrome di Down è clinica, è comunque importante stabilire il cariotipo
per capire come si è presentata l’anomalia, cioè se è una trisomia libera ed omogenea o se è dovuta
a traslocazione Robertsoniana o mosaicismo, poiché questo dopo cambia il rischio di ricorrenza
nella famiglia.
33
In ogni caso, la maggior parte di questi feti viene abortita spontaneamente:
Sindrome di Edwards (trisomia 18): malattia genetica rara che si manifesta con una frequenza di
1:7.000. I nati vivi sono prevalentemente femmine (per una possibile maggiore incidenza di aborti
spontanei tra i maschi affetti), e la frequenza della patologia aumenta all'aumentare dell'età della
madre. Viene anche chiamata trisomia 18 poiché il cariotipo di questi individui presenta tre
cromosomi 18 invece di due, è dunque un'aneuploidia. Nell'80% dei casi è dovuta a trisomia libera,
causata da una non disgiunzione meiotica per lo più materna; nel 10-15% è dovuta a una nondisgiunzione post zigotica con presenza di mosaicismo; nel 5-10% è dovuta a traslocazione
robertsoniana.
34
La sindrome si manifesta con malformazioni congenite multiple in quasi tutti gli organi, ciclopia,
ritardo generale di sviluppo, anche durante la gravidanza e, se sopravvivono, ritardo sviluppo psicomotorio; ipotonia muscolare; pugno chiuso con indice sovrapposto al medio (a uncino);
micrognazia (mento piccolo); impianto basso dei padiglioni auricolari; malformazioni cardiache e
renali, dermatoglifi specifici, piede equino. Un segno malformativo evidente in epoca prenatale
quale l'onfalocele (malformazione congenita grave nella quale i visceri addominali protrudono
all'interno di una sacca membranosa attraverso un difetto della parete addominale, generalmente
sott’ombelicale), può costituire un marcatore della trisomia 18 e rende opportuno lo studio del
cariotipo fetale. Il 90% dei bambini affetti muore nei primi sei mesi di vita per problemi cardiaci.
Sindrome di Patau (trisomia 13): malattia genetica molto rara con frequenza 1/5000, 1/20000
bambini nati, colpendo per lo più femmine. Il cariotipo dell'individuo che ne è affetto presenta tre
copie del cromosoma 13 invece delle normali due.
Le anomalie fenotipiche sono numerose: labioschisi e palatoschisi, polidattilia (dita delle mani e dei
piedi in soprannumero), occhi piccoli, ritardo psico-motorio, cardiopatia, encefalopatia, anoftalmia,
criptoftalmia o ciclopia. La maggior parte degli individui muore entro i primi tre mesi di vita.
Provoca danni al sistema nervoso centrale ed all'apparato cardiaco.
Sindrome di Turner: nota anche come disgenesia gonadica, è una patologia che interessa
esclusivamente soggetti di sesso femminile dovuta a un’anomalia del cromosoma sessuale X.
L’incidenza è di 1 femmina su 2500 nate. Nella gran parte dei casi è causata dall’assenza di un
intero cromosoma X in ciascuna cellula dell’organismo che normalmente, nei soggetti di sesso
femminile, ne possiede due, più raramente sono riscontrabili forme a mosaico (il cromosoma è
assente solo in alcune cellule mentre in altre è presente in duplice copia) e forme causate da
anomalie cromosomiche strutturali nelle quali uno dei due cromosomi X risulta in qualche modo
danneggiato. La Sindrome si manifesta in modo più sfumato in quei soggetti che presentano forme a
mosaico o causate da alterazioni strutturali, mentre è più evidente nelle forme dovute a monosomia
completa del cromosoma X. Le manifestazioni cliniche includono tipicamente bassa statura, 1,45 m
in media, torace a scudo (gabbia toracica ampia e piatta), collo corto, gomito valgo, gonfiore di
piedi e mani (linfedema periferico), caratteri sessuali secondari poco sviluppati a causa di anomalie
ovariche che comportano una carente produzione di estrogeni. Le donne affette da Sindrome di
Turner presentano inoltre amenorrea primaria (assenza del ciclo mestruale) o menopausa precoce.
Meno dell’1% è in grado di procreare.
Sindrome di Klinefelter: frequente disordine cromosomico causato dalla presenza di un
cromosoma X in soprannumero, omogenea o in mosaico. L'incidenza di questa condizione è stimata
35
pari a circa 1:700 nati vivi di sesso maschile. Tra i soggetti affetti nella popolazione generale,
attualmente solo un quarto viene diagnosticato. Circa il 10% dei casi di Klinefelter ha diagnosi
prenatale. Le prime caratteristiche cliniche possono comparire nella prima infanzia o, più spesso,
durante la pubertà, come il mancato sviluppo dei caratteri sessuali secondari, la microrchidia e
l'aspermatogenesi. La tendenza all'alta statura è difficilmente diagnosticabile durante la pubertà.
Nonostante la presenza di testicoli piccoli, solo un quarto di maschi affetti sono riconosciuti alla
pubertà. Il 25% viene diagnostica tardivamente in età adulta. Si stima che circa il 64% degli
individui affetti non vengano riconosciuti come tali. L'analisi dei cromosomi sui linfociti è lo
standard genetico di diagnosi. In passato si osservava il corpo di Barr. Per confermare il
mosaicismo si usano anche fibroblasti cutanei o tessuto testicolare
Diversamente da altre sindromi da polisomia dell'X che presentano ritardo mentale con una
prevalenza più alta, nella sindrome di Klinefelter solo il 10% presenta un ritardo mentale. I
problemi cognitivi sono meno pervasivi e più selettivi. Sul piano neurologico, la sindrome di
Klinefelter è associata a ridotto sviluppo del linguaggio, con problemi di espressività, anomia
(difficoltà a trovare i termini esatti con cui esprimersi), disartria (difficoltà ad articolare parole). Sul
piano comportamentale si possono riscontrare immaturità, poca sicurezza, timidezza.
La triploidia è un’anomalia che si verifica quando c’è un errore nella fertilizzazione:
2 contributi materni
2 contributi paterni
Il cariotipo che ne deriva può essere:
69, XXX
69, XXY
69, XYY
La causa della formazione di questi cariotipi aberranti è dovuta ad un difetto di fertilizzazione,
come ad esempio la formazione dello zigote a partire da ovuli aploidi e spermatozoi diploidi. In
caso di concepimento di un feto con sindrome da triploidia la gravidanza è complicata da fenomeni
emorragici, gestosi, polidramnios o oligoamnios. I casi che giungono alla nascita mostrano basso
peso, prematurità, oltre che una pletora di dismorfismi tra cui asimmetrie cranio-facciali, difetti di
ossificazione cranica, microftalmia, sindattilia del terzo e quarto dito.
Sindrome di Smith-Magenis: sindrome genetica che si manifesta con anomalie comportamentali e
disordini dello sviluppo. La SMS è causata da una microdelezione a livello del braccio corto del
cromosoma 17 (17p11.2) con conseguente aploinsufficienza del prodotto genico di RAI1; mutazioni
del gene inattivi possono produrre lo stesso effetto della microdelezione. La SMS deve essere
dunque considerato con una sindrome clinica dovuta ad un disordine monogenico come la sindrome
di Angelman.
Che cosa succede quando il cariotipo non ce la fa?
Può accadere che non si riesca a trovare le anomalie con l’esame del cariotipo: le anomalie
cromosomiche possono essere evidenziate dal cariotipo standard solo se interessano porzioni molto
ampie del cromosoma delezioni, duplicazioni al di sotto delle 3-5 Megabasi possono non essere
evidenziate con il normale esame del cariotipo.
36
Dobbiamo studiare porzioni più piccole del DNA
citogenetica molecolare: mette insieme le metodiche standard di citogenetica assieme alle
metodiche di biologia molecolare per studiare porzioni di cromosoma. Permette un’analisi mirata di
una regione cromosomica, consentendo così di mettere in evidenza riarrangiamenti di alcune
centinaia di kilobasi (es: FISH, PRINS, PCR in situ, CGH).
01/12/2011
Quando non riusciamo a determinare la presenza di un’anomalia cromosomica in un bimbo con un
ritardo mentale sindromico, possiamo andare un po’ oltre tramite la metodica della citogenetica
molecolare per andare a mettere in evidenza riarrangiamenti di alcune centinaia di kilobasi.
Metodica FISH (fluorescence in situ hybridization): se abbiamo un probe, questo può essere
marcato con molecole fluorescenti: denaturo i cromosomi in metafase, la sonda può attaccarsi alla
sequenza complementare ed essere visto tramite microscopio a fluorescenza.
tecnica di ibridazione che permette, dopo fissazione di metafasi e nuclei in interfase su vetrino,
di identificare sequenze specifiche negli acidi nucleici.
Tale identificazione avviene mediante sonde marcate in maniera non isotopica, impiegando
fluorocromi che emettono a diverse lunghezze d’onda.
marcatura della
sonda
denaturazione della
sonda
nucleotidi biotinilati
sonda biotinilata
ibridazione
sonda ibridata al DNA
cromosomico
cromosomi denaturati
visualizzazione al
microscopio
avidina fluorescinata
cromosomi con sonda fluorescinata
37
cromosomi con segnali fluorescenti in
corrispondenza del segmento di DNA
riconosciuto dalla sonda
L’assenza del segnale determina l’assenza della regione regione è deleta.
Si utilizzano diverse tipologie di sonde: posso utilizzarle per studiare la cromatina pericentromerica
e il centromero, i telomeri o regioni specifiche di eucromatina.
Tipi di sonde:
1) sonde di sequenze ripetute:
alfa satellite (sequenza caratteristica del centromero)
beta satellite
satelliti classici
telomeriche
2) sonde di sequenze uniche, specifiche del genoma:
DiGeroge
Prader-Willi
Williams
etc…
3) probe painting (sonde regione-specifiche per lo stesso cromosoma che legandosi tutte insieme
danno l’impressione di averlo colorato completamente)
ABERRAZIONI
TIPI DI SONDE
Sequenze ripetute
CROMOSOMICHE
IDENTIFICABILI
- Trisomie
- Monosomie
MATERIALE
IMPIEGATO
Nuclei in interfase
- Riarrangiamenti
Painting
cromosomici
- Identificazione di
Metafasi
cromosomi marcatori
- Microdelezioni e
Sequenze uniche
duplicazioni
- Riarrangiamenti
Metafasi e
nuclei in interfase
cromosomici
A seconda del probe che uso, posso studiare alcune situazioni in particolare
esempio: le sequenze ripetute posso utilizzarle per andare ad analizzare trisomia e monosomie,
cioè vado a vedere quanti centromero ci sono di determinati cromosomi (alfa satelliti); il painting lo
uso soprattutto per riarrangiamenti cromosomici o per identificare cromosomi marcatori; le
sequenze uniche sono utilizzate per lo studio di microdelezioni e riarrangiamenti cromosomici, ma
qua devo avere una indicazione clinica specifica.
38
Chromosome painting
Mette in evidenza riarrangiamenti: vedo dove è traslocato un pezzo di cromosoma.
(serve soprattutto per valutare se il figlio di un genitore con traslocazione bilanciata ha esattamente
lo stesso quadro genotipo fenotipicamente silente o se è più complicato).
Principali sindromi da microdelezioni
LOCALIZZAZIONE
CROMOSOMICA
SOGGETTI CON
MICRODELEZIONE
Prader Willi/Angelman
15q11.13
70%
Williams
7q11.23
90%
DiGeorge/Velocardiofacciale
22q11.2
75%
Smith-Magenis
17p11.2
95%
Miller-Dieker
17p13.3
90%
SINDROME
DIAGNOSI DI ANEUPLOIDIE
39
Con la FISH, utilizzando un probe che riconosce il centromero del cromosoma Y, un probe che
riconosce una regione distale specifica del cromosoma 21 e un probe che riconosce il centromero
del cromosoma X: in questo caso, si tratta di una femmina con trisomia 21
ho la risposta in 24 ore
Sindrome di Williams affetti hanno una facies caratteristica, presentano:
labbra carnose
iride stellata
problemi cardiaci
problemi psicologici
sono molto socievoli
Presentano una delezione ricorrente di circa 180 kilobasi sul braccio lungo del cromosoma 7: si può
utilizzare anche qui un probe specifico per vedere se quel tratto è presente o è deleto.
Si utilizzano due probe:
quello rosso specifico per la regione della sindrome
quello azzurro più distale: è un controllo interno, perché se utilizzassi solo il probe della
sindrome di Williams e non vedessi il segnale, potrebbe anche essere un errore tecnico.
Utilizzando il controllo interno mi assicuro che l’esperimento sia veramente riuscito.
40
Si vede un cromosoma 7 senza il segnale della sonda per la regione responsabile della sindrome di
Williams, perciò il bambino è affetto.
Nella diagnosi di Prader-Willi si utilizzano tre probe: in questo caso si vedono tutti e tre i segnali,
quindi non c’è stata delezione. Se si è ancora convinti che il bimbo sia affetto, si va a ricercare la
disomia uniparentale materna.
I vantaggi della FISH sono molti:
rapidità
identificazione di microdelezione e riarrangiamenti complessi
diagnosi su nucleo
Il problema con la FISH, a parte i costi, è che la diagnosi non è completa: con la FISH io studio solo
una regione del genoma, non tutto il DNA in generale, quindi devo avere un sospetto diagnostico.
Di recente si è fatto un ulteriore passo avanti
noi sappiamo che la parte distale dei nostri cromosomi ha sequenze comuni, famiglie complesse
e sequenze uniche.
Le regioni subtelomeriche delle sequenze uniche
sono veramente ricche di isole CpG e di geni,
tant’è che si pensa abbiano la più alta densità
genica dell’intero genoma.
Quindi si pensa che anomalie cromosomiche
coinvolgenti queste aree possano essere
associate ad anomalie fenotipiche e ritardo mentale.
È stato recentemente dimostrato che il ritardo mentale
può essere dovuto a riarrangiamenti cromosomici
subtelomerici non evidenziabili con le classiche
metodiche di citogenetica, ma solo con la
citogenetica molecolare.
41
Perciò, quando non trovo anomalie riconducibili a sindromi come la Prader-Willi o altre con la
FISH in un bambino con ritardo mentale, passo a studiare le regioni subtelomeriche delle sequenze
uniche.
Visualizzazione delle sequenze subtelomeriche sul cromosoma 1
Non si riscontrano problemi.
Visualizzazione delezione subtelomerica 2q
Manca il segnale rosso sul secondo cromosoma: c’è un riarrangiamento subtelomerico della
porzione terminale del braccio lungo del cromosoma 2.
Più di 500 malattie genetiche sono associate a ritardo mentale:
Cromosomiche (Sindrome di Down, ecc.)
Monofattoriali (S. FraX, errori congeniti del metabolismo, ecc.)
Multifattoriali (craniostenosi, ecc.)
Riarrangiamenti criptici subtelomerici, si possono considerare alla base di circa il 7.4%
dei casi di ritardo mentale idiopatico da moderato a severo e dello 0.5% dei casi di
ritardo mentale lieve. In questi casi, si tratta quasi sempre di un ritardo mentale da
42
moderato a severo poiché, essendo regioni ricche in geni, molti geni vengono persi e
quindi si ha un fenotipo più complesso rispetto a quello di un ritardo mentale lieve.
Ancora oggi tuttavia, molti bambini con ritardo mentale rimangono senza una diagnosi precisa:
l’approccio alla citogenetica moderna è cambiato con un lavoro del 2004, in cui per prima cosa si è
parlato di array-CGH (comparative genomic hybridization)
si vanno a ricercare riarrangiamenti piccoli del genoma senza nessuna indicazione, studiando in
una sola volta tutto il genoma.
In questa tecnica, si prende il DNA di controllo e lo si marca con un fluorocromo, poi si prende il
DNA del paziente e lo si marca con un fluorocromo diverso: i due DNA vengono poi mischiati
assieme. La miscela di DNA viene ibridata su array (vetrino) dove ci sono molti spot, ciascuno dei
quali contiene una sequenza di circa 70 paia di basi del genoma (ci sono vetrini da 44.000 spot, da
105.000, da 200.000, etc.): questi frammenti di DNA spaziano su tutto il genoma.
Così, andando a calcolare per tutti questi spot il rapporto tra le due fluorescenze, avrò che se io ho
una delezione nel paziente ci sarà un segnale più basso rispetto alla media, mentre se nel paziente
c’è una duplicazione otterrò un segnale più alto.
In pratica il genoma del paziente e un genoma di controllo vengono marcati con fluorocromi diversi
e ibridati contemporaneamente su un vetrino, sul quale sono adese una serie di sonde
oligonucleotidiche che coprono tutto il genoma. DNA di campione e controllo competono per le
sonde e, valutando la fluorescenza, è possibile vedere se c’è un’abbondanza di uno rispetto all’altro,
cioè se nel paziente c’è una delezione o una duplicazione. In particolare per ogni sonda viene
valutato il logaritmo in base 2 del rapporto di fluorescenza tra Cy5(paziente) e Cy3(controllo). Se
questo è 0 la regione è normale, cioè ha un numero di copie uguali nei due genomi. Se è diverso da
0 nella regione è presente un’alterazione; in particolare nella figura vediamo che il logaritmo è circa
-1, indicando così la presenza di una delezione.
Studiando con l’array-CGH paziente con dismorfismi, ritardi mentale e cariotipo normale troviamo
quasi un 20% di bimbi con alterazioni.
Vantaggi dell’array-CGH:
Indipendenza da cellule in divisione
Capacità di analizzare l’intero genoma in un esperimento
Elevata specificità, grande sensibilità e alta risoluzione
Brevi tempi di analisi
Svantaggi:
Incapacità di rilevare riarrangiamenti bilanciati e poliploidie
Limitata abilità di individuare mosaicismi
43
Si possono vedere alterazioni strutturali (delezioni, inversioni, duplicazioni, traslocazioni), da
alcune centinaia di basi a diverse megabasi.
Esempio:
signore ha la moglie in gravidanza e va in consulenza per chiedere quale probabilità ha il figlio di
avere un ritardo mentale, dato che due suoi fratelli e la madre erano affetti da questo disturbo
Si visitano la sorella e il fratello: con l’array-CGH si è trovata una duplicazione sul braccio corto
del cromosoma X, ritrovata poi in altre donne con ritardo mentale.
Queste erano persone adulte rimaste senza diagnosi per anni: trovata nuova alterazione causa di
ritardo mentale con caratteristiche X-linked dominante.
Esempio:
due sorelle gemelle presentano un fenotipo simile (lieve ritardo mentale, cardiomiopatia dilatativa):
all’array-CGH è stata trovata una delezione di circa 6 megabasi sul cromosoma 9.
La stessa delezione è stata riscontrata in un’altra ragazza con le stesse caratteristiche, delineando
così una nuova sindrome da delezione del 9q.
44
MALATTIE MULTIFATTORIALI
I caratteri “quantitativi” o “continui” riguardano più individui e si possono misurare nella
popolazione, ottenendone una distribuzione a curva di Gauss, come avviene per esempio per la
misurazione della pressione arteriosa, dell’altezza, etc.
Certi caratteri hanno l’ “effetto soglia”: situazioni su base multifattoriale determina una
suscettibilità (sull’asse delle x nel grafico), specie per gli individui che si trovano agli estremi della
curva ad un’estremità ci saranno individui con una suscettibilità molto bassa (sono protetti),
mentre dall’altra ci saranno gli individui affetti.
Se si considerano i parenti di primo grado degli individui malati, la curva sarà traslata verso destra
rispetto alla popolazione generale, perché appartenendo alla stessa famiglia condividono molti
fattori di suscettibilità e così ci sarà un numero più alto di individui aldilà della soglia
Questo è il rischio di ricorrenza dei parenti di primo grado di soggetti ammalati, che è più alto
rispetto alla popolazione generale.
IMPORTANZA DI FATTORI GENETICI NELLE PATOLOGIE MULTIFATTORIALI
E’ necessario capire quanto di ereditario c’è in una malattia complessa: innanzitutto si stabilisce il
rischio relativo, che è il rischio di un fratello o di una sorella di un individuo affetto rispetto al
rischio della popolazione generale:
= rischio nel fratello di un individuo malato
rischio nella popolazione generale
45
Gli studi classici erano quelli fatti sui gemelli monozigotici e dizigotici: si andava a vedere se i
gemelli monozigoti, che hanno il 100% di DNA in comune, rispetto agli zigotici che sono come
fratelli (50% di DNA in comune), condividevano molto di più una malattia rispetto ai dizigotici.
Se c’è concordanza tra i monozigoti e discordanza tra i dizigoti, significa che quel carattere ha una
componente genetica molto forte; se c’è discordanza in entrambi i casi, significa che quella malattia
ha una forte componente ambientale.
Si sono fatti studi anche su individui adottati: due fratelli adottati da famiglie diverse venivano
esaminati (se per esempio uno dei due sviluppava una patologia e l’altro no, significava che c’era
una forte componente ambientale).
Gli studi sui gemelli hanno consentito di capire che c’è una forte componente genetica nell’autismo,
che si riteneva avesse invece una forte componente ambientale
Il rischio nei fratelli è di circa il 3%,
da 10 a 50 volte più alto che nella
popolazione generale.
Oggi si fanno studi più specifici per poter determinare quali sono le regioni di suscettibilità di
queste malattie: analisi di linkage, studio di coppie di fratelli affetti che condividono una certa
malattia hanno il 50% di DNA in comune: posso identificare quale parte di genoma condividono
e analizzare in questa porzione la regione condivisa sempre da tutte le coppie di fratelli affetti
studiati e così capire qual è la regione che è sempre presente in tutti i soggetti e che sarà la regione
dove si trova il gene di suscettibilità.
N.B.: nello studio di malattie multifattoriali complesse si usano metodi diverse dal classico linkage
Si fanno anche studi di controllo tra una popolazione affetta dalla malattia e una popolazione di
controllo che presenta caratteristiche in comune (sesso, età, etc) ma che non presenta la malattia: si
cercano variazioni di frequenza che si trovano più frequentemente nella popolazione con la malattia
rispetto a quella di controllo. Non si fa altro che comparare la sequenza allelica di polimorfismi di
due gruppi, uno di pazienti e uno di controllo.
46
È molto importante anche valutare se lo SNP preso in considerazione ha anche un valore
funzionale, cioè se può essere collegato ad una funzione di quel determinato gene
esempio: trovo uno SNP nella regione promotrice di un gene. Facendo studi funzionali trovo che
quella variazione determina una minore trascrizione di quel gene e quindi una minore produzione
della proteina. Questo è uno SNP funzionale, cioè uno SNP cui è legato un effetto a livello della
proteina. Sono varianti che di per sé non danno una patologia.
L’analisi di linkage non parametrici (studio di coppie di fratelli) stabiliscono se c’è una discrepanza
tra quello che ci si attende che due fratelli condividano normalmente e quello che noi vediamo,
quindi si basano sulla condivisione di alleli di un marcatore da parte di individui affetti all’interno
della stessa famiglia.
In una qualsiasi regione del genoma, due fratelli condividono due alleli nel 25% dei casi, un allele
nel 50% e nel 25% dei casi nessun allele in comune: questo è ciò che capita normalmente.
Questo è ciò che ci aspettiamo di vedere, tranne che in una regione, dove probabilmente risiede il
gene di suscettibilità per quella malattia: in questo punto è molto più frequente che i due fratelli
condividano lo stesso allele o entrambi gli alleli.
Le proporzioni 25%-50%-25% sono mantenute in tutto il genoma, tranne che in quella regione dove
si trova il gene di suscettibilità.
Qui si entra nel concetto del LINKAGE DISEQUILIBRIUM: il linkage classico stabilisce una
concatenazione tra un allele di un determinato polimorfismo e un locus-malattia all’interno di una
famiglia (segregano insieme). Il linkage disequilibrium è una associazione allelica non casuale
all’interno di una popolazione: per esempio, nella spondilite anchilosante il 98% dei pazienti ha
l’allele HLA-B27, il che non significa che tutti coloro che hanno l’allele HLA-B27 sviluppano la
malattia, ma quasi la totalità dei pazienti ha quell’allele, quindi c’è un’associazione allelica non
casuale tra quel gruppo di pazienti e quell’allele.
Posso studiare il linkage disequilibrium tramite studi di associazione o tramite il Trasmission
Disequilibrium Test (TDT): secondo le leggi di Mendel, ci si aspetta che un allele segreghi fino al
50%. Posso studiare se un allele è trasmesso dai genitori al figlio affetto più frequentemente rispetto
ai controlli (> 50%).
47
a/b
c/d
a/d
a/c
a/c
b/d
b/b
a/d
c/d
b/c
c/d
a/d
c/b
b/a
c/d
a/d
c/d
c/a
a/b
c/c
a/d
a/b
a/c
c/d
b/c
a/d
a/b
c/d
a/d
a/a
Qui abbiamo tutti individui sani: l’allele “a” è’ stato trasmesso 6 volte su 12, con una probabilità del
50%, quindi è nella norma.
Se invece andiamo a prendere una serie di soggetti i cui figli sono ammalati, vediamo che in questo
caso la trasmissione avviene 10 volte su 12, creando un divario significativo tra quello che ci si
attende per caso tra il 50% che mi aspetto e l’83% che osservo.
a/
b
c/
d
a/
d
a/
c
a/
c
b/
d
b/
b
a/
d
a/
c
a/
c
Phenocopy
a/
c
c/
b
b/
a
c/
d
c/
d
a/
d
a/
d
a/
a
c/
a
a/
d
a/
a
c/
a
c/
b
a/
c
a/
d
c/
a
c/
d
a/
b
a/
d
a/
a
Se questo allele è associato alla patologia, significa che in qualche modo ogni volta che ho un
paziente ritrovo più frequentemente la presenza di quell’allele.
48
K1
J1
I1
H1 Mutazione
G1 N -> D
N
F1
E1
D1
C1
B1
A1
K1
J1
I1
H1
10
G1 generaz.
D
F1
E1
D1
C1
B1
A1
K2
J2
I2
H1
90
G1 generaz.
D
F1
E1
D1
C2
B2
A2
K1
J1
I1
H1
G1
D
F1
E1
D1
C1
B1
A1
Abbiamo un cromosoma iniziale, su cui abbiamo molti loci: a un certo punto il locus N è mutato in
D. Via via che si va avanti nella popolazione, si hanno ricombinazioni per cui a un certo punto il
cromosoma iniziale si riduce a un frammento talmente piccolo che difficilmente ricombinerà
ulteriormente: questo è detto BLOCCO IN DISEQUILIBRIUM quel tratto di DNA è sempre
lo stesso, G1-D-F1, e segrega sempre all’interno del gruppo di pazienti affetti. Bisogna poi capire
all’interno di quel pezzo qual è la variante che da la suscettibilità.
Il linkage disequilibrium diminuisce con le generazioni e le frazioni di ricombinazione.
In realtà il nostro genoma si eredita in blocchi, perché alcune parti sono talmente piccole che sono
sempre in disequilibrium e vengono sempre trasmesse dal genitore al figlio.
Concetto importante possiamo avere un’associazione con uno SNP, ma in realtà bisogna tenere
in considerazione non il singolo SNP, ma il blocco in disequilibrium con quella malattia, perché in
realtà lo SNP funzionale che determina la suscettibilità non è quello identificato, ma un altro
presente nello stesso blocco.
49