Risultati
4.2. Quantificazione dei livelli di espressione genica di
MYCN in linee di cancro colorettale
Per quantificare l’espressione genica dell’oncogene MYCN nelle linee
cellulari di cancro colorettale è stata utilizzata l’analisi quantitativa PCR RealTime (qPCR) al fine di confrontare i dati ottenuti tramite FISH. L’analisi è
stata realizzata normalizzando la media dei Ct di MYCN sui valori di Ct del
gene housekeeping, la β-actina, utilizzato come controllo endogeno perché
normalmente espresso in maniera quasi costante in tutte le cellule
dell’organismo.
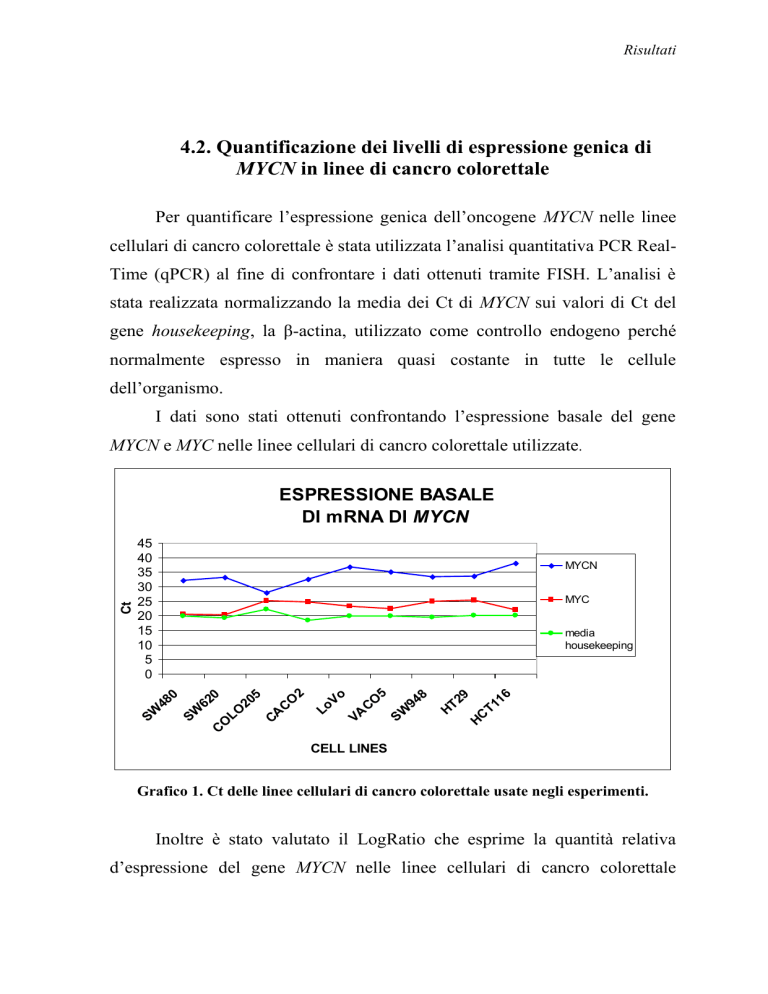
I dati sono stati ottenuti confrontando l’espressione basale del gene
MYCN e MYC nelle linee cellulari di cancro colorettale utilizzate.
Ct
ESPRESSIONE BASALE
DI mRNA DI MYCN
45
40
35
30
25
20
15
10
5
0
MYCN
MYC
media
housekeeping
SW
0
48
SW
0
62
CO
LO
5
20
CA
CO
2
Vo
Lo
VA
CO
5
SW
8
94
16
29
T1
HT
HC
CELL LINES
Grafico 1. Ct delle linee cellulari di cancro colorettale usate negli esperimenti.
Inoltre è stato valutato il LogRatio che esprime la quantità relativa
d’espressione del gene MYCN nelle linee cellulari di cancro colorettale
Risultati
relativamente alla linea cellulare di riferimento H526, linea di Small Cell
Lung Cancer che sovraesprime MYCN (Ct=24).
Log Ratio
Espressione MYCN
0
-2
-4
-6
-8
-10
-12
-14
-16
CO
5
20
LO
0
48
SW
29
HT
8
94
SW
0
62
SW
2
CO
CA
Vo
Lo
5
CO
VA
16
T1
HC
Cell lines
Grafico 2. Espressione basale di mRNA di MYCN nelle linee cellulari di CRC.
Analogamente a quanto fatto per l’oncogene MYCN, si è valutata
l’espressione dell’oncogene MYC relativamente alla linea di riferimento H82,
linea di Small Cell Lung Cancer, che sovraesprime MYC (Ct=18,1) (Grafico
3).
Log Ratio
Espressione MYC
0
-2
-4
-6
-8
-10
-12
-14
-16
-18
CA
CO
2
SW
94
8
HT
29
Lo
Vo
CO
LO
20
5
VA
CO
5
HC
T1
16
SW
62
0
SW
48
0
Cell lines
Grafico 3. Espressione basale di mRNA di MYC nelle linee cellulari di CRC.
4.3. Western Blot: valutazione dell’espressione
dell’oncoproteina N-Myc
Per verificare l’effettiva corrispondenza tra livelli genici e proteici
relativi all’oncogene MYCN si è valutata, mediante Western Blot,
Risultati
l’espressione basale della proteina N-Myc nelle diverse linee cellulari di
cancro colorettale utilizzate (fig. 9-10).
Verificata l’espressione di tale proteina nelle linee cellulari, si è
una seconda proteina, la β-actina, in quanto quest’ultima viene
valutata
costitutivamente espressa nelle cellule per cui rappresenta un utile mezzo di
controllo al fine di esaminare che sia stata caricata un uguale quantità di
proteine sul gel (fig. 9-10).
SW480
SW620
SW948
LoVo
65 kDa
N-Myc
50 kDa
β-actina
Fig. 9 Visualizzazione dell’oncoproteina N-Myc e della
proteina β-actina nelle linee di CRC
VACO5 COLO205 CACO2 HT29 HCT116
65 kDa
N-Myc
50 kDa
β-actina
Fig. 10 Visualizzazione dell’oncoproteina N-Myc e della
proteina β-actina nelle linee di CRC
4.4. Trattamento con PNA anti MYCN:
valutazione mediante qPCR
Dopo aver analizzato i livelli basali d’espressione relativi agli oncogeni
MYCN e MYC si è passato al trattamento alle 12 ore con PNA anti-MYCN
Risultati
sulla linea COLO 205 per testarne l’effetto sul trascritto. Come si può notare
in grafico 4, non si riscontra alcuna significativa risposta nè a seguito del
trattamento con PNA mutato nè dopo trattamento di PNA anti-MYCN (i valori
di LogRatio inferiori all’unità sono infatti trascurabili).
LIVELLO DI ESPRESSIONE DI N-MYC E C-MYC DOPO
TRATTAMENTO CON PNA 12 ore
0,8
LOG RATIO
0,6
0,4
0,2
N MYC
C MYC
0
-0,2
-0,4
-0,6
PNA
PNA mutato
Grafico 4. Livello di trascritto degli oncogeni MYCN e MYC nella linea
COLO205 alle 12 ore dopo trattamento con PNA.
4.5. Trattamento con siRNA anti-MYCN e anti-MYC:
valutazione mediante qPCR
Dopo aver valutato gli effetti del trattamento con il PNA, si è passati al
trattamento con siRNA anti-MYCN e anti-MYC sulla linea COLO 205 per
testarne l’effetto sul trascritto. Come si può notare nei grafici 5 e 6, vi è una
significativa inibizione per MYCN e MYC specifica in relazione al tipo di
siRNA utilizzato.
Il trattamento con il siRNA anti-MYCN è stato condotto a diverse
concentrazione e a diversi tempi (24 e 48 ore). Di seguito sono riportati i
risultati:
Risultati
LIVELLI DI EPRESSIONE DI MYCN
DOPO TRATTAMENTO CON siRNA anti-MYCN
0
LOG RATIO
-0,5
-1
24h
-1,5
48h
-2
-2,5
-3
siRNA NMYC
100nM
siRNA NMYC
250nM
siRNA NMYC
500nM
siRNA NMYC
1μM
scramble
Grafico 5. Analisi dell’attività dei siRNA anti-MYCN in funzione delle dosi e dei tempi
di trattamento.
Le concentrazioni 100nM, 250nM, 500nM e 1μM evidenziano una
significativa risposta in termini di calo del trascritto; quella che meglio di tutte
esplica l’effetto inibente è la concentrazione 1μM, con un buon effetto già alle
24 ore e un incremento dell’azione alle 48 ore (LogRatio rispettivamente di 1,8 e -2,4).
Analogamente per i siRNA anti-MYC sono state saggiate varie
concentrazioni alle 24 e 48 ore.
LIVELLI DI EPRESSIONE DI MYC
DOPO TRATTAMENTO CON siRNA anti-MYC
0,5
0
LOG RATIO
-0,5
-1
24h
-1,5
48h
-2
-2,5
-3
siRNA CMYC
100nM
siRNA CMYC
250nM
siRNA-C MYC
500nM
siRNA-C MYC
1μM
scramble
Risultati
Anche in questo caso, è stata determinata la concentrazione a maggiore
capacità inibente, che risulta essere la 100nM (con valori di LogRatio pari a 1,1 e -2,5 rispettivamente alle 24 e 48 ore).
Come visibile nei grafici 5 e 6, sono stati usati siRNA a sequenza casuale
(scrambled) come verifica della specifica azione dei siRNA progettati su MYC
e MYCN. Tale specificità è inoltre verificata dal fatto che il trattamento con
siRNA anti-MYCN non porta al silenziamento di MYC e la stessa cosa vale
anche per il siRNA anti-MYC (Grafico 7 e 8).
Grafico 6. Analisi dell’attività dei siRNA per MYC in funzione delle dosi e
dei tempi di trattamento.
LOG RATIO
LIVELLI DI EPRESSIONE DI MYC
DOPO TRATTAMENTO CON siRNA anti-MYCN
0,4
0,2
0
-0,2
-0,4
-0,6
-0,8
-1
-1,2
-1,4
-1,6
-1,8
-2
24h
48h
siRNA NMYC
100nM
siRNA NMYC
250nM
siRNA NMYC
500nM
siRNA NMYC
1μM
scramble
Grafico 7. Analisi dell’effetto su MYC dei siRNA anti-MYCN
LOG RATIO
LIVELLI DI EPRESSIONE DI MYCN
DOPO TRATTAMENTO CON siRNA anti-MYC
0,8
0,6
0,4
0,2
0
-0,2
-0,4
-0,6
-0,8
-1
-1,2
-1,4
-1,6
-1,8
-2
24h
48h
siRNA CMYC
100nM
siRNA CMYC
250nM
siRNA-C MYC
500nM
siRNA-C MYC
1μM
scramble
Grafico 8. Analisi dell’effetto su MYCN dei siRNA anti-MYC
Risultati
Il trattamento con i siRNA è stato effettuato anche sulle linee cellulari
SW480 e SW620 alle 24 ore a diverse concentrazioni con i seguenti risultati:
Grafico 9. Analisi dell’attività dei siRNA anti-MYCN in funzione della
dose sulla linea SW480
Grafico 10. Analisi dell’attività dei siRNA anti-MYC in funzione della
dose sulla linea SW480
Le concentrazioni che mostrano efficacia sulla linea cellulare SW480
sono sono la 100nM per il siRNA anti-MYCN, mentre per il siRNA anti-MYC
Risultati
la concentrazione inibente è la 500nM (con un LogRatio rispettivamente di 2,5 e-1,5).
Per la linea SW620, invece, le concentrazioni efficaci sono la 100nM
per il siRNA anti-MYCN e le 250 e 500 nM per il siRNA anti-MYC (con un
LogRatio rispettivamente di -1,3, -1,2 e -1,5)
Grafico 11. Analisi dell’attività dei siRNA anti-MYCN in funzione della dose
sulla linea SW620
Grafico 12. Analisi dell’attività dei siRNA anti-MYC in funzione della
dose sulla linea SW620
Risultati
4.6. Effetto del trattamento con siRNA anti MYCN e antiMYC sulla proliferazione cellulare
Le cellule sono state piastrate (circa 15000 per pozzetto) e sono state
trattate con siRNA anti-MYCN ed anti-MYC a diverse concentrazioni.
Ad una prima analisi delle cellule al microscopio ottico è già visibile
l’effetto del trattamento; si può infatti notare una ridotta densità cellulare nel
caso dei trattati se confrontati con le cellule di controllo (non trattate), che
tendono a formare aggregati con presenza di cellule morte (Fig.11-12).
Fig 11. A.Cellule di controllo alle 24 ore. B.Cellule di controllo alle 48 ore
Fig 12.A. Cellule trattate alle 24 ore. B.Cellule trattate alle 48 ore
Risultati
Per valutare l’effettiva vitalità cellulare è stato fatto il saggio ATPlite e,
come evidente dal grafico 13, per le concentrazioni efficaci l’effetto del
trattamento è già visibile alle 24 ore e raggiunge un picco di intensità alle 48
ore, per poi iniziare a decadere.
24h
Effetto siRNA su COLO205
72h
10
00
YC
50
0
si
M
YC
25
0
si
M
YC
10
0
si
M
si
M
N
YC
YC
10
00
50
0
N
si
M
si
M
YC
N
10
0
YC
N
si
M
YC
si
M
AM
SC
R
25
0
100
80
60
40
20
0
BL
E
% proliferazione
48h
siRNA
Grafico 13. Percentuale di proliferazione nei tre giorni dopo trattamento
con siRNA anti-MYCN e anti-MYC a diverse concentrazioni
La percentuale d’inibizione maggiore per i siRNA anti-MYCN è quella
osservata alla concentrazione di 1μM che risulta del 26% alle 24 ore per
arrivare al 55% alle 48 ore e calare al 39,5% alle 72 ore. Tuttavia sono
particolarmente significativi anche i dati osservati per le concentrazioni
100nM, 250nM e 500nM che raggiungono rispettivamente il picco del 46%
38%, 52,5% di inibizione alle 48 ore.
Risultati
Curva di crescita
280000
240000
CTRL
siMYCN 100
siMYCN 1000
siMYCN 250
siMYCN 500
SCRAMBLE
200000
Cps
160000
120000
80000
40000
0
24h
48h
72h
Tempo (h)
Grafico 14. Curva di crescita nei tre giorni dopo trattamento con
siRNA anti-MYCN a diverse concentrazioni
Per ciò che concerne i siRNA anti-MYC l’unica concentrazione efficace
risulta essere la 100nM che porta ad un calo della proliferazione del 31% alle
prime 24 ore fino al 49% alle 48 ore.
Curva di crescita
280000
240000
Cps
200000
CTRL
siMYC 100
siMYC 1000
siMYC 250
siMYC 500
160000
120000
80000
40000
0
24h
48h
72h
Tempo (h)
Grafico 15. Curva di crescita nei tre giorni dopo trattamento con
siRNA anti-MYC a diverse concentrazioni
Risultati
4.7. Il silenziamento causa un differente profilo
d’espressione genica
Effettuato il trattamento sulle cellule COLO 205 con gli siRNA antiMYCN alle diverse concentrazioni e osservato il decremento a livello
trascrizionale di MYCN, sono stati analizzati alle 24 e 48 ore dopo il
trattamento i profili di espressione di alcuni geni importanti nella regolazione
del processo apoptotico.
I geni presi in considerazione sono stati DR5, TRAIL, TNF, TNFAIP3,
DDIT3, GADD45A e SURVIVINA con i seguenti risultati:
.
24 ORE
siRNA NMYC 100nM
siRNA NMYC 250nM
siRNA NMYC 500nM
siRNA NMYC 1μM
3
LOG RATIO
2
1
0
RV
IV
IN
A
FA
IP
3
TR
AI
L
TN
TN
F
5
DR
IT
3
DD
45
A
K3
DK
AD
D
G
SU
BE
TA
CA
TE
N
-2
IN
A
CI
CL
IN
A
D
-1
GENES
Grafico 16. Effetto del trattamento con siRNA anti-MYCN alle 24 ore sulla linea
cellulare COLO 205.
Risultati
48 ORE
siRNA NMYC 100nM
siRNA NMYC 250nM
siRNA NMYC 500nM
siRNA NMYC 1μM
LOG RATIO
5
3
1
-1
A
IN
IV
RV
L
AI
SU
TR
3
IP
FA
5
F
TN
TN
DR
A
45
3
IT
D
AD
DD
G
K3
DK
D
A
A
IN
IN
CL
N
CI
TE
CA
-5
TA
BE
-3
GENES
Grafico 17. Effetto del trattamento con siRNA anti-MYCN alle 48 ore sulla linea
cellulare COLO 205.
.
Alle 24 ore, l’unico gene che mostra una significativa risposta è DR5 che
presenta un aumento di espressione superiore alle 2 unità di LogRatio alla
concentrazione di siRNA 1μM con una variazione superiore alle 48 ore
(LogRatio pari a 3,3).
Alle 48 ore anche gli altri geni in analisi modificano in modo
significativo la loro espressione.
TRAIL mostra una variazione pari a 3,2 unità di LogRatio per i siRNA a
concentrazione 100nM e 1μM.
Il gene GADD45A mostra un aumento del livello di espressione pari a
2,3 unità per la concentrazione di siRNA 1μM.
Il gene DDIT3 (DNA-damage-inducible transcript 3) mostra alle 48 ore
un aumento importante che nel caso del siRNA alla concentrazione 1μM è
pari a 3,3 unità di LogRatio.
Il gene TNF e TNFAIP3 mostrano rispettivamente un incremento e una
riduzione nell’espressione dopo il trattamento con un valore di LogRatio
rispettivamente di 1,7 e -1,3 alla concentrazione di siRNA 1μM.
Risultati
Infine la survivina evidenzia un’importante diminuzione del livello di
trascritto (con un LogRatio di circa -3 unità per tutte le concentrazioni di
siRNA usate).
Sono stati inoltre studiati geni che codificano per proteine agenti a
diverso livello nel pathway WNT: il gene codificante DKK3, il gene per la
beta-catenina e per la ciclina D. La beta-catenina e la ciclina D, mostrano
alle 48 ore un’evidente riduzione del trascritto (rispettivamente di -3,3 e -3,8
unità di LogRatio per la concentrazione 1μM); l’antagonista del pathway
WNT, DKK3 (Dickkopf homolog 3), mostra invece in risposta al trattamento
un incremento di espressione (1,8 unità di LogRatio per la concentrazione
1μM).
I dati osservati sono specifici per il trattamento con siRNA anti-MYCN;
infatti l’utilizzo del siRNA scrambled non determina alcuna risposta
significativa sui geni analizzati (dati non riportati in grafico).
Conclusioni
CONCLUSIONI
Il cancro colorettale rappresenta i due terzi di tutti i tumori maligni
gastrointestinali.
A monte del processo di tumorigenesi c’è la deregolazione del pathway
WNT. Da studi sul neuroblastoma si è visto che tale pathway è collegato con
l’oncogene MYCN mediante l’antagonista DKK3. Si può pensare ad un
analogo collegamento anche nel CRC e ipotizzare che la sovraespressione di
MYCN agisca in questo tumore, in modo sinergico con la ricorrente
inattivazione di APC, nel deregolare il pathway WNT.
Studi condotti su campioni di colon provenienti da pazienti hanno
mostrato che è presente un’amplificazione di MYCN significativamente più
comune per frequenza ed intensità nel tessuto tumorale piuttosto che in quello
normale ed è inoltre stato osservato che tutti gli adenocarcinomi hanno più alti
livelli di proteine Myc rispetto alla normale mucosa del colon. Alcune delle
linee usate in questo studio hanno mostrato un’amplificazione e una relativa
sovraespressione di MYCN il che le rende un buon modello rappresentativo di
quella percentuale di campioni provenienti da pazienti in cui è stata trovata
un’amplificazione e una sovraepressione dell’oncogene stesso.
In questo lavoro si è focalizzata l’attenzione sulla valutazione e
validazione del ruolo degli oncogeni MYC come potenziali bersagli
farmacologici per il trattamento del cancro colorettale.
Dopo un’iniziale caratterizzazione delle diverse linee cellulari da un
punto di vista citogenetico, trascrizionale e proteico si è voluto valutare
l’effetto derivante dall’inibizione degli oncogeni di interesse.
Lo studio si è inizialmente avvalso dell’utilizzo di una molecola
inibitrice, il PNA anti-gene anti-MYCN: la linea COLO 205 è stata sottoposta
al trattamento per valutarne gli effetti a livello trascrizionale, tuttavia non si è
Conclusioni
osservata alcuna risposta. Probabilmente le cellule in questione sono molto
resistenti e l'ingresso del PNA è risultato di conseguenza difficoltoso; per
questo motivo per saggiare gli effetti di un’inibizione su MYCN si è passati al
trattamento con gli siRNA.
Le cellule COLO 205 sono state trattate con siRNA anti-MYC e antiMYCN a diverse concentrazioni. È stato valutato l’effetto del trattamento sulla
proliferazione e avendo osservato che per le concentrazioni efficaci c’è una
riduzione della capacità inibitoria dopo le 48 ore, si è studiata la risposta in
termini trascrizionali al trattamento alle 24 e 48 ore, con risultati compatibili
con quanto osservato per la crescita cellulare. L’azione degli siRNA è stata
inoltre valutata sulle linee SW-480 e SW-620, tale da ottimizzare la
concentrazione efficace di siRNA su tali cellule.
Si è poi valutato se l’inibizione dell’oncogene MYCN con gli siRNA,
oltre a influenzare la crescita cellulare, abbia avuto effetto su altri aspetti
cellulari e a tal fine si è analizzata, dopo il trattamento, la risposta
trascrizionale di alcuni geni coinvolti nel pathway apoptotico. Da letteratura è
stato visto che uno stress chimico indotto da farmaci provoca una
transattivazione di alcuni dei geni come DR5, GADD45A e DDIT3, con
successivo innesco del pathway apoptotico. I risultati evidenziano che alle 24
ore l’unico gene fra quelli analizzati che subisce una modifica importante in
termini trascrizionali è il gene DR5 con un aumento del segnale alle 24 ore e
un ulteriore incremento ale 48 ore. Il recettore DR5 (Death receptor 5) è uno
dei recettori di TRAIL (tumor necrosis factor-related inducing apoptosis
ligand); quest’ultimo è un membro della famiglia di citochine TNF e
promuove l’apoptosi in modo mitocondrio dipendente o indipendente. Per gli
altri geni si è osservata una variazione in termini di espressione solo alle 48
ore. Lo stesso TRAIL subisce un aumento del trascritto a seguito del
trattamento, dopo 48 ore; analogamente, GADD45A presenta valori di
Conclusioni
LogRatio positivi: i geni GADD45 (Growth Arrest and DNA Damage) sono
GADD45B, GADD45A, GADD45G, sensori di stress che modulano la risposta
a stress di varia natura e il loro livello trascrizionale tende ad aumentare a
seguito di condizioni stressorie o dopo il trattamento con agenti danneggianti
il DNA. DDIT3 è il principale protagonista indotto da stress a livello del
reticolo endoplasmatico e anche in questo caso si osserva un’importante
aumento di espressione alle 48 ore. TNF e TNFAIP3 hanno risposte opposte al
trattamento: il primo mostra un aumento, mentre il secondo una riduzione del
trascritto. TNF è una citochina coinvolta in diversi processi cellulari fra i quali
l’induzione dell’apoptosi a seguito del legame al recettore TNF-R1, con
successiva attivazione a cascata delle caspasi. TNFAIP3 (TNF alpha-induced
protein 3) codifica per una zinc-finger protein responsabile dell’inibizione
trascrizionale del gene NF-kB e dell’apoptosi TNF mediata. La survivina,
infine, membro della famiglia di geni inibitori dell’apoptosi, risponde al
trattamento con un calo dell’mRNA di varie unità. Riassumendo, i geni in
analisi, in diverso modo coinvolti nel processo apoptotico, mostrano
un’importante risposta a seguito del trattamento, evidenziando una risensibilizzazione delle cellule all’apoptosi.
Sono inoltre stati presi in analisi geni relativi al pathway WNT. La βcatenina e la ciclina D mostrano una marcata riduzione a livello trascrizionale
a seguito del trattamento, mentre DKK3 risponde con un incremento. Tali
osservazioni sono compatibili e supportano la possibilità di una connessione,
nel CRC, fra MYCN e il pathway WNT analoga a quella riportata in letteratura
per il neuroblastoma.
La prospettiva futura è anzitutto quella di investigare l’effettiva
motivazione del mancato effetto del PNA sulla linea COLO 205, ad esempio
con l’utilizzo di un PNA-NLS rodaminato valutando il suo uptake da parte
delle cellule mediante microscopio a fluorescenza.
Conclusioni
Data la risposta in termini trascrizionali al trattamento con gli siRNA ci
si propone di andare a verificare l’efficacia anche a livello dell’oncoproteina.
Il prossimo passo sarà inoltre quello di proseguire lo studio sulle linee
tumorali che ancora non sono state valutate o sulle quali il lavoro è stato solo
iniziato (SW-480 e SW-620) e cercare così di avere un quadro il più completo
possibile.
Sarà anche importante una caratterizzazione di MYCN nel CRC in
funzione dell’avanzamento del tumore con uno studio critico del ruolo
dell’oncogene e con una valutazione della sua inibizione in linee cellulari
derivate da pazienti a differente stadio oncologico.
Il ruolo di MYCN come oncogene nel CRC deve essere dunque
ulteriormente esplorato, tuttavia i preliminari risultati ottenuti sono
incoraggianti: l’efficacia inibitoria degli siRNA, con conseguente riduzione
della proliferazione cellulare e variazione nell’espressione di geni coinvolti in
diversi
pathway
cellulari,
rappresentano
buoni
presupposti
per
il
proseguimento degli studi, schiudendo gli orizzonti alla possibilità di sviluppo
di un agente terapeutico basato sull’azione inibente dello siRNA anti-MYCN,
orientato al trattamento di quella percentuale di casi che mostrano
sovraespressione e amplificazione di tale oncogene.
Bibliografia
BIBLIOGRAFIA
Ballinger, A.B. and C. Anggiansah (2007).
Colorectal cancer. BMJ; Volume 335
Fodde, R. (2002)
The APC gene in colorectal cancer. European Journal of Cancer 38; 867-871.
Arnold, N.C., A.Goel et al. (2005)
Molecular pathogenesis of Colorectal Cancer. American Cancer Society; volume 104;
number 10
Lamprecht, S.A., M. Lipkin (2002)
Migrating colonic crypt epithelial cells: primary targets for transformation.
Carcinogenesis vol.23 no.11 pp.1777–1780, 2002
Shtutman, M. et al. (1999)
The cyclin D1 gene is a target of the b-cateninyLEF-1 pathway Cell Biology Vol. 96, pp.
5522–5527.
Bell, E. et al. (2007)
Cell cycle Regulation Targets of MYCN Identified by Gene Expression Microarrays
Cell Cycle; vol.6; Issue 10.
Solomon, D.L., B. Amati, and H. Land (1993).
Distinct DNA binding preferences for the c-Myc/Max and Max/Max dimers. Nucleic Acids
Res 21(23): 5372-6.
Grandori, C. and R.N. Eisenman (1997).
Myc target genes. Trends Biochem Sci, 22(5): p. 177-81.
Leonetti, C., I. D’Agnano, et al. (1996).
Bibliografia
“Antitumor effect of c-myc antisense phosphorothioate oligodeoxynucleotides on human
melanoma cells in vitro and in mice.” J Natl Cancer Inst 88(7): 419-29.
Pession Andrea and Tonelli Roberto (2005).
The MYCN Oncogene as a specific and selective drug target for peripheral and central
nervous system tumors. Current Cancer Drug Targets 5: 273-283.
Tonelli R, Pession A. et al. (2005).
Antigene peptide nucleic acid specifically inhibits MYCN expression in human
neuroblastoma cells leading to cell growth inhibition and apoptosis. Mol. Cancer Ther
4(5).
Pession A, Tonelli R, Fronza R, et al. (2004)
Targeted inhibition of MYCN by peptide nucleic acid (PNA) in N-mycamplified human neuroblastoma cells: cell-cycle inhibition with induction of
neuronal cell differentiation and apoptosis. Int J Oncol;24:265–72
Sakamuro, D., V. Eviner, et al. (1995).
“c-Myc induce apoptosis in epithelial cells by both p53-dependent and p53-independent
mechanisms.” Oncogene 11(11): 2411-8.
Schwab, M., et al. (1984).
Chromosome localization in normal human cells and neuroblastomas of a gene related to
c-myc. Nature, 308(5956): p. 288-91.
Ray, A. and B. Norden (2000).
“Peptide nucleic acid (PNA): its medical and biotechnical applications and promise for
the future.” Faseb J 14(9): 1041-60.
Hanvey, J. C., N. J. Peffer, et al. (1992).
“Antisense and antigene properties of peptide nucleic acids.” Science 258(5087): 1481
Wittung P., Kajanus J. et al. (1995).
Bibliografia
“Phospholipid membrane permeability of peptide nucleic acid.” FEBS Lett 375(3): 27-29.
Yan Wang, Ingo H. Engels, D. A. Knee et al. (2004).
Synthetic lethal targeting of MYC by activation of the DR5 death receptor pathway.Cancer
cell 4, 501-512.
Cutrona, G., E. M. Carpaneto, et al. (2000).
“Effects in live cells of a c-myc anti-gene PNA linked to a nuclear localization signal.”
Nat Biotechnol 18(3): 300-3.
Mologni, L., P. leCoutre, et al. (1998).
“Additive antisense effects of different PNAs on the in vitro translation of the
PML/RARalpha gene.” Nucleic Acids Res 26(8): 1934-8.
Tyler B.M., Jansen K. et al. (1999).
“Peptide nucleic acids targeted to the neurotensin receptor and administered i.p. cross the
blood-brain barrier and specifically reduce gene expression.” Proc Natl Acad Sci USA
96(12): 7053-7058.
Aldrian-Herrada G., Desarmenien M.G. et al. (1998).
“A peptide nucleic acid (PNA) is more rapidly internalized in cultured neurons when
coupled to a retro-inverso delivery peptide. The antisense activity depresses the target
mRNA and protein in magnocellular oxytocin neurons.” Nucleic Acids Res 26(21): 49104916.
Ray, A. and B. Norden (2000).
“Peptide nucleic acid (PNA): its medical and biotechnical applications and promise for
the future.” Faseb J 14(9): 1041-60.
Sei S., Yang Q.E. et al. (2000).
“ Identification of a key target sequence to block human immunodeficiency virus type 1
replication within the gag-pol transframe domain.” J Virol 74(10): 4621-4633.
Boffa L.C., Morris P.L. et al. (1996).
Bibliografia
“Invasion of the CAG triplet repeats by a complementary peptide nucleic acid inhibits
transcription of the androgen receptor and TATA binding protein genes and correlates
with refolding of an active nucleosome containing a unique AR gene sequence.” J Biol
Chem 271(22): 13228-13233.
Norton J.C., Piatyszek M.A. et al. (1996).
“Inhibition of human telomerase activity by peptide nucleic acids.” Nat Biotech 14(5):
615-619.
Faruqi A.F., Egholm M. et al. (1998).
“Peptide nucleic acid-targeted mutagenesis of a chromosomal gene in mouse cells.” Proc
Natl Acad Sci USA 95(4): 1398-1403.
Branden U., Mohamed A.J. et al. (1999).
“A peptide nucleic acid-nuclear localization signal fusion that mediates nuclear transport
of DNA.” Nat Biotechnol 17(8): 784-787.
Basu S. and Wickstrom E. (1997).
“Synthesis and characterization of a peptide nucleic acid conjugated to a D-peptide
analog of insulin-like growth factor 1 for increased cellular uptake.” Bioconj Chem 8(4):
481-488.
Derossi D., Joliot A.H. et al. (1994).
“The third helix of the Antennapedia homeodomain translocates through biological
membranes”.
J Biol Chem 269(14): 10444-10450.
Hamilton S.E., Simmons C.G. et al. (1999).
“Cellular delivery of peptide nucleic acids and inhibition of human telomerase.” Chem
Biol 6(6): 343-51.
Bienz, M., H.Clevers (2000)
Linking colorectal cancer to WNT signalling Cell, Vol 103, 311-320
Bibliografia
Özakyol, A. et al (2006).
Fish detected p53 deletion and N-Myc amplification in colorectal cancer. Hepatogastroenterology; 53; 192-195
Melhem, M.F. et al. (1992).
Distribution of cells expressing myc proteins in human colorectal epithelium, polyps and
malignant tumors. Chem Biol 6(6): 343-51
SA Amundson, Q Zhan, LZ Penn and AJ Fornace Jr. (1998).
Myc suppresses induction of the growth arrest genes gadd34, gadd45, and gadd153 by
DNA-demaging agents. Oncogene 17, 2149-2154.
Yan Wang, Ingo H. Engels, D. A. Knee et al. (2004).
Synthetic lethal targeting of MYC by activation of the DR5 death receptor pathway.Cancer
cell 4, 501-512.
Smyth, M.J., Takeda, K., Hayakawa et al. (2003).
Nature’s TRAIL-on a path to cancer immunotherapy. Immunity 18, 1-6.
Ashkenazi, A. (2002).
Targeting death and decoy receptors of the tumour-necrosis factor superfamily. Nat. Rev.
Cancer 2, 240-430.
S Oyadomari and Mori (2004).
Roles of Chop/Gadd153 in endoplasmatic reticulum stress. Cell death and differentiation
11,381-389).
Shi-Yong Sun, Xiangguo Liu, Ping Yue et al.(2007).
The farnesyltransferase inhibitor Lofarnib induces CHOP-dependent expression of death
receptor5 leading to induction of apoptosis in human cancer cells. Biochemistry and
Molecular Biology, 2007.
Bibliografia
George C Prendergast (1999)
“Mechanisms of Apoptosis by c-Myc” Oncogene 1999, 2967-2987
Huang, E.H. and M.S.Wicha (2008)
Colon cancer stem cells: implications for prevention and therapy Trends in molecular medicine;
Vol.14; No.11.
Ilyas, M. and I.P.M. Tomlinson(1996)
Genetic pathways in colorectal cancer. Histopathology 28, 389-399.
Ringraziamenti
RINGRAZIAMENTI
Eccoci qua alla fine di queste sudate pagine..eccoci qua alla fine di tutto
questo percorso.... malinconia per quanto ormai rimane alle spalle....curiosità
per quello che c’è dietro l’angolo.... l’impazienza di gettarsi in ciò che
arriverà....malinconia perchè come succede in questi casi è piu forte il ricordo
dalle sfumature rosa, quello positivo e dolce del passato che non il pensiero
delle difficoltà e delle crisi attraverso le quali inevitabilemnte si è passati....
d’altro canto ci si inizia a chiedere cosa ci aspetti al di là di questo piccolo
grande traguardo....
Arrivati a questo punto come poter fare a meno di chiamare in ballo tutte le
persone importanti che ci sono state a fianco a me per una piu o meno grande
parte di questo lungo percorso e quelle che ancora mi accompagnano,
essenziali per me ognuna a suo modo.
Anzitutto grazie alla mia famiglia, in particolar modo ai miei genitori e al mio
fratellone. Grazie per tutto, grazie perchè in un modo o nell’altro sono sempre
presenti nei momenti migliori pronti a condividere con me le gioie ma
soprattutto nei momenti più duri pronti a farmi sentire il loro affetto e il loro
essenziale e indispensabile supporto.
Ringrazio il laboratorio che mi ha “ospitato” per un anno. Anzitutto
ringrazio il Prof. Andrea Pession, direttore medico del “Laboratorio di
Oncologia ed Ematologia Pediatrica del Policlinico Sant’Orsola –
Malpighi” dove ho svolto il tirocinio durante quest’ultimo anno e dove ho
imparato tante cose tecniche e non solo.
Ringraziamenti
Ringrazio il Dott. Roberto Tonelli per avermi seguito con serietà nel lavoro
svolto.
Ringrazio tutte le persone del laboratorio di Oncoematologia Pediatrica alle
quali in un modo o nell’altro devo qualcosa di quello che ho imparato nel
corso di questi mesi. Un grazie speciale a Consu, Ester, la Berganza ed Erika
che sono riuscite con gran premura ad essermi vicino, ad essere vicino a noi
tesisti e che non solo ci hanno supportato nella nostra formazione in
laboratorio ma ci hanno offerto tutta la loro disponibilità e il loro affetto,
hanno sempre cercato di alleggerire con una risata o con una parola
consolatoria il peso di queste ultime settimane.
Grazie anche a Valeria per tutte le indimenticabili pause caffè vissute insieme
e per tanti tentativi (puntulamente falliti) di organizzzazione di incontri extralab.
Ringrazio i miei compagni di avventura Marco, Elena e Antonio per lo
scambio e il confronto costante su preoccupazioni dubbi e quant’altro, senza il
quale sarebbe stata molto più dura soppportare la fatica soprattutto di
quest’ultimo mese.
In particolar modo ringrazio Elena per avermi permesso di riscoprirla e di
apprendere di lei molte piu cose in questa manciata di mesi piuttosto che negli
anni passati e malgrado incomprensioni incontrate lungo la via spero le nostre
strade non si tornino a separare e di mantenere il legame con le speciale
persona che si è rivelata.
Ringrazio Fascio per il reciproco scambio di idee e preoccupazioni, per i
pichhi di euforia e i momenti di immensa agitazione condivisi....ringrazio il
mio amico perche spesso ha avuto la parola giusta al momento giusto, per
avermi in più e più momenti davvero sopportato...grazie Fascio di tutto.
Ringraziamenti
Un particolare ringraziamento va al Prof. Giorgio Gallinella per la sua
immensa umanità e disponibiità e per aver creduto in me ed essere stato
capace di infondermi fiducia nelle mie possibiità anche nei momenti in cui ero
io per prima a dubitarne.
Ringrazio tutti gli amici che si sono susseguiti in momenti diversi per la loro
immensa pazienza, per il loro affetto e per essermi stati a fianco in questi
ultimi tempi o in qualche modo lungo il cammino.
Ringrazio la mia Carmenuzza che dopo così tanti anni c’è ancora, sempre
presente allo stesso modo, anche se i chilometri ci sono avversi....sempre e
incondizionatamente.
Ringrazio Marco, Simone, Saretta Petta, Federica e i piu lontani Alessia
Alessandra e Alberto, compagni di tante serate, risate, bevute in compagnia,
pomeriggi di studio insieme e di tutti gli altri bei momenti vissuti in questi
ultimi anni.
Ringrazio Vale, Fra e Sabrina per essermi state vicino in quest’ultimo periodo
e avermi sopportato, per aver condiviso con me piacevoli serate e per aver
sopportato le mie fasi diciamo di “latitanza”.
Ringrazio inoltre persone che mi hanno dato tanto....grazie e Chiara e Anissa
con i nostri incontri sporadici ma tanto piacevoli ...ringrazio Nicoletta
compare di tanti bei momenti e parte importante di tanti miei bei ricordi di
questi anni. Ringrazio Daniela.. così capita che due strade che sembrano
imboccare direzioni diverse possano tornare a riavvicinarsi.
Ringraziamenti
Ringrazio fra gli altri (riportati solo ora ma nn perchè meno importanti) amici
che ci sono sempre stati o ultimamente riscoperti ...Carmela, Luigi, Pio,
Mariachiara, Roberto, Ilaria.
Infine poche parole ma preziose per ringraziare una persona speciale lasciata
per ultima perchè particolarmente importante per me...una pesrona che nella
sua discrezione mi è stata e mi è immensamente vicina...che a modo suo mi
ha supportato in questo periodo davvero più di quanto potessi credere
aiutandomi a sentire tutto un pò più leggero, un pò più semplice. Un GRAZIE
per esserci e forse ancora un piu grande SCUSA per aver messo a dure prova
la tua pazienza....GRAZIE PER TUTTO!!
Grazie a tutti!!!!!!!
26 marzo 2009