Corso di Laurea Specialistica in Biologia Sanitaria, Universita' di Padova
C.I. di Metodi statistici per la Biologia, Informatica e Laboratorio di Informatica (Mod. B)
Docente: Dr. Stefania Bortoluzzi
Materiale del corso: http://telethon.bio.unipd.it/bioinfo/AA2006-2007/HomeStatBioinfo.html
IV ESERCITAZIONE
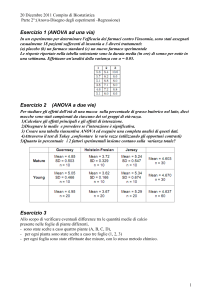
Analisi di dati sperimentali (misure di luminescenza) sull'efficienza di diversi
costrutti nell'attivazione dell' espressione di un gene reporter in una linea
cellulare: ANOVA PER CONFRONTI MULTIPLI.
Metodi: Statistiche descrittive, ANOVA
Programmi: EXCEL e SPSS.
Uno dei metodi sperimentali per lo studio dell’attivita’ di un promotore genico e’ quello delle
delezioni progressive. A partire dalla sequenza del promotore si isolano delle regioni di dimensioni
diverse e ciascuna di queste sequenze viene clonata insieme ad un gene reporter in un vettore
d’espressione e in uno specifico tipo cellulare, con lo scopo di saggiarne l’attivita’.
Se il gene reporter e’ quello per la luciferasi il livello d’espressione viene misurato attraverso la
rilevazione dell’intensita’ di luminescenza di un campione di cellule lisate. Dall’intensita’ di
luminescenza osservata si stima il numero di molecole di luciferasi nel campione, cioe’ l’attvita’
trascrizionale . Per fare cio’ e’ necessario normalizzare l’intensita’ di luminescenza osservata con
quella di un campione di riferimento contenente una quantita’ nota di luciferasi. Inoltre, una volta
stimata la quantita’ di luciferasi di ciascun campione, e’ necessario normalizzarla rispetto al numero
originale di cellule in esso contenute, per avere un stima dell’attivita’ trascrizionale di ciascun
costrutto. Per far cio’, si usa come riferimento la quantita’ totale di proteine del campione,
proporzionale al numero di cellule contenute. Questa viene misurata rilevando l’assorbanza allo
spettrofotometro alla lunghezza d’onda di 595 nm, dopo lisi cellulare e trattamento con un reagente
specifico.
Il dato finale e’ quindi espresso come pico grammi di luciferasi per microgrammi di proteine
cellulari. Generalmente lo schema sperimentale prevede diverse repliche per ciascun campione.
DATI
4 diversi costrutti ottenuti per delezioni progressive (-400/-1, -300/-1, -200/-1, -100/-1) di
DNA clonati in associazione con un gene reporter (codificante per la luciferasi) in cellule di
mammifero.
Per ciascun costrutto, 6 duplicati (24 campioni) e relative misure di luminescenza ottenute al
luminometro .
Per ciascun campione, stima del contenuto in proteine come assorbanza allo
spettrofotometro a lunghezza d’onda 595 nm.
Una misura di riferimento della luminescenza di una quantita’ nota di luciferasi (100 pg).
1
Scaricare il file di excel con la tabella originale contenete i dati sperimentali;
campione
di
riferimento
-400/-1
-400/-1
-300/-1
-300/-1
-200/-1
-200/-1
-100/-1
-100/-1
lum * 100 intensita' di micro intensita' di micro intensita' di micro intensita' di micro
pg
luminescenza grammi luminescenza grammi luminescenza grammi luminescenza grammi
(LC)
proteine (LC)
proteine (LC)
proteine (LC)
proteine
cellulari
cellulari
cellulari
cellulari
(CP)
(CP)
(CP)
(CP)
1055
200
260
210
230
225
216
5,0
8,0
6,0
6,0
5,5
6,1
400
531
387
423
386
509
7,1
9,0
7,0
7,3
7,3
8,9
376
405
430
461
395
345
8,0
6,5
7,9
8,0
6,3
5,5
18
10
20
13
35
21
6,0
8,0
6,0
5,1
10,5
6,1
Utilizzando Excel:
2
Normalizzare l’intensita’ di luminescenza di ciascun campione (LC) rispetto al campione
di riferimento (LR):
LN = (LC / LR) * 100 pg
Si ottiene una misura in picogrammi.
3
Normalizzare poi la quantita’ di luciferasi di ciascun campione (LN) rispetto al
contenuto proteico (CP):
LNN = LN / CP
Si ottiene una misura dell’attivita’ trascrizionale (LNN) espressa in picogrammi
luciferasi su microgrammi proteine cellulari.
4
Ottenuti sei valori di LNN per ciascuno dei quattro campioni, calcolare la media
campionaria e la deviazione standard () per ciascun costrutto;
5
Display grafico dei dati con un istogramma con intervallo di confidenza al 95%
(media 1.96
N
)
6
Interpretazione descrittiva dei risultati.
7
Preparare il file per SPSS.
Utilizzando SPSS:
8
Cut and paste dei dati preparati al punto 7;
9
Controllo dei calcoli precedenti attraverso l’opzione”statistiche descrittive”;
10
ANOVA con il post hoc (statistiche descrittive, test di omogeneita’ delle varianze,
ANOVA univariata e test post hoc LSD e Tamhane)
11
12
Interpretazione dei risultati.
-
Le varianze sono omogenee?
-
Le differenze osservate tra le medie dei campioni sono significative?
-
Quale test post hoc e’ applicabile?
-
Quali sono le medie che differiscono significativamente dalle altre?
CONSEGNARE UNA RELAZIONE CONTENENTE:
-
Riassunto dei contenuti dell’esercitazione (massimo 150 parole).
-
Risposte ai punti in grassetto.
VADEMECUM
Analisi della varianza (ANOVA)
Il metodo ANOVA permette di investigare l’effetto della variazione dei fattori sulla variabilità dei
risultati sperimentali del sistema sotto osservazione, determinando quale variazione è imputabile ai
fattori stessi e quale ad effetti casuali. Per ogni “trattamento” si devono eseguire alcune repliche, in
modo da poter avere una stima della variabilità del trattamento. Quando il numero di misure
replicate è uguale per tutti i trattamenti si ha un esperimento bilanciato (balanced experiment)
altrimenti si ha un esperimento sbilanciato (unbalanced experiment).
Per effettuare un’analisi one-way ANOVA ci sono due condizioni che debbono essere rispettate:
a)
le osservazioni per ogni trattamento devono essere distribuite normalmente,
b)
la varianza globale deve essere costante.
Il metodo ANOVA, come il metodo del t di Student, si propone di verificare l’ipotesi nulla, cioè che
non ci siano differenze dovute ai trattamenti, sul risultato dell’analisi ovvero, in altre parole, che le
differenze osservate siano dovute a fluttuazione casuali.
Per effettuare l’analisi si testa l’ipotesi nulla, considerando che la varianza dei dati può esser
distribuita in due parti:
tra le medie dei trattamenti,
nei trattamenti.
Si confrontano quindi le varianze con un test F, per verificarne l’uguaglianza.
I gradi di libertà totali associati all’analisi sono pari a:
I gradi di libertà “between-treatment” sono:
I gradi di libertà dell’errore residuo sono:
In conclusione si ottiene una tabella come la seguente:
Source of variation
Between-treatment
Residual error
Total
d.f.
k-1
N-k
N-1
Sum of Square
SA
SR
Mean of Square
SA/(k-1)
SR/(N-k)
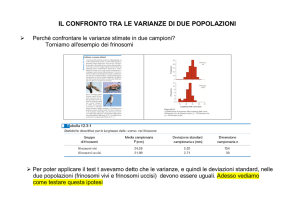
A questo punto abbiamo due valori di varianza da confrontare: si può dire subito che se la varianza
dovuta ai trattamenti è molto superiore all’errore residuo, ci sono poche speranze che l’ipotesi nulla
possa essere accettata.
In generale, per confrontare tra loro le varianze si opera un test-F, facendo il rapporto tra la varianza
between-treatment e quella dei residui.
Si utilizza la distribuzione ad una coda riferendosi ai gradi di libertà per entrambi i fattori calcolati
in precedenza.
In sintesi, il test ANOVA ci dice SE vi sono delle differenze significative tra diversi campioni nel
gruppo dei campioni considerati.
Comparazione multipla tra i trattamenti (post-hoc).
Una volta eseguito il test ANOVA ed evidenziata la presenza di disuguaglianze tra i trattamenti, è
interessante andare a vedere quali differenze sussistono tra i vari livelli, cioè quali trattamenti sono
significativamente diversi dagli altri e quali no. Esistono diversi test che ci aiutano nell’interpretare
le k(k-1)/2 combinazioni possibili tre i k trattamenti. La comparazioni sono definite post-hoc, cioè
esplorano i dati per scoprire differenze significative, senza limitare l’analisi formulando alcuna
teoria a priori. I test post hoc sono concepiti per evitare che il grande numero di confronti necessari
comporti un aumento dell’incertezza con la quale si può affermare che l’ipotesi nulla è verificata o
meno.
Fisher last significance difference (LSD)
Confronta tutte le medie, ma va impiegato solamente dopo che il test F dell’ANOVA ha indicato
l’effettiva presenza di trattamenti diversi. Può essere impiegato anche nel caso che i trattamenti
abbiamo un numero di campioni diversi. E’ il test meno conservativo.
Tamhane test
Adatto per fare tutti i confronti multipli quando le varianze non sono uguali.