Valutazione della sensibilità in vitro agli antimicrobici, ricerca dei fattori di virulenza e di geni
responsabili dell’antibiotico-resistenza in Escherichia coli enteropatogeni isolati da coniglio
Anna Giannina Perugini1, Anna Cerrone1, Fabrizio Agnoletti2, Lorella Barca1, Elena Mazzolini3, Giovanni
Cattoli4, Vincenzo Caligiuri1, Mario Bartoli1, Federico Capuano1
1Istituto Zooprofilattico Sperimentale del Mezzogiorno, Via Salute 2, 80055 Portici (Na)
2Istituto Zooprofilattico Sperimentale delle Venezie, Laboratorio di Treviso, Viale Brigata Treviso 13/A, 31100 Treviso, 3Istituto Zooprofilattico
Sperimentale delle Venezie, Laboratorio di Udine, Via Della Roggia 100, 33030 Campoformido (UD)
4Istituto Zooprofilattico Sperimentale delle Venezie, Laboratorio di Virologia, Viale dell’Università 10, 35020 Legnaro (PD)
Introduzione
Le infezioni da Escherichia coli enteropatogeni (EPEC) rappresentano
una frequente causa di patologie gastro-intestinali nel coniglio. Tali
patogeni sono in grado di indurre non solo specifiche lesioni intestinali,
definite “attachment-and-effacement (A/E) lesion”, ma producono anche
un caratteristico pattern di aderenza, definito ‘localized adherence’ (LA)
(Adu-Bobie et al., 1998). Geni responsabili di LA e A/E sono stati
localizzati in due regioni, definite ‘LEE region’ e EAF, all’interno delle
quali, oltre a geni codificanti per sistemi di secrezione, è localizzato il
gene dell’intimina (eae in LEE). A promuovere l’adesione alle cellule
epiteliali intestinali del coniglio intervengono anche le fimbrie AF/R1 e
AF/R2, descritte rispettivamente nel ceppo RDEC-1 (O’Hanley e
Cantey, 1978) e negli isolati O103:H2 che non fermentano il ramnosio
(Milon et al, 1990).
Nonostante sia noto il meccanismo responsabile della patogenicità degli
EPEC nel coniglio, i trattamenti per le enterocoliti da Escherichia
risultano spesso inutili forse anche a causa della resistenza agli
antimicrobici, in grado di favorire la sopravvivenza nonché la
proliferazione di tali patogeni.
In questo studio per la prima volta ceppi REPEC (Rabbit
Enterophatogenic Escherichia coli), caratterizzati per la presenza dei
geni di patogenicità eae, AF/R1 o AF/R2, sono stati analizzati sia per la
sensibilità in vitro ad alcuni antimicrobici, comunemente usati in
medicina veterinaria, in particolar modo nell’allevamento del coniglio
da carne, sia per la presenza di alcuni dei determinanti genetici di
resistenza agli antibatterici potenzialmente trasferibili ad altri patogeni.
Materiale e metodi
Isolamento
198 ceppi di Escherichia coli sono stati isolati da conigli deceduti per
enterocolite in Italia. I campioni, raccolti durante gli esami necroscopici,
sono stati isolati su McConkey agar, incubato a 37° C per 24h. La
caratterizzazione biochimica delle colonie identificate è stata realizzata
mediante gallerie API 20E System (bioMerieux, Marcy l’Etoile, France).
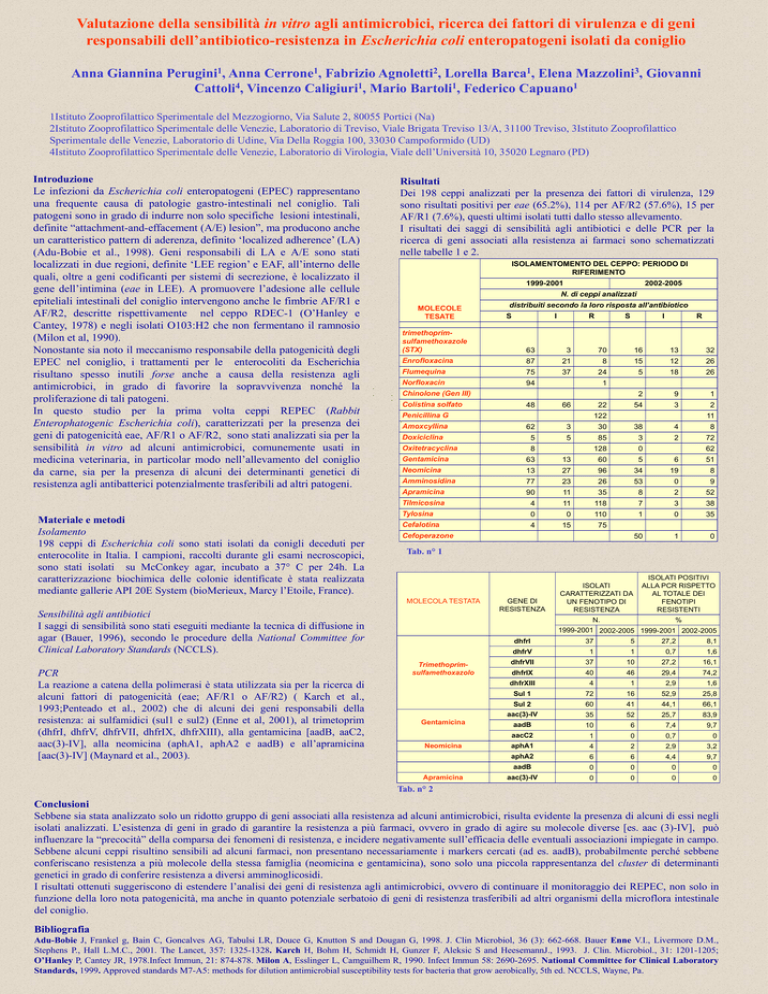
Risultati
Dei 198 ceppi analizzati per la presenza dei fattori di virulenza, 129
sono risultati positivi per eae (65.2%), 114 per AF/R2 (57.6%), 15 per
AF/R1 (7.6%), questi ultimi isolati tutti dallo stesso allevamento.
I risultati dei saggi di sensibilità agli antibiotici e delle PCR per la
ricerca di geni associati alla resistenza ai farmaci sono schematizzati
nelle tabelle 1 e 2.
ISOLAMENTOMENTO DEL CEPPO: PERIODO DI
RIFERIMENTO
1999-2001
N. di ceppi analizzati
MOLECOLE
TESATE
distribuiti secondo la loro risposta all’antibiotico
S
I
R
S
I
R
trimethoprimsulfamethoxazole
(STX)
63
3
70
16
13
32
Enrofloxacina
87
21
8
15
12
26
Flumequina
75
37
24
5
18
26
Norfloxacin
94
2
9
1
54
3
2
1
Chinolone (Gen III)
Colistina solfato
48
66
22
Penicillina G
Amoxcyllina
122
11
62
3
30
38
4
8
Doxiciclina
5
5
85
3
2
72
Oxitetracyclina
8
128
0
62
Gentamicina
63
13
60
5
6
51
Neomicina
13
27
96
34
19
8
Amminosidina
77
23
26
53
0
9
Apramicina
90
11
35
8
2
52
Tilmicosina
4
11
118
7
3
38
Tylosina
0
0
110
1
0
35
Cefalotina
4
15
75
50
1
0
Cefoperazone
Tab. n° 1
MOLECOLA TESTATA
Sensibilità agli antibiotici
I saggi di sensibilità sono stati eseguiti mediante la tecnica di diffusione in
agar (Bauer, 1996), secondo le procedure della National Committee for
Clinical Laboratory Standards (NCCLS).
PCR
La reazione a catena della polimerasi è stata utilizzata sia per la ricerca di
alcuni fattori di patogenicità (eae; AF/R1 o AF/R2) ( Karch et al.,
1993;Penteado et al., 2002) che di alcuni dei geni responsabili della
resistenza: ai sulfamidici (sul1 e sul2) (Enne et al, 2001), al trimetoprim
(dhfrI, dhfrV, dhfrVII, dhfrIX, dhfrXIII), alla gentamicina [aadB, aaC2,
aac(3)-IV], alla neomicina (aphA1, aphA2 e aadB) e all’apramicina
[aac(3)-IV] (Maynard et al., 2003).
2002-2005
GENE DI
RESISTENZA
ISOLATI
CARATTERIZZATI DA
UN FENOTIPO DI
RESISTENZA
ISOLATI POSITIVI
ALLA PCR RISPETTO
AL TOTALE DEI
FENOTIPI
RESISTENTI
N.
%
1999-2001 2002-2005 1999-2001 2002-2005
Trimethoprimsulfamethoxazolo
Gentamicina
Neomicina
Apramicina
dhfrI
37
5
27,2
8,1
dhfrV
1
1
0,7
1,6
dhfrVII
37
10
27,2
16,1
dhfrIX
40
46
29,4
74,2
dhfrXIII
4
1
2,9
1,6
Sul 1
72
16
52,9
25,8
Sul 2
60
41
44,1
66,1
aac(3)-IV
35
52
25,7
83,9
aadB
10
6
7,4
9,7
aacC2
1
0
0,7
0
aphA1
4
2
2,9
3,2
aphA2
6
6
4,4
9,7
aadB
0
0
0
0
aac(3)-IV
0
0
0
0
Tab. n° 2
Conclusioni
Sebbene sia stata analizzato solo un ridotto gruppo di geni associati alla resistenza ad alcuni antimicrobici, risulta evidente la presenza di alcuni di essi negli
isolati analizzati. L’esistenza di geni in grado di garantire la resistenza a più farmaci, ovvero in grado di agire su molecole diverse [es. aac (3)-IV], può
influenzare la “precocità” della comparsa dei fenomeni di resistenza, e incidere negativamente sull’efficacia delle eventuali associazioni impiegate in campo.
Sebbene alcuni ceppi risultino sensibili ad alcuni farmaci, non presentano necessariamente i markers cercati (ad es. aadB), probabilmente perché sebbene
conferiscano resistenza a più molecole della stessa famiglia (neomicina e gentamicina), sono solo una piccola rappresentanza del cluster di determinanti
genetici in grado di conferire resistenza a diversi amminoglicosidi.
I risultati ottenuti suggeriscono di estendere l’analisi dei geni di resistenza agli antimicrobici, ovvero di continuare il monitoraggio dei REPEC, non solo in
funzione della loro nota patogenicità, ma anche in quanto potenziale serbatoio di geni di resistenza trasferibili ad altri organismi della microflora intestinale
del coniglio.
Bibliografia
Adu-Bobie J, Frankel g, Bain C, Goncalves AG, Tabulsi LR, Douce G, Knutton S and Dougan G, 1998. J. Clin Microbiol, 36 (3): 662-668. Bauer Enne V.I., Livermore D.M.,
Stephens P., Hall L.M.C., 2001. The Lancet, 357: 1325-1328. Karch H, Bohm H, Schmidt H, Gunzer F, Aleksic S and HeesemannJ., 1993. J. Clin. Microbiol., 31: 1201-1205;
O’Hanley P, Cantey JR, 1978.Infect Immun, 21: 874-878. Milon A, Esslinger L, Camguilhem R, 1990. Infect Immun 58: 2690-2695. National Committee for Clinical Laboratory
Standards, 1999. Approved standards M7-A5: methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically, 5th ed. NCCLS, Wayne, Pa.












