I LOVE RESEARCH
CatanzaroOrienta
Promosso dalla Fondazione Università Magna Graecia
in collaborazione con l’Amministrazine Provinciale
TITOLO DEL PROGETTO:
IDENTIFICAZIONE DI MUTAZIONI SOMATICHE IN LINFOCITI INFILTRANTI IL
TUMORE IN SOGGETTI AFFETTI DA CANCRO COLORETTALE
STUDENTI partecipanti:
FALVO MARCO
MARUCA FRANCESCO
MAIDA ANGELO
MANCUSO TOMMASO
SCALZO GUGLIELMO
LICEO SCIENTIFICO I.I.S. “L.COSTANZO” DECOLLATURA
TUTORS:
DOTT.SSA EMILIA DORA GIOVANNONE
DOTT.SSA AURORA ANNA RITA NOCERA
OBIETTIVO FINALE DEL PROGETTO
Ad oggi diversi studi hanno considerato il microambiente tumorale geneticamente normale,
focalizzando l’attenzione sulle mutazioni presenti nelle cellule neoplastiche. Ipotizzando che ci
possano essere mutazioni a carico del microambiente in grado influenzare la crescita e progressione
tumorale, è stato deciso di valutare l’eventuale presenza di mutazioni nei linfociti infiltranti il
tumore in pazienti affetti da cancro colorettale.
BASE DI PARTENZA SCIENTIFICA
Il cancro colorettale (CRC) è una delle
neoplasie più frequenti, rappresenta la
terza causa più comune di cancro e la
quarta causa di morte su scala mondiale.
L’incidenza di tale neoplasia aumenta
progressivamente con l’età e la diagnosi
mediamente viene effettuata in età
compresa tra i 50 e 70 anni. Si registra
una maggiore incidenza nei Paesi
Europei, in Nord America ed Oceania
mentre una più bassa incidenza è
registrata
nell’Asia
Centrale
e
Meridionale e in Africa. La prognosi dei
pazienti affetti da cancro colorettale ha
subito
un
lento
ma
costante
miglioramento nel corso degli ultimi
decenni in molti paesi. La sopravvivenza
a 5 anni ha raggiunto quasi il 65% nei Paesi sviluppati (Australia, Canada, USA e paesi Europei)
ma resta più bassa (circa il 50%) nei Paesi sottosviluppati, risultando in generale maggiore nelle
donne rispetto agli uomini.
Diversamente dalle altre forme neoplastiche, per il cancro colorettale così come per il cancro al
polmone, non esiste un solo fattore di rischio. Oltre ad età e sesso, gli altri fattori di rischio
identificati sono: storia familiare, patologie infiammatorie intestinali, fumo, eccessivo consumo di
alcool e carne rossa, obesità e diabete (Brenner et al., 2014).
Il microambiente tumorale è formato dalle cellule stromali (fibroblasti, cellule endoteliali, periciti e
cellule mesenchimali), da cellule dell’immunità innata (macrofagi, neutrofili, mastociti, cellule
dendritiche e cellule natural killer) e da cellule dell’immunità adattativa (linfociti B e T). Queste
cellule comunicano tra di loro sia mediante contatto diretto sia mediante la produzione di citochine
e chemochine e controllano la crescita della massa tumorale mediante segnalazione paracrina o
autocrina (Grivennikov et al., 2010). Il cancro al colon è una delle neoplasie più studiate per la
relazione tra tumore e immunità poiché è altamente immunogenico e comunemente presenta
leucociti infiltranti. È noto che la risposta immunitaria di tale forma tumorale è diversa rispetto alla
mucosa sana adiacente. Questi dati implicano che il cancro al colon-retto induce una specifica
risposta immunitaria che però differisce ampiamente tra i pazienti. L’immunità tumorale sta dunque
emergendo come un fattore importante, sebbene non completamente chiaro, della progressione
tumorale. In particolare, la capacità di un tumore di evadere e sopprimere la risposta dei linfociti T,
influisce sulla sua sopravvivenza, così come sulla sua progressione e capacità di metastatizzare.
La presenza di linfociti tumorali infiltranti (TIL) è spesso interpretata come un tentativo di
rispondere alla proliferazione cellulare. (Sherwood A.M. et al., 2013). E’ noto in letteratura, che gli
stessi linfociti T citotossici così come i linfociti T helper di tipo 1 (Th1), di tipo 2 (Th2) e di tipo 17
1 (Th17), sono coinvolti nella
progressione tumorale. Allo
stesso modo le cellule Treg che
di solito mostrano un effetto
tumorigenico, in alcuni casi
presentano
un’azione
antitumorale, consistente nella
soppressione
del
processo
infiammatorio associato al
tumore.
L’unica
tipologia
cellulare che ad oggi non
sembra mostrare un’azione
tumorigenica sono le cellule
natural killer (NK).
L’espressione
dei
vari
modulatori e mediatori della
risposta immune, così come la
presenza di diverse tipologie
cellulari nel microambiente
tumorale,
può
quindi
promuovere la crescita tumorale
o agire da immunità antitumorale. Come detto in precedenza, inoltre, la stessa tipologia cellulare
può avere in alcuni casi un effetto tumorigenico ed in altri un effetto antitumorale. Ad oggi non è
nota la causa di tale eterogeneità. (Grivennikov et al., 2010). In molti tumori, incluso il cancro al
colon-retto, la presenza, la densità e la tipologia dei TIL correlano con la prognosi e la
sopravvivenza. Nel cancro al colon-retto, le correlazioni sono così forti da poter considerare la
risposta antitumorale come un indicatore clinico prognostico affidabile. In particolare la presenza e
l’alta densità di CD3+ (CD8+, CD4+) e cellule della memoria sono associati ad una prognosi
favorevole, mentre la presenza e l’alta densità di linfociti CD4+Treg correla con una cattiva
prognosi. Inoltre l’alta espressione di geni coinvolti nella risposta delle cellule T citotossiche (ad
esempio interferone gamma) è associata ad un basso rischio di ricaduta (Sherwood A.M. et al
2013). Recenti studi sul cancro alla mammella hanno evidenziato la presenza di alterazioni a carico
delle cellule del microambiente tumorale, suggerendo che queste alterazioni possano favorire la
cancerogenesi. In particolare sono state inoltre riscontrate, nei linfociti tumorali infiltranti mutazioni
somatiche non presenti nelle cellule neoplastiche. (Kleppe M. et al 2015). Alla luce di queste
osservazioni, considerare le cellule del microambiente come un compartimento geneticamente
instabile, potrebbe fornire nuovi elementi per comprendere il motivo per il quale la medesima
tipologia cellulare in alcuni casi promuova e in altri inibisca la progressione tumorale.
RIFERIMENTI BIBLIOGRAFICI
-
-
Brenner H, Kloor M, Peter Pox C. Colorectal cancer. Lancet 2014; 383:1490-1501
Sherwood A.M., Emerson Ryan O., Scherer D., Habermann N., Buck K., Staffa J.,
Desmarais C., Halama N., Jaeger D. Tumor- infiltrating lymphocytes in colorectal tumors
display a diversity of T cell receptor sequences that differ from T cells in adjacent mucosal
tissue. Cancer Immunology, Immunotherapy 2013; 62:1453-1461.
Kleppe M. et al Somatic mutation in leucocyte infiltranting primary breast cancer - npj
Breast Cancer (2015).
Sergei I. Grivennikov, Florian R. Greten, and Michael Karin. Immunity, Inflammation, and
Cancer. Cell. 2010; 140(6): 883–899.
2 METODOLOGIA
Arruolamento pazienti
Nello studio verranno arruolati 100 pazienti affetti da carcinoma del retto e del colon, qualunque sia
la sezione interessata (colon ascendente, trasversale, discendente, sigmoideo), che rispondano ai
seguenti criteri:
• 18 anni compiuti;
• qualunque etnia o razza e senza preferenza di sesso;
• affetti da tumore allo Stadio I (T1/T2 N0 M0) o Stadio IIA (T3 N0 M0) o Stadio IIB ( T4
N0 M0);
• non sottoposti a terapia cito riduttiva (chemio o radio terapia).
Tutti i pazienti arruolati firmeranno un consenso informato.
Isolamento e caratterizzazione immunofenotipica
Il campione bioptico sarà sottoposto a dissociazione. La sospensione cellulare così ottenuta
contenente cellule tumorali, stromali e gli infiltrati leucocitari sarà filtrata, lavata in PBS 1X e
risospesa in Lysing buffer (BD) allo scopo di rimuovere i globuli rossi. Per ciascun paziente verrà
raccolto un campione di sangue periferico da utilizzare come controllo. Lo staining (incubazione 30
min, 4°C) sarà effettuato utilizzando anticorpi anti-Human (BD Bioscence). Le cellule saranno
successivamente lavate due volte in PBS 1X, fissate in 300 ul Cytofix (BD) incubando a 4°C per 20
min e risospese in 500 µl in PBS 1X. Per i campioni che necessitano di staining intracellulare, le
cellule saranno permeabilizzate allo scopo di effettuare lo staining per proteine intracellulari. Tutti i
campioni così trattati saranno isolati utilizzando il FACSAriaIII cell sorter (BD).
Estrazione del DNA
Il DNA verrà estratto utilizzando il PureLink Genomic DNA kits (Invitrogen). La qualità del DNA
estratto verrà analizzato utilizzando lo spettrofotometro Multiskan Go (ThermoFisher Scientific).
Next Generation Sequencing
Il DNA estratto sarà quantizzato utilizzando il Qubit (Invitrogen). Per ciascun campione sarà
preparata una libreria (utilizzando 10 ng di DNA) utilizzando Ion AmpliSeq™ Library Kit 2.0 (Life
Technologies) e il pannello Ion AmpliSeq Comprehensive Cancer Panel. La qualità del DNA sarà
valutata utilizzando il sistema BioAnalyzer (Agilent Thecnologies). Le librerie così ottenute
saranno sottoposte ad una PCR in emulsione mediante l’utilizzo del OneTouch System. I campioni
verranno sequenziati utilizzando la piattaforma IonTorrent/Proton. I dati così ottenuti saranno
analizzati mediante l’applicazione di diversi algoritmi bioinformatici.
3 ARTICOLAZIONE DEL PROGRAMMA DI RICERCA
I soggetti affetti da cancro colorettale, una volta reclutati, saranno sottoposti a intervento chirurgico.
Verrà prelevato un campione bioptico che sarà consegnato ai nostri laboratori, contestualmente al
campione di sangue dello stesso paziente precedentemente prelevato.
Allo scopo di caratterizzare e isolare le cellule tumorali e le diverse sottopopolazioni linfocitarie
(linfociti T helper, linfociti T citotossici, linfociti Treg), le biopsie tumorali saranno dissociate a
fresco allo scopo di ottenere una miscela di cellule monodisperse contenente cellule tumorali,
stromali e infiltrati leucocitari. La caratterizzazione delle sottopopolazioni linfocitarie sarà
effettuata secondo il pannello mostrato di seguito:
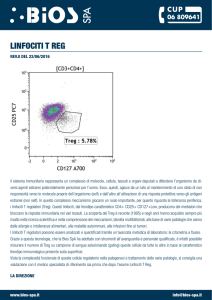
LINFOCITI T HELPER
LINFOCITI T CITOTOSSICI
LINFOCITI T REG
CD45
CD3
CD4
CD45
CD3
CD8
CD4
CD25
FOXP3
FITC
PE
APC
FITC
PE
APCCy5
APC
PerCp
PECy5
Ogni popolazione linfocitaria sarà positiva per ciascun antigene.
Per isolare le cellule tumorali dalle stromali, si utilizzerà il seguente pannello:
CELLULE TUMORALI
EpCAM
CD45
CK8 (Citocheratina 8)
CK18 (Citocheratina 18)
CK19 (Citocheratina 19)
CK20 (Citocheratina 20)
mCherry
FITC
ACPH7
ACPH7
ACPH7
ACPH7
Le cellule negative per i marcatori EpCAM e CD45 identificheranno la popolazione di cellule
stromali che sarà eliminata perché non oggetto di studio. Le cellule positive per i marcatori
EpCAM, CK8, CK18, CK19 e CK20 e negative per il marcatore CD45 identificheranno la
popolazione di cellule tumorali. La procedura di staining sarà eseguita come da protocollo in
precedenza illustrato. Le popolazioni cellulari (cellule tumorali e linfociti T CD8, CD4 e Treg),
identificate mediante caratterizzazione immunofenotipica, saranno opportunamente isolate mediante
cell sorting. Le sottopopolazioni linfocitarie (linfociti T CD8, CD4 e Treg) presenti nei campioni di
sangue saranno identificate e isolate utilizzando le stesse metodiche e gli stessi pannelli descritti per
l’infiltrato linfocitario.
Allo scopo di valutare il profilo mutazionale delle sottopopolazioni così ottenute, i campioni
saranno analizzati mediante Next Generation Sequencing (NGS) utilizzando il Comprensive Cancer
panel. Tale pannello consiste in 16000 coppie di primer in grado di amplificare 409 geni implicati
nel processo di cancerogenesi. I dati saranno sottoposti ad analisi bioinformatica.
4 OUTPUT DEL PROGRAMMA
L’impatto globale dell’immunità sugli eventi tumorigenici precoci è molto difficile da comprendere,
soprattutto per quanto riguarda le cause che determinano la diversa azione dei leucociti infiltranti il
tumore.
Recenti studi hanno evidenziato la presenza di alterazioni a carico del microambiente tumorale,
suggerendo che queste alterazioni possano favorire la cancerogenesi. In particolare mutazioni
somatiche assenti nelle cellule neoplastiche sono state invece riscontrate nei linfociti tumorali
infiltranti. Alla luce di queste osservazioni, considerare le cellule del microambiente come un
compartimento geneticamente instabile, potrebbe fornire nuovi elementi per comprendere il motivo
per il quale la medesima tipologia cellulare in alcuni casi promuova e in altri inibisca la
progressione tumorale.
In questo studio, mediante sequenziamento di nuova generazione, si cercherà di comprendere se, in
soggetti affetti da cancro al colon-retto, i linfociti T infiltranti possano presentare mutazioni
somatiche in geni notoriamente coinvolti nel processo di cancerogenesi. In futuro saranno necessari
studi funzionali al fine di stabilire eventuali correlazioni tra i linfociti mutati e la progressione
tumorale. Tale correlazione potrebbe avere importanti sviluppi sia da un punto di vista terapeutico
che prognostico.
In conclusione l’identificazione di mutazioni somatiche clonali a carico dei linfociti tumorali
infiltranti (TIL) potrebbe avere numerose implicazioni sullo sviluppo di nuove terapie che abbiano
come target non solo le cellule tumorali ma anche quelle del microambiente.
5