Macromolecole Biologiche
Biotecnologie applicate alla progettazione e
sviluppo di molecole biologicamente attive
A.A. 2010-2011
Modulo di Biologia Strutturale
Regolazione allosterica
Marco Nardini
Dipartimento di Scienze Biomolecolari e Biotecnologie
Università di Milano
Allosteria
Proteine allosteriche
- 2 o più siti di legame topologicamente distinti (in grado di legare substrati,
inibitori, attivatori) che interagiscono in modo funzionale fra loro
⇒ la formazione del legame di un ligando ad un sito altera le proprietà
dell’altro sito, in particolare la sua affinità per il secondo ligando
⇒ cooperatività:
- positiva: la modificazione aumenta la capacità di legame
- negativa: la modificazione diminuisce la capacità di legame
- effettori allosterici:
1) effettori allosterici omotropici: (il substrato è esso stesso effettore)
2) effettori allosterici eterotropici (effettore diverso da substrato)
es: inibizione/attivazione a feed-back
Allosteria
Proteine allosteriche
- cinetica non obbedisce al modello di Michaelis-Menten
E+S
VMAX = k2 [E]T
k1
k-1
ES
k2
E+P
velocità massima di una reazione
(quando l’enzima è totalmente nella forma ES ed [S] è elevata)
Eq. di Michaelis-Menten
v0 =
VMAX [S]
KM + [S]
iperbole rettangolare
Allosteria
Proteine allosteriche
- cinetica non obbedisce al modello di Michaelis-Menten
v ([S]) sigmoidale ⇒ v prop. [S]n con n>1
- a basse [S], un aumento di [S] causa solo un
lieve incremento di velocità
⇒ pochi siti sono occupati dal substrato ed
essi hanno una bassa affinità per S
- a più alte [S] si ha un drammatico incremento d velocità
⇒ man mano che si lega S, aumenta l’abilità dell’enzima di legare S
(cooperatività positiva)
- ad alte [S] andamento asintottico ⇒ raggiungimento saturazione (VMAX)
Allosteria
Proteine allosteriche
- gli effettori allosterici (attivatori/inibitori)
alterano la velocità di reazione dell’enzima
attivatore: aumenta la velocità di reazione dell’enzima
diminuisce il carattere sigmoide della curva
di saturazione
inibitore: diminuisce la velocità di reazione dell’enzima
aumenta il carattere sigmoide della curva di saturazione
Tali effettori operano sulla capacità di legare il substrato e quindi sulla KM
lasciando alterata la VMAX (Sistemi K)
Nel caso in cui l’effettore cambi la VMAX si parla di Sistemi V
Allosteria
Proteine allosteriche
struttura polimerica
- le proteine ed enzimi allosterici possiedono una struttura polimerica o
quaternaria
- l’interazione fra le subunità (interazioni deboli) è responsabile della
cooperatività da substrato
agenti denaturanti
- se sottoposti a blanda denaturazione, molti enzimi allosterici perdono le
loro proprietà allosteriche da substrato pur mantenendo attività catalitica
risposta bifasica a inibitori competitivi (inibitori che mimano il substrato)
- a basse [S], l’inibitore competitivo può aumentare le capacità dell’enzima a
legare substrato (agisce come un attivatore)
- ad alte [S], l’inibitore competitivo si comporta in modo normale rallentando
la reazione
Allosteria
Modelli di allosteria
(1) modello concertato o simmetrico (Monod, Wyman, Changeux)
(2) modello sequenziale (Koshland, Nemethy, Filmer)
Modello simmetrico
Premessa:
proteina allosterica formata da subunità che esistono in 2 stati
Stato R (rilassato)
Stato T (teso)
Allosteria
Modello simmetrico
Assunzioni:
- le subunità devono essere tutte nello stesso stato (R o T)
- stato R: alta affinità per il substrato
stato T: bassa affinità per il substrato
kT >> kR
- in assenza di ligandi i 2 stati sono in equilibrio (spostato verso T)
⇀ R0
T0 ↽
Esempio:
costante di equilibrio L = [T0]/[R0] = 104
(T0 e S0 in assenza di substrato S)
Allosteria
Modello simmetrico
Substrato S aggiunto a proteina allosterica all’equilibrio:
- legame prevalente a proteine in stato R
- spostamento dell’equilibrio verso R (diminuzione della concentrazione
della proteina nello stato T)
⇒ incremento dell’affinità complessiva
enzima-substrato
⇒ cooperatività positiva da substrato
Allosteria
Modello simmetrico
Effettori allosterici eterotropici
attivatore: si lega prevalentemente allo stato R
⇒ spostamento dell’equilibrio verso R
(stato ad alta affinità per il substrato)
⇒ diminuzione dell’apparente stato di cooperatività
(riduzione piede della sigmoide e tendenza della curva di saturazione a
diventare iperbolica)
inibitore: si lega prevalentemente allo stato T
⇒ spostamento dell’equilibrio verso T
(stato a bassa affinità per il substrato)
⇒ aumento del piede della sigmoide ⇒ necessaria una quantità maggiore
di substrato per raggiungere lo
stesso grado di cooperazione
Allosteria
Modello simmetrico
Effettori allosterici eterotropici
Allosteria
Modello sequenziale
Assunzione:
- le subunità possono essere sia nello stato R o che nello stato T
- equilibri complessi di cui l’equilibrio tra le forme pure R e T sono un
caso particolare (modello simmetrico)
- il substrato S ha una influenza diretta sulla forma dell’enzima
S aggiunto a proteina allosterica (prevalentemente nello stato T0):
- adattamento indotto
cioè il substrato entra nel sito attivo a causa di collisioni casuali e la
subunità si sistema attorno al substrato per produrre un buon adattamento
⇒ conversione nello stato R
Allosteria
Modello sequenziale
- se il cambio T→R in una subunità tende a spingere le altre subunità ad
assumere la forma R ⇒ cooperatività positiva
- il cambio conformazionale propagato può avvenire prima che si leghi
il nuovo substrato o rendere il processo di adattamento indotto più
semplice
- se il cambio T→R in una subunità rende più difficile alle altre subunità
la assunzione della forma R ⇒ cooperatività negativa
la cooperatività negativa non è spiegabile con il modello simmetrico (che
dipende solo dalla legge d’azione di massa tra le forme R e T)
Effettori allosterici eterotropici
attivatore: si lega in un sito ≠ dal substrato ma opera allo stesso modo
inibitore: rende l’enzima più rigido, rendendo più difficoltoso
l’adattamento indotto T→R
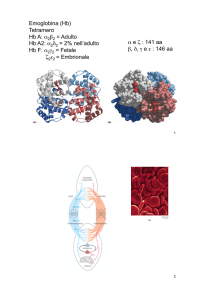
Mioglobina ed Emoglobina
Mioglobina
- Struttura (Kendrew, 1959, Mb di capodoglio)
(prima struttura determinata col metodo dei raggi X)
- 153 residui (proteina monomerica)
- 8 α-eliche (A-H), sandwich 3-su-3 (BEF-AGH)
C
D
- prototipo strutturale per le globine
F
- gruppo eme legato in tasca idrofobica
fra elica E ed F
A
Mioglobina ed Emoglobina
Cinetica di legame O2 per la Mioglobina (Mb):
- legame reversibile di O2 a Mb:
Mb + O2
reazione all’equilibrio
K=
costante di dissociazione
MbO2
[Mb][O2]
[MbO2]
La dissociazione dell’O2 da Mb può essere caratterizzata mediante la sua
“saturazione frazionale YO2 ”
YO2 =
[MbO2]
=
[Mb] + [MbO2]
essendo O2 un gas
[O2] come pO2
[O2]
K + [O2]
YO2 =
pO2
K + pO2
frazione di siti di legame di O2
occupati
pO2 = pressione parziale
(tensione ossigeno)
Mioglobina ed Emoglobina
Cinetica di legame O2 per Mb:
YO2 =
pO2
K + pO2
iperbole
rettangolare
- la Mb ha una p50 molto bassa (circa 2.8 torr)
e quindi una alta affinità per l’O2
- la Mb deve essere in grado di catturare, nel citoplasma di cellule
metabolicamente molto attive, l’O2 proveniente dal torrente circolatorio
(pO2 circa 30 torr) ed essere in grado di rilasciarlo ai mitocondri (pO2<4 torr)
- alle pO2 fisiologiche nel sangue (100 torr arterie, 30 torr vene) Mb è sempre
saturata (pO2 = 100 torr ⇒ YO2 = 0.97; pO2 = 30 torr ⇒ YO2 = 0.91)
⇒ Mb molto efficace per favorire il passaggio di O2 dai capillari alle
cellule muscolari
Mioglobina ed Emoglobina
Mioglobina
Eme:
- deossimioglobina: Fe(II) fuori dal piano dell’eme di 0.055 nm
(struttura a cupola)
- ossimioglobina: Fe(II) fuori dal piano dell’eme di 0.026 nm
- cambiamenti non rilevanti biologicamente per Mb ma fondamentali per la
regolazione allosterica di Hb
Mioglobina ed Emoglobina
Comportamento di ipotetiche proteine di trasporto dell’O2
dotate di curve di saturazione iperboliche
- una proteina di trasporto che lega l’O2 in modo analogo alla Mb non può
essere un efficiente trasportatore di O2 nel sistema circolatorio
Mioglobina ed Emoglobina
Cinetica di legame O2 per Hb:
- la curva sigmoide indica che Hb può
trasportare più O2 ai tessuti rispetto
al caso iperbolico (a pari p50)
- pressione venosa = 30 torr ⇒ YO2 = 0.55
pressione arteriosa = 100 torr ⇒ YO2 = 0.95
- differenza nella saturazione da O2 come misura della capacità della Hb di
rilasciare O2 dai polmoni (alle pO2 degli alveoli) ai tessuti (alle pO2 dei
capillari) :
fattore 0.40 nella saturazione da O2 (caso sigmoide)
fattore 0.25 nella saturazione da O2 (se la curva fosse iperbolica )
Mioglobina ed Emoglobina
Cinetica di legame O2 per Hb:
- curva di legame di O2 sigmoidale
⇒ cooperatività fra i siti di legame
- p50 = 26 torr per Hb
- inizialmente:
la pendenza della curva dell’affinità per
l’O2 è bassa perché le subunità competono
indipendentemente per il legame al primo O2
p50
- dopo:
quando una molecola di O2 si è legata ad una subunità, l’affinità delle altre
subunità aumenta e quindi aumenta la pendenza della curva
Mioglobina ed Emoglobina
Cinetica di legame O2 per Hb:
in ogni sistema di legame, una curva sigmoide è indicativa di interazioni
cooperative fra i siti di legame
Hb + nO2
Hb(O2)n
n = numero O2 legate in una singola tappa da Hb (cooperatività infinita)
equazione di Hill
(pO2)n
YO2 =
(p50)n + (pO2)n
grado di saturazione di Hb in funzione di pO2
-n (costante di Hill) non integrale come parametro di cooperatività fra
subunità nel legame di O2 (piuttosto che numero di subunità in grado di
legare O2 in una singola tappa) ⇒ relazione empirica per fittare i dati
n = 1 ⇒ iperbole (non-cooperatività)
n > 1 ⇒ cooperatività positiva (cioè il legame di O aumenta l’affinità di Hb per il legame di altro O )
n < 1 ⇒ cooperatività negativa (cioè il legame di O riduce l’affinità di Hb per il legame di altro O )
2
2
2
2
Mioglobina ed Emoglobina
Emoglobina
Struttura: α2β2 (tetramero)
- dimero di protomeri αβ (simmetria C2)
- pseudosimmetria binaria fra α e β (pseudosimmetria D2)
- α e β evoluzionisticamente
correlate fra loro e con Mb
(stessa struttura, no elica D in α)
- il legame dell’O2 altera la
struttura del tetramero
Mioglobina ed Emoglobina
α2
β2
β1
α1
α2
β2
deossi-Hb
β1
α1
ossi-Hb
il legame dell’O2 altera la struttura del tetramero:
- cambiano i contatti α1-β2 e α2-β1
- rotazione 15° fra i 2 dimeri αβ avvicinamento
subunità β
- restrizione canale centrale contenente solvente
(all’interfaccia α1-α1 e β1-β1)
Mioglobina ed Emoglobina
Meccanismo di Perutz
Stato T = deossi-Hb (stato ”teso”)
Stato R = ossi-Hb (stato “rilassato”)
il legame dell’O2 innesca una serie di movimenti coordinati che altera la
struttura quaternaria iniziale e determina il passaggio T→R
la transizione T→R consiste in una
rotazione reciproca dei 2 dimeri αβ
Mioglobina ed Emoglobina
Meccanismo di Perutz
(1) stato T: Fe(II) fuori dal piano dell’eme (0.06 nm) verso HisF8
(struttura a cupola)
(2) si lega O2: modifica stato elettronico dell’eme, si accorciano i legami
Fe-Nporfirina, si annulla la forma a cupola dell’eme
Mioglobina ed Emoglobina
Meccanismo di Perutz
passaggio T→R:
- movimento di 0.6Å di HisF8 verso l’eme
- traslazione di ~ 1Å di tutta l’elica F
(per evitare interferenze steriche)
- rotazione dell’angolo FG
Mioglobina ed Emoglobina
Meccanismo di Perutz
(3) accoppiamento variazioni terziarie e riorganizzazione quaternaria
modifiche all’interfaccia α1-β2 e α2-β1
stato T: contatto His(97)FG4(β2) - Thr(41)C6(α1)
stato R: contatto His(97)FG4(β2) - Thr(38)C3(α1) (un giro di elica prima)
in entrambi i casi buon impaccamento protuberanze/scanalature
Mioglobina ed Emoglobina
Meccanismo di Perutz
(4) modifiche ai residui C-terminali
stato T: Arg(141)α, His(146)β parte di reticolo di coppie ioniche intered intra-subunità che stabilizza lo stato T
stato R: rottura coppie ioniche
stato T
Mioglobina ed Emoglobina
Meccanismo di Perutz
- Hb nello stato T ha bassa affinità per O2
(lunghezza dei legami Fe-N superiore di 0.1 Å rispetto a stato R)
- a seguito del legame dell’O2 ad una subunità nello stato T, l’atomo di Fe2+
torna nel piano della porfirina (da 0.6 Å a 0.2 Å)
- tale movimento si trascina dietro l’elica F e modifica i tratti EF ed FG
- tali movimenti sono trasmessi all’interfaccia tra le subunità e determinano la
rottura dei ponti salini al C-terminale
- tutte le subunità vengono convertite simultaneamente nello stato R (alta
affinità per O2) indipendentemente dal fatto che O2 sia ad esse legato
- le subunità nello stato R ma non impegnate da O2 hanno aumentato la loro
affinità per O2 in quanto nella conformazione corretta
- nello stato T, l’O2 è accessibile solo ai gruppi eme delle subunità α (ma non
le β) per ingombro sterico ⇒ impedimento assente dopo la transizione T→R
Mioglobina ed Emoglobina
Meccanismo di Perutz
- le subunità α e β sono così saldamente accoppiate che una modificazione
della struttura terziaria in una subunità non può non influenzare la struttura
quaternaria dell’intera proteina
- Hb ha solo 2 stati quaternari, T ed R: i contatti inter-subunità agiscono
come “interruttori binari”
- la mancata flessibilità alle interfacce α1-β1 e α2-β2 fa sì che la transizione
T→R avvenga simultaneamente alle interfacce α1-β2 e α2-β1
- esistono effettori allosterici: H+, CO2, Cl-, BPG (D-2,3-bisfosfoglicerato)
Hb e allosteria
Hb ed allosteria
il legame di O2 ad Hb esprime proprietà associate ad entrambi i modelli
- la transizione quaternaria T→R è concertata come nel modello simmetrico
- il legame del ligando nello stato T determina modifiche alla struttura
terziaria delle subunità, necessarie per la conversione nello stato R
(come richiesto dal modello sequenziale)
Allosteria
Fosfofruttochinasi (PFK)
Reazione 3 della glicolisi
fosforilazione del fruttosio-6-fosfato (F6P) a formare fruttosio-1,6-bisfosfato (FBP)
- attacco nucleofilico del gruppo C1-OH dell’F6P sull’atomo di fosforo γ elettrofilico del
complesso Mg2+-ATP
+ H+
- PFK opera in condizioni lontane dall’equilibrio (rappresenta il punto di controllo
maggiore della glicolisi
Allosteria
Fosfofruttochinasi
- enzima tetramerico in 2 stati
conformazionali R e T in equilibrio
- ATP è sia substrato sia inibitore
allosterico di PFK (2 siti di legame
distinti)
- legame ATP (substrato): uguale efficienza
di legame sia per stato T che R
Mg2+-ATP
Mg2+-ADP
F6P
F6P
- legame ATP (inibitore): legame di ATP
quasi solo in conformazione T
- legame F6P:
legame di preferenza in stato R
Mg2+-ATP
Mg2+-ADP
2 subunità dell’enzima tetramerico
(ulteriore asse binario verticale)
Allosteria
Fosfofruttochinasi
ad alta conc. l’ATP agisce come inibitore allosterico di PFK legandosi allo stato T e
spostando l’equilibrio T
R verso T (curva iperbolica ⇒ curva sigmoidale)
- gli attivatori AMP e ADP bilanciano l’inibizione da ATP legandosi allo stato R
(stabilizzandolo) e spostando l’equilibrio T
R verso R
- alta [ATP] (bassa richiesta metabolica) ⇒ PFK inibita
- bassa [ATP], PFK non inibita ⇒ sintesi ATP
Allosteria
Fosfofruttochinasi
- lo stato R di PFK viene stabilizzato dal legame del substrato F6P
(Arg162 forma coppia ionica con gruppo fosforico di F6P)
- stato R → stato T ⇒ srotolamento elica
- nello stato T Arg162 è sostituita da Glu161
⇒ affinità diminuita del gruppo fosforico
per lo stato T
- srotolamento dell’elica impedito
dal legame dell’attivatore ADP
- legame di ATP (inibitore) possibile
solo con elica srotolata (stato T)