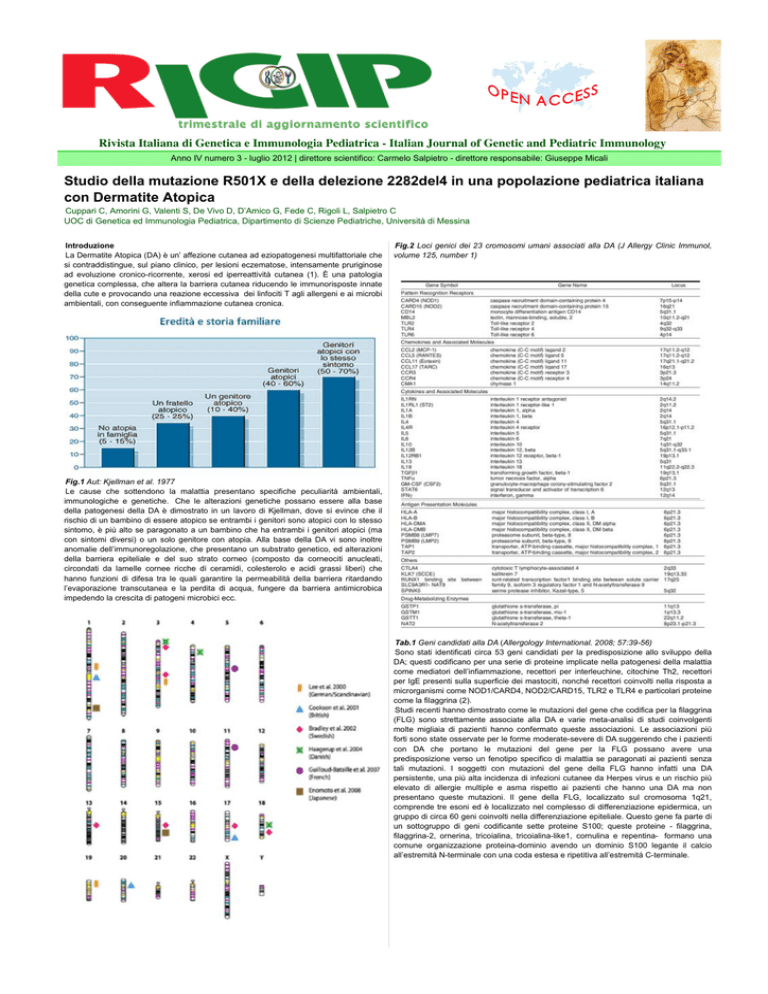
Rivista Italiana di Genetica e Immunologia Pediatrica - Italian Journal of Genetic and Pediatric Immunology
Anno IV numero 3 - luglio 2012 | direttore scientifico: Carmelo Salpietro - direttore responsabile: Giuseppe Micali
Studio della mutazione R501X e della delezione 2282del4 in una popolazione pediatrica italiana
con Dermatite Atopica
Cuppari C, Amorini G, Valenti S, De Vivo D, D’Amico G, Fede C, Rigoli L, Salpietro C
UOC di Genetica ed Immunologia Pediatrica, Dipartimento di Scienze Pediatriche, Università di Messina
Introduzione
La Dermatite Atopica (DA) è un’ affezione cutanea ad eziopatogenesi multifattoriale che
si contraddistingue, sul piano clinico, per lesioni eczematose, intensamente pruriginose
ad evoluzione cronico-ricorrente, xerosi ed iperreattività cutanea (1). È una patologia
genetica complessa, che altera la barriera cutanea riducendo le immunorisposte innate
della cute e provocando una reazione eccessiva dei linfociti T agli allergeni e ai microbi
ambientali, con conseguente infiammazione cutanea cronica.
Fig.2 Loci genici dei 23 cromosomi umani associati alla DA (J Allergy Clinic Immunol,
volume 125, number 1)
Fig.1 Aut: Kjellman et al. 1977
Le cause che sottendono la malattia presentano specifiche peculiarità ambientali,
immunologiche e genetiche. Che le alterazioni genetiche possano essere alla base
della patogenesi della DA è dimostrato in un lavoro di Kjellman, dove si evince che il
rischio di un bambino di essere atopico se entrambi i genitori sono atopici con lo stesso
sintomo, è più alto se paragonato a un bambino che ha entrambi i genitori atopici (ma
con sintomi diversi) o un solo genitore con atopia. Alla base della DA vi sono inoltre
anomalie dell’immunoregolazione, che presentano un substrato genetico, ed alterazioni
della barriera epiteliale e del suo strato corneo (composto da corneociti anucleati,
circondati da lamelle cornee ricche di ceramidi, colesterolo e acidi grassi liberi) che
hanno funzioni di difesa tra le quali garantire la permeabilità della barriera ritardando
l’evaporazione transcutanea e la perdita di acqua, fungere da barriera antimicrobica
impedendo la crescita di patogeni microbici ecc.
Tab.1 Geni candidati alla DA (Allergology International. 2008; 57:39-56)
Sono stati identificati circa 53 geni candidati per la predisposizione allo sviluppo della
DA; questi codificano per una serie di proteine implicate nella patogenesi della malattia
come mediatori dell’infiammazione, recettori per interleuchine, citochine Th2, recettori
per IgE presenti sulla superficie dei mastociti, nonché recettori coinvolti nella risposta a
microrganismi come NOD1/CARD4, NOD2/CARD15, TLR2 e TLR4 e particolari proteine
come la filaggrina (2).
Studi recenti hanno dimostrato come le mutazioni del gene che codifica per la filaggrina
(FLG) sono strettamente associate alla DA e varie meta-analisi di studi coinvolgenti
molte migliaia di pazienti hanno confermato queste associazioni. Le associazioni più
forti sono state osservate per le forme moderate-severe di DA suggerendo che i pazienti
con DA che portano le mutazioni del gene per la FLG possano avere una
predisposizione verso un fenotipo specifico di malattia se paragonati ai pazienti senza
tali mutazioni. I soggetti con mutazioni del gene della FLG hanno infatti una DA
persistente, una più alta incidenza di infezioni cutanee da Herpes virus e un rischio più
elevato di allergie multiple e asma rispetto ai pazienti che hanno una DA ma non
presentano queste mutazioni. Il gene della FLG, localizzato sul cromosoma 1q21,
comprende tre esoni ed è localizzato nel complesso di differenziazione epidermica, un
gruppo di circa 60 geni coinvolti nella differenziazione epiteliale. Questo gene fa parte di
un sottogruppo di geni codificante sette proteine S100; queste proteine - filaggrina,
filaggrina-2, ornerina, tricoialina, tricoialina-like1, cornulina e repentina- formano una
comune organizzazione proteina-dominio avendo un dominio S100 legante il calcio
all’estremità N-terminale con una coda estesa e ripetitiva all’estremità C-terminale.
Fig.3 Organizzazione del gene della filaggrina (New England Journal of Medicine
365;14)
Le cellule dello strato granuloso della cute contengono granuli citoplasmatici densi
costituiti da profilaggrina che insieme ad altre componenti proteiche è necessaria alla
formazione delle squame, le cellule morte dello strato corneo, responsabili della
funzione di barriera della cute per impedire la perdita di acqua attraverso la superficie
cutanea del corpo e per ostacolare l’ingresso di sostanze estranee (patogeni, antigeni,
allergeni e sostanze chimiche irritanti) dall’ambiente esterno. Sono stati condotti molti
studi che hanno dimostrato come il deficit della filaggrina o una sua alterazione
qualitativa possano contribuire alla patogenesi della DA attraverso una varietà di
cambiamenti specifici strutturali, biofisici, funzionali e avere un ruolo importante nella
differenziazione dell’epidermide come il fallimento della funzione di barriera della cute.
È stato evidenziato che mutazioni a carico del gene della FLG (R501X, 2282del4,
polimorfismi a carico dell’inibitore protesico quale SPINK5, ecc.) inducano l’insorgenza
del fenotipo “flaky tail-mice” (“coda a scaglie”) consistente nella presenza di uno strato
corneo facilmente attraversabile da apteni che, determinando un eccessivo stimolo
infiammatorio, inducono una dermatosi grave che determina un ulteriore danno di
barriera legato a un’espressione linfocitaria del tipo Th2 (3).
periferico addizionato all’anticoagulante EDTA e conservato a -20°C. Per ogni bambino
è stato ottenuto un consenso informato dettagliato specifico per questo protocollo di
ricerca. Il DNA genomico è stato ottenuto utilizzando un protocollo modificato per
estrazione di acidi nucleici basato sul metodo del fenolo-cloroformio. L’analisi
molecolare delle mutazioni R501X e 2282del4 del gene della FLG è stata eseguita
allestendo 2 reazioni di PCR, usando 2 coppie di primers, FLG1-3 (2)F:5’CACGGAAAGGCTGGGCTGA-3’ e FLG 1-3 (1)R:5’- GACCCCGATGATTGTTCCTGT-3’,
FLG_2282del4seqF:5’-CAGTCAGCAGACAGCTCCAG-3’ e FLG_2282del4seqR:5’GCTTCATGGTGATGCGACCA-3’ per amplificare rispettivamente i frammenti con
all’interno le mutazioni R501X ed 2282del4. Le reazioni sono state effettuate in un
termociclatore Gene Amp 9700 (Applied Biosystems) in un volume totale di 25 µL con
100 ng di DNA genomico, 2.5 µL di Gold Buffer, 2 mM MgCl2, 0.75 unità di AmpliTaq
Gold polimerasi e acqua bidistillata a volume. Dopo una denaturazione iniziale di 5
minuti a 95°C, i cicli successivi prevedono 94°C (30 secondi), 58-61°C (30 secondi),
72°C (30-45 secondi) ripetuto per 30 cicli. Successivamente i campioni sono stati
verificati si gel d’agarosio all’1, 8% caricando 3 µl di amplificato per controllare l’esatta
amplificazione dei frammenti desiderati. I due frammenti amplificati con all’interno il sito
mutazionale sono stati sottoposti al sequenziamento diretto. Per tale metodica, il DNA è
stato amplificato in un volume di reazione di PCR di 50 µl, con i primer relativi allo
specifico amplicone analizzato.
I prodotti della reazione di PCR, dopo controllo su gel d’agarosio all’1.8%, sono stati
purificati con il kit commerciale “Montage PCR µ96 Plate“ (MILLIPORE) per eliminare
eventuali nucleotidi non incorporati nella reazione, i sali, i primers, e i dimeri di primers,
al fine di evitare interferenze nella successiva reazione di sequenza. Gli amplificati
purificati sono stati ricontrollati su gel d’agarosio all’1.8% per valutare la concentrazione
del purificato. Gli amplificati sono stati sequenziati in entrambe le direzioni mediante il kit
commerciale BigDye Terminator (Applied Biosistem) che fornisce i substrati per
effettuare una reazione di sequenza con il metodo di Sanger. Sono state preparate due
reazioni separate, una per ciascun primer (forward e reverse). Per ogni reazione sono
stati utilizzati quantità variabili di DNA a seconda della concentrazione del purificato, 10
pmol/µl di primer, 1 µl di mix di sequenza BigDye e 1 µl di buffer di sequenza.
La reazione è stata eseguita con un termociclatore Gene Amp PCR System 9700
(Perkin Elmer, Applied Biosystems, Foster City, CA) in tre fasi successive: 1)
denaturazione a 96°C per 10’’; 2) annealing a temperatura specifica per 5”; 3)
estensione a 60°C per 4 minuti. Le tre fasi sono state ripetute 30 volte e si sono
concluse con una fase finale a 4°C per 5’. La reazione di sequenza è stata purificata
per eliminare i primers e i nucleotidi liberi fluorescenti non incorporati nella reazione che
altrimenti interferirebbero con la lettura al sequenziatore automatico. I campioni sono
stati analizzati mediante elettroforesi capillare utilizzando il sequenziatore capillare ABI
PRISM 3130 (Perkin Elmer Applied Biosystems, Foster City, CA). Gli elettroferogrammi
ottenuti sono stati analizzati al software “Chromas” per la lettura delle sequenze e
l’eventuale caratterizzazione della mutazione riscontrata.
Risultati
Relativamente alla mutazione R501X, lo screening del gene della FLG ha consentito di
identificarne la presenza nel 13, 2% dei pazienti con DA (12/91 probandi) e nel 1, 7%
dei bambini sani del gruppo di controllo (1/60 controlli).
Fig.4 Filaggrin Deficiency and Possible mechanisms of Disease modified (New
England Journal of Medicine 365;14)
Studio sperimentale: pazienti e metodi
Nel nostro studio ci siamo proposti di valutare la presenza delle mutazione R501X e
della delezione 2282del4 del gene della filaggrina in un gruppo di bambini affetti da DA.
A tal fine sono stati reclutati 91 bambini affetti da DA (53 femmine e 38 maschi, età
media 7 anni +/- 4.9) e 60 bambini sani dello stesso range di età presso l’UOC di
Genetica ed Immunologia Pediatrica del Policlinico di Messina. Dopo la compilazione di
una scheda di arruolamento ogni bambino è stato sottoposto a visita medica ed a
prelievo ematochimico, previo consenso informato. I pazienti selezionati non
dimostravano alcuna patologia al di fuori delle allergie, né risultavano sottoposti a
trattamenti con corticosteroidi. Tutti i pazienti affetti da DA presentavano prick test
positivo per D. pteronyssinus. Il grado di severità è stato valutato mediante lo Scoring
Atopic Dermatitis Index (SCORAD). Lo SCORAD basato sull'obiettività clinica della DA
(segni clinici e l'estensione della malattia, punteggio totale 83) è utilizzato per
classificare il grado di gravità della DA in lieve (<15), moderato (15-40) o grave (> 40). I
bambini con DA sono stati quindi suddivisi in 3 sottogruppi in relazione alla gravità della
DA:
1. 30 bambini con DA lieve (SCORAD medio 26)
2. 30 bambini con DA moderata (SCORAD medio 67)
3. 31 bambini con DA severa (SCORAD medio 82)
I dati statistici sono stati elaborati con l’ausilio del t student e sono stati sintetizzati
indicando il valore della media e della deviazione standard per ogni parametro indicato.
Sono stati ritenuti significativi valori in cui la variabile P calcolata dal confronto tra il
gruppo controllo e quello dei pazienti per ciascun dosaggio assumeva valori inferiori a 0,
01 (P< 0, 001).
Dopo il prelievo ematico il DNA genomico è stato estratto a partire da 2-10 ml di sangue
Fig. 5
Da questi primi risultati sulla mutazione R501X nel confronto tra il gruppo dei pazienti
con DA e il gruppo dei controlli è emersa una p= 0.016 statisticamente significativa e
una Odds Ratio (OR) di 8.962 (intervallo di confidenza del 95% compreso tra 1.441 e
54.842) ed un Rischio Relativo (RR) di 1.612 (intervallo di confidenza del 95%
compreso tra 1.141 e 1.741).
Successivamente è stata studiata la frequenza della mutazione R501X nei tre gruppi
clinici suddivisi in base al fenotipo al fine di valutare l’eventuale correlazione con la
severità della DA.
Fig.6
Nel primo gruppo costituito da 30 probandi con fenotipo lieve è stato riscontrato 1/30
probando con mutazione R501X (3, 3% dei casi).
Nel secondo gruppo costituito da 30 probandi con fenotipo moderato sono stati
riscontrati 4/30 probandi con mutazione R501X (13, 3% dei casi).
Nel terzo gruppo costituito da 31 probandi con fenotipo severo sono stati riscontrati 7/31
probandi con mutazione R501X (22, 6% dei casi).
Dall’analisi genetica della mutazione R501X è quindi emersa, nel confronto tra i tre
gradi di severità della DA, una p=0, 08 statisticamente significativa.
Relativamente alla delezione 2282del4, lo screening del gene della FLG ha consentito
di identificarne la presenza nel 12, 1% dei pazienti con DA (11/91 probandi) e nel 3, 3%
dei bambini sani del gruppo di controllo (2/60 controlli).
Fig.7
Da questi primi risultati sulla delezione 2282del4 nel confronto tra il gruppo dei pazienti
con DA e il gruppo dei controlli è emersa una p= 0.077 statisticamente significativa e
una Odds Ratio (OR) di 3.988 (intervallo di confidenza del 95% compreso tra 0.947 e
16.571) ed un Rischio Relativo (RR) di 1.460 (intervallo di confidenza del 95%
compreso tra 0.978 e 1.680).
Successivamente è stata studiata la frequenza della delezione 2282del4 nei tre gruppi
clinici suddivisi in base al fenotipo al fine di valutare l’eventuale correlazione con la
severità della DA.
Fig.8
Nel primo gruppo costituito da 30 probandi con fenotipo lieve è stato riscontrato 1/30
probando con mutazione 2282del4 (3, 3% dei casi).
Nel secondo gruppo costituito da 30 probandi con fenotipo moderato sono stati
riscontrati 3/30 probandi con mutazione 2282del4 (10, 0% dei casi).
Nel terzo gruppo costituito da 31 probandi con fenotipo severo sono stati riscontrati 7/31
probandi con mutazione 2282del4 (22, 6% dei casi).
Dall’analisi genetica della delezione 2282del4 è quindi emersa, nel confronto tra i tre
gradi di severità della DA, una p=0, 06 statisticamente significativa.
Discussione
La Dermatite Atopica (DA) rappresenta oggi uno dei principali problemi di sanità
pubblica. Una maggiore comprensione dei meccanismi patogenetici responsabili di
questa malattia e dei polimorfismi dei geni implicati nella predisposizione ereditaria è
importante per garantire una diagnosi corretta e l’attuazione di strategie preventive e
terapeutiche innovative ed efficaci. In particolare il nostro studio rappresenta un’ulteriore
conferma del ruolo fondamentale svolto dalla genetica nella patogenesi di questa
malattia e della possibilità, oggi sempre più evidente, di correlare il genotipo di un
individuo al suo fenotipo e individuare quei particolari polimorfismi genici associati a
manifestazioni cliniche più severe. A tal proposito è emerso infatti come più alta sia la
percentuale (22, 6%) di bambini con la mutazione R501X nel gruppo di pazienti che
presentano un fenotipo severo di DA (SCORAD medio di 82), se paragonata alla
percentuale di bambini che presentano la stessa mutazione ma hanno una DA di grado
lieve (3, 3%) o moderato (13, 3%). Risultati simili sono stati ottenuti nel corso dell’analisi
genetica della mutazione 2282del4 grazie alla quale è possibile mettere in evidenza
come più alta sia la percentuale (22, 6%) di bambini con la mutazione 2282del4 nel
gruppo di pazienti che presentano un fenotipo severo di DA (SCORAD medio di 82), se
paragonata alla percentuale di bambini che presentano la stessa mutazione ma hanno
una DA di grado lieve (3, 3%) o moderato (10, 0%). Questi risultati ci inducono a
riflettere sulla possibilità che la mutazione genetica R501X e quella 2282del4 individuate
in questi bambini siano maggiormente correlate a un fenotipo più severo di DA e quindi
a manifestazioni cliniche più gravi della malattia con una peggiore qualità di vita di
questi pazienti e scarsa risposta alla terapia farmacologica. A tale scopo, cinque unità
operative (U.O.) con sede a Messina, Catania, Napoli, Genova e Pavia, ognuna con
competenze ben consolidate in specifici settori scientifici e ampiamente riconosciute a
livello internazionale, portano avanti da più di un anno un progetto di ricerca
coinvolgente il gene della filaggrina per definire la ricorrenza delle due mutazioni e
riuscire a delineare la possibilità di una correlazione genotipo-fenotipo nel contesto della
patologia atopica. Studi in tutta Europa hanno inoltre rilevato come le due mutazioni in
questione siano le più frequenti tra i soggetti con DA del nostro continente con
percentuali che variano tra il 33% in Irlanda, 27% in Gran Bretagna, 9, 5% in Svezia e
Danimarca, 9% in Germania e 3% in Francia (5). Pochi sono però gli studi effettuati su
gruppi di pazienti di altre etnie nonostante ci consentano di dire che, nelle etnie diverse
dalla razza caucasica, la frequenza delle mutazioni R501X e 2282del4 sia piuttosto
bassa e lontana dai valori che contraddistinguono l’Europa e in particolare alcuni Paesi.
La ricerca centrata sulla correlazione genotipo-fenotipo è comunque tutt’oggi in itinere:
ulteriori studi su nuovi polimorfismi genici e sulle loro ripercussioni cliniche
contribuiranno a fare sempre più luce sulla DA e potranno fornire indicazioni al fine di
ricavare strategie terapeutiche innovative per questa malattia.
Bibliografia
(1) C. Cuppari, R. Gallizzi, M. Sturiale, S. Manti, C. Grosso, L. Colavita, L. Grasso, E.
Moschella, V. Ferraù, P. Vicchio, M. Miraglia Del Giudice, M. La Rosa, C. Salpietro, G.
Marseglia; Livelli serici di IL-18 in bambini con dermatite atopica: dati preliminari-Rigip
Anno II Numero 3-luglio 2010.
(2) Chikako Kiyohara, Keiko Tanaka and Yoshihiro Miyake ‘’Genetic Susceptibility to
Atopic Dermatitis’’ Allergology International 2008;57:39-56.
(3) Alan D. Irvine, M.D., W.H. Irwin McLean, Ph.D., D.Sc., and Donald Y.M. Leung,
M.D., Ph.D. ’’ Filaggrin Mutations Associated with Skin and Allergic Diseases’’. The New
England Journal of Medicine, 2011, 365;14.
(4) T. Schimming, Q. Parwez, E. Petrasch-parwez, M. Nothangel, J. T. Epplen, S.
Hoffjan; ‘’Association of toll-interacting protein gene polymorphisms with atopic
dermatitis’’ BMC Dermatology 2007, 7:3.
(5) H. Baurecht, MSc, Alan D. Irvine, N. Novak, MD, T. Illig, PhD, B. Buhler, MSc, J.
Ring, MD, S. Wagenpfeil, S. Weidinger; ‘’ Toward a major risk factor for atopic eczema:
Meta-analysis of filaggrin polymorphism data’’ J Allergy Clin Immunol 2007;120:1406-12.
(6) Kathleen C. Barnes, PhD; ‘’ An update on the genetics of atopic dermatitis:
Scratching the surface in 2009’’ J Allergy Clin Immunol January 2010; Volume 125,
Number 1.
(7) Ercan H, Ispir T et al. Predictors of atopic dermatitis phenotypes and severity: Roles
of serum immunoglobulins and filaggrin gene mutation R501X. Allergol Immunopathol
(Madr). 2012 Sep 15
(8) Margolis DJ, Apter AJ et al. The persistence of atopic dermatitis and filaggrin (FLG)
mutations in a US longitudinal cohort. J Allergy Clin Immunol. 2012 Aug 27.
(9) Naomi Takahashi, Mitsuteru Akahoshi, Akira Matsuda, Kouji Ebe, Naoko Inomata,
Kazuhiko Obara, Tomomitsu Hirota, Kazuko Nakashima, Makiko Shimizu, Mayumi
Tamari, Satoru Doi, Akihiko Miyatake, Tadao Enomoto, Hitoshi Nakashima, Zenro
Ikezawa and Taro Shirakawa: ‘’Association of the IL12RB1 promoter polymorphisms
with increased risk of atopic dermatitis and other allergic phenotypes’’. Human Molecular
Genetics, 2005, Vol. 14, No. 21 3149–3159.
(10) Natalija Novak, Susanne Kruse, Jana Potreck, Laura Maintz, Claudia Jenneck,
Stephan Weidinger,
Rolf Fimmers and Thomas Bieber: ‘’Single nucleotide
polimorphisms of the Il-18 gene are associated with atopic eczema’’. J. Allergy Clinical
Immunology Volume 115, number 4.
(11) Grainne M. O’Regan, Aileen Sandilands, W. H. Irwin McLeanand Alan D. Irvine:
‘’Filaggrin in atopic dermatitis’’. October 2008
(12) John W. Holloway, Ian A. Yang and Stephen T. Holgate, FMed Sci: ‘’Genetic of
allergy desease’’. J. Allergy Clinical Immunology, February 2010.
(13) Cecilia Sinibaldi ‘’Analisi genetica della malattie complesse: loci comuni e loci
specifici’’ dottorato di ricerca a.a. 2008/09.
(14) Yin-Hsiu Chien & Wuh-Liang Hwu & Bor-Luen Chiang. ‘’The Genetics of Atopic
Dermatitis’’. Clinic Rev Allerg Immunol (2007) 33:178–190.
(15) Peter M. Elias and Matthias Schmuth: ‘’Abnormal skin barrier in the
etiopathogenesis of atopic dermatitis’’. Curr Opin Allergy Clin Immunol. 2009 October ; 9
(5): 437–446.
(16) Jerzy Jarzab, Barbara Filipowska, Jadwiga Zebracka, Małgorzata Kowalska,
Andrzej Bozek, Regina Rachowska, Elzbieta Gubala, Alicja Grzanka, Ewa Hadas and
Barbara Jarzab. ‘’Locus 1q21 Gene Expression Changes in Atopic Dermatitis Skin
Lesions: Deregulation of Small Proline-Rich Region 1’’. Int Arch Allergy Immunol
2010;151:28–37.
(17) John W. Holloway, Ian A. Yang and Stephen T. Holgate, FMed Sci. ‘’Genetics of
allergic disease’’. J. Allergology clinical Immunology volume 125, number 2.
(18) Joshua A. Boyce, Fred Finkelman, William T. Shearer and Donata Vercelli. ‘’An
update on the genetics of atopic dermatitis: Scratching the surface in 2009’’. J.
Allergology Vlinical Immunology volume 125, number 1.
(19) Tobias T Schimming, Qumar Parwez, Elisabeth Petrasch-Parwez, Michael
Nothnagel, Joerg T Epplen and Sabine Hoffjan: ‘’Association of toll-interacting protein
gene polymorphisms with atopic dermatitis’’. BMC Dermatology 2007, 7:3.
(20) Winge MC, Bilcha KD et al Novel filaggrin mutation but no other loss-of-function
variants found in Ethiopian patients with atopic dermatitis. Br J Dermatol 2011 Nov;165
(5).
(21) Thyssen JP, Carlsen BC et al. Individuals who are homozygous for the 2282del4
and R501X filaggrin null mutations do not always develop dermatitis and complete
long-term remission is possible. J Eur Acad Dermatol 2012 Mar;26 (3):386-9.
(22) Gruber R, Janecke AR et al. Lower prevalence of common filaggrin mutations in a
community sample of atopiceczema: is disease severity important? Wien Klin
Wochenschr. 2010 Oct;122 (19-20):551-7.
(23) Brown SJ, Relton CL, et al. Filaggrin haploinsufficiency is highly penetrant and is
associated with increased severity of eczema: further delineation of the skin phenotype
in a prospective epidemiological study of 792 school children’. Br J Dermatol 2009
Oct;161 (4):884-9.
(24) De Jongh CM, Khrenova L, et al. Loss-of-function polymorphisms in the filaggrin
gene are associated with an increased susceptibility to chronic irritant contact dermatitis:
a case-control study’. Br J Dermatol 2008 Sep;159 (3):621-7.
(25) Monar N, Cookson WO et al. Filaggrin mutations in children with severe atopic
dermatitis. Invest Dermatol. 2007 Jul;127 (7):1667-72.
www.geneticapediatrica.it trimestrale di divulgazione scientifica dell'Associazione Pediatrica di Immunologia e Genetica
Legge 7 marzo 2001, n. 62 - Registro della Stampa Tribunale di Messina n. 3/09 - 11 maggio 2009
Direttore scientifico
Carmelo Salpietro - Direttore responsabile
Giuseppe Micali - Segreteria redazione
Basilia Piraino - Piera Vicchio
Direzione-Redazione: UOC Genetica e Immunologia Pediatrica - AOU Policlicnico Messina