aa 2013-14
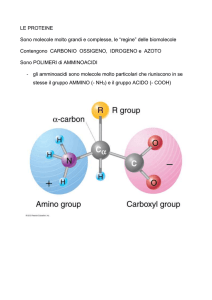
Proteine
• Biopolimeri degli α-amino acidi.
• Amino acidi sono uniti attraverso il legame peptidico.
• Alcune funzioni:
Struttura (collagene, cheratina ecc.)
Enzimi (maltasi, deidrogenasi ecc)
Trasporto (albumine, emoglobina ecc.)
Protezione (tossine, allergeni ecc.)
Ormoni (insulina, ormoni della crescita ecc.)
Struttura delle Proteine
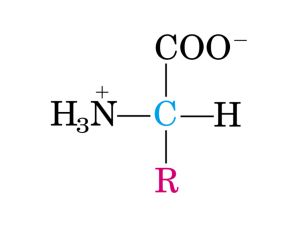
α Amminoacidi
COOH
H2N
Cα
R
H
-NH2 sul carbonio adiacente al -COOH.
Glicina, NH2-CH2-COOH, è il più semplice.
Con -R come catena laterale, essi sono chirali.
La maggior parte degli amminoacidi naturali
sono L-amminoacidi.
1
aa 2013-14
Stereochimica degli α-Amminoacidi
Amminoacidi costituenti le proteine
• Ventidue α-amminoacidi standard.
• Differiscono nella natura della catena laterale:
-H o alchile
contiene un –OH
Contiene lo zolfo
Contiene un azoto non basico.
Un gruppo –COOH addizionale
Un azoto basico
Amminoacidi Essenziali
•
•
•
•
Treonina (Thr)
Lisina (Lys)
Valina (Val)
Fenilalanina (Phe)
•
•
•
•
Triptofano (Trp)
Metionina (Met)
Leucina (Leu)
Isoleucina (Ile)
2
aa 2013-14
Proteine Complete
forniscono tutti gli amminoacidi essenziali.
esempi: quelle nella carne, pesce, latte, uova.
Le proteine nei vegetali sono generalmente incomplete.
I vegetariani devono mangiare molti tipi differenti di
vegetali, oppure supplire la dieta ad esempio con uova o
latte.
3
aa 2013-14
Aminoacidi Rari
4-idrossiprolina, 5-idrossilisina presente nel collagene.
Acido D-Glutammico nelle pareti cellulari dei batteri.
D-Serina
nei lombrichi.
Acido γ-amminobutirrico, è un neurotrasmettitore.
β-Alanina, costituente della vitamina acido pantotenico.
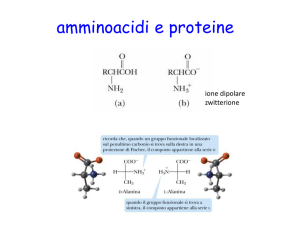
Zwitterione
Gli Amminoacidi esistono come ioni dipolari.
-COOH perde H+,mentre -NH2 acquista H+.
• La struttura effettiva dipende dal pH
Proprietà degli Amminoacidi
Punti di fusione alti, oltre i 200°C
Più solubili in acqua che in solventi non polari.
Un momento di dipolo più grande di quello di acidi o
ammine semplici.
Più acidi della maggior parte degli acidi carbossilici e
meno basici di molte ammine.
pKa = 10
O
_
+
H3N CH C O
R
pKb = 12
4
aa 2013-14
Struttura e pH
Punto isoelettrico
pH a cui gli amminoacidi esistono come zwitterione
(neutro).
Dipende dalla struttura della catena laterale.
Amminoacidi acidi, pI ~ 3.
Amminoacidi basici, pI ~ 9.
Amminoacidi neutri, pI ~ 5-6.
Elettroforesi
Proteine cariche
negativamente si muovono
verso lanodo
Posizione della
miscela originale
Proteine cariche
positivamente si muovono
verso il catodo
5
aa 2013-14
alanina
OH
pKa=2.35
O
H
O
+
N
H3C
-
N
H3C
H
-
H
O
+
H
O
pKa=9.87
H
O
H
N
H
H3C
H
pI = 6.11
arginina
H
O
O
-
O
+
+
N
H
N
H
H
N
pKa=9.04
H
NH
H
H
pKa=12.48
NH
NH
H2N
+
H
O
H
H
H2N
N
-
O
N
pKa=2.01
NH
H2N
+
-
O
H
O
H
O
N
H
+
H
N
H
H2N
N
H
H
H
pI = 10.76
6
aa 2013-14
lisina
H
O
O
+
N
+
N
pKa=2.18
H
H
pKa=8.95
-
O
H
N
pKa=10.53
H
H
+
H
O
H
N
+
N
N
+
-
O
H
H
N
H
O
H
H
H
H
-
O
H
O
H
H
H
N
H
H
H
pI = 9.74
istidina
OH
O
H
O
-
O
H
O
+
+
H
N
H
H
H
H
H
N
pKa=9.18
H
H
+
+
N
-
O
N
pKa=6.10
H
N
N
O
H
+
H
N
pKa=1.77
H
-
O
N
H
H
N
N
H
N
N
pI = 7.64
Acido aspartico
O
H
O
H
O
+
N
H
N
pKa=2.10
H
O
O
+
O
O
+
N
H pK =3.86
a
-
O
H
H
N
pKa=9.82
H
H
O
H
-
H
O
H
O
H
-
H
O
O
O
O
-
O
-
pI = 2.98
7
aa 2013-14
cisteina
OH
O
H
O
+
N
-
O
H
O
+
N
H
-
H
pKa=2.05
+
N
pKa=8.00
-
O
H
H
N
pKa=10.25
H
H
SH
O
H
O
H
H
-
-
S
SH
S
pI = 5.02
Tirosina
O
OH
O
H
+
N
O
H
pKa=2.20
-
O
H
H
N
OH
O
pKa=9.11
H
pKa=10.07
N
OH
-
O
H
H
H
-
O
+
OH
H
N
H
O
-
pI = 5.63
Equazione di Henderson-Hasselbalch
pH = pK a + log
[ base coniugata ]
[acido debole]
8
aa 2013-14
Alcune
Sintesi di laboratorio degli amminoacidi
Amminazione riduttiva
Sintesi da a aloacidi
Sintesi di Gabriel-estere malonico
sintesi di Strecker
Amminazionone Riduttiva
Questo metodo di sintesi è biomimetico, cioè mima il
processo biologico di sintesi.
Si fa reagire un α-chetoacido con ammoniaca, poi si
riduce limina con H2/Pd.
Nel processo di laboratorio si ottiene una miscela
racemica!
O
R C COOH
NH3
N H
_
+
R C COO NH4
H2
Pd
NH2
_
R C COO
H
Sintesi da α-aloacidi
La reazione di Hell-Volhard-Zelinsky permette di
sintetizzare gli α-bromoacidi.
Il bromo può essere sostituito con un gruppo
ammino facendo reagire lα-bromoacido con un
eccesso di ammoniaca.
Si ottiene una miscela racemica.
9
aa 2013-14
Sintesi di Gabriel-Estere Malonico
Lammino gruppo viene protetto come ammide.
Il gruppo carbossilico viene protetto come estere.
La posizione α è attivata ulteriormente dalla
presenza del gruppo estereo temporaneo
Sintesi di Strecker
La prima sintesi nota di un amminoacido risale al 1850.
La reazione di unaldeide con NH3 dà unimina.
Lo ione cianuro attacca limmina protonata.
Il risultato è un α-ammino nitrile che viene idrolizzato
ad acido carbossilico.
Meccanismo della sintesi di Strecker
Stadio 1: formazione
dellimmina
Stadio 2: attacco
dello ione cianuro
Lidrolisi del nitrile porta allamminoacido.
10
aa 2013-14
Risoluzione degli Amminoacidi
• solitamente, solo lenantiomero L- e biologicamente
attivo.
• Si converte lamminoacido in un sale, utilizzando un
acido o una base chirale. Si ottiene cosi la miscela
dei due Sali diastereomerici che possono essere
separati
• Si utilizza un enzima come ldeacilasi che reagisce
solo con uno dei due enantiomeri.
Risoluzione di racemati: alcuni esempi
Formazione di esteri diasteromerici
R
+
O
−
H3N CH C O
+ R*
OH
H+
+
R
O
H3N CH C OR* + H2O
Risoluzione enzimatica
Struttura dei Peptidi
• Il legame peptidico è un legame ammidico.
• Le ammidi sono composti molto stabili e neutri.
11
aa 2013-14
Alternanza dei piani in un polipeptide
Bradichinina
• Un oligopeptide non proteico.
• Convenzionalmente le strutture dei peptidi
vengono disegnate con lamminoacido N-terminale
sulla sinistra.
• Il nome dei peptidi è costruito nominando tutti gli
amminoacidi che lo compongono a partire
dallamminoacido N-terminale: arginilprolilprolil……
arginina.
Legame disolfuro
La cisteina può formare ponti disolfuro.
12
aa 2013-14
Determinazione della struttura di un peptide
•
•
•
•
•
Rottura dei ponti disolfuro
Determinazione della composizione in amminoacidi
Sequenziazione dallN-terminale
Analisi dei residui C-terminali
Idrolisi parziale
Il primo passaggio è la rottura del ponte disolfuro
Il passaggio successivo è la determinazione del tipo e
del numero degli amminoacidi contenuti
• Si Separano le catene polipeptidiche.
• Si mette a bollire con HCl 6 M per 24 ore.
• Gli amminoacidi si separano con un analizzatore.
13
aa 2013-14
Reazione con la Ninidrina
• Utilizzata per visualizzare le macchie o le bande degli
amminoacidi separati per cromatografia o
elettroforesi.
• Colore porpora intenso formato con tutti gli
amminoacidi
La prolina e lidrossiprolina danno una colorazione
gialla.
14
aa 2013-14
Sequenziazione dallN-terminale
• Degradazione di Edman: reazione con fenil
isotiocianato seguita dallidrolisi rimuove
lamminoacido N-terminale.
• La feniltioidantoina viene identificata per via
gascromatografica.
• È utilizzata con buoni risultati per polipeptidi con
meno di 30 amminoacidi.
Esempio di degradazione di Edman
per ossitocina bovina
Sequenziazione del residuo C terminale
• Lenzima carbossipeptidasi rompe selettivamente
il C-terminale.
• Riduzione della funzione carbossilica a funzione
alcolica
• Idrazinolisi del peptide.
15
aa 2013-14
Idrolisi parziale
Formazione di un legame ammidico
16
aa 2013-14
Sintesi di un dipeptide
Sintesi di un peptide in fase liquida
• Dapprima, si protegge il gruppo N terminale.
• Si attiva il gruppo carbossilico del medesimo
amminoacido.
• Si fa avvenire la reazione con lamminoacido
successivo.
• Per sintetizzare un polipeptide si ripete
loperazione di attivazione ed accoppiamento.
• Terminata la sintesi si rimuove il gruppo protettivo.
Sintesi di un tripeptide
17
aa 2013-14
Sintesi di un tripeptide
Sintesi di peptidi su fase solida
• La catena polipeptidica da sintetizzare è ancorata ad
un letto di piccoli granuli di polistirene.
• Gli intermedi non devono essere purificati.
• I reagenti in eccesso vengono lavati via con un idoneo
solvente.
• Il processo può essere automatizzato.
• Permette di costruire peptidi più lunghi rispetto alla
sintesi in soluzione.
Sintesi automatica di Merrifield
1° Stadio
18
aa 2013-14
Sintesi automatica di Merrifield
2° Stadio
Sintesi automatica di Merrifield
3° Stadio
Sintesi automatica di Merrifield
4° Stadio
19
aa 2013-14
Sintesi automatica di Merrifield
5° Stadio
Classificazione delle proteine
• Semplici: idrolizzano solo ad amminoacidi.
• Coniugate: sono legate ad un gruppo non proteico
come zuccheri, acidi nucleici, lipidi.
• Fibrose: lunga e filamentosa, insolubile in acqua,
funzione strutturale.
• Globulari: ripiegata in forma sferica, funzione
enzimatica, ormonale, o di trasporto ormonale.
Proteine fibrose
Proteine globulari
20
aa 2013-14
Legame disolfuro
Schema di una catena polipeptidica con legami a ponte
disolfuro intercatena ed intracatena
Note: i ponti disolfuro intracatena legano insieme due
parti dello stesso polipeptide, mentre il legame
intercatena unisce due catene di polipeptidi differenti.
Formazione del legame disolfuro
Il ponte disolfuro contribuisce alla forma della proteina
21
aa 2013-14
Livelli delle struttura delle proteine
• Primaria: la sequenza degli amminoacidi nella
catena ed i legami disolfuro.
• Secondaria: struttura formata dai legami
idrogeno. Esempio è lα-elica e il foglietto piegato
(struttura β ).
• Terziaria: la conformazione completa 3-D.
• Quaternaria: lassociazione di due o più catene
peptidiche per formare le proteine.
Livelli delle struttura delle proteine
Alfa elica
Ogni ossigeno carbonilico può formare un
legame idrogeno con lN-H dellavvolgimento
successivo dellelica.
22
aa 2013-14
Vista laterale di un
segmento di unα
elica di una proteina
Vista dallalto di un
segmento di unα
elica di una proteina
Foglietti piegati (struttura β )
Ogni ossigeno carbonilico forma un
legame idrogeno con un N-H di una
catena peptidica adiacente.
23
aa 2013-14
STRUTTURA DELLE PROTEINE
Struttura primaria: sequenza aminoacidica
Struttura secondaria: ripiegamento della catena
lungo un asse
Cheratine: α- e β-cheratine
Collageno: tessuto connettivo
Struttura terziaria: ripiegamento tridimensionale
delle catene polipeptidiche.
Struttura terziaria
Spesso determinata per diffrazione ai raggi X
Esempi di proteine globulari sono mioglobina,
lisozima, ribonucleasi, citocromo C
Caratteristiche comuni:
Ciascuna proteina ha una struttura caratteristica
Ripiegamento compatto con poco spazio allinterno
Localizzazione allesterno di quasi tutti i gruppi idrofilici
Localizzazione allinterno dei gruppi idrofobici
Possono contenere tratti sia di α-elica sia di β-foglietto
24
aa 2013-14
Mioglobina, una proteina globulare
La sequenza determina la struttura
La denaturazione dà luogo a conformazioni casuali
della catena polipeptidica.
Alcune proteine denaturate possono riacquistare la
loro attività biologica
Esempio classico la ribonucleasi.
Denaturazione e
rinaturazione della
ribonucleasi
25
aa 2013-14
Stabilizzazione della struttura terziaria
Legame idrogeno tra gruppi peptidici
Legame idrogeno tra residui R polari
Interazioni idrofobiche
Legame idrogeno tra residui R carichi
Linterazione predominante è linterazione idrofobica
Le interazioni
Struttura quaternaria
Caratteristica delle proteine oligomeriche
La struttura quaternaria designa linterazione tra
le varie unità della proteina.
Alcune proteine che hanno struttura quaternaria
dopo la denaturazione possono essere rinaturate.
26
aa 2013-14
Sommario delle Strutture
Alterazioni chimiche negli amminoacidi
Alterazioni chimiche negli amminoacidi
27
aa 2013-14
Alterazioni chimiche
negli amminoacidi
28