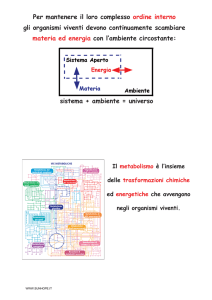
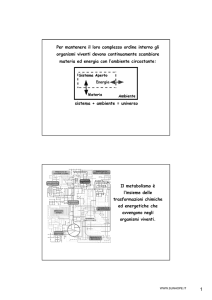
Scaricato da www.sunhope.it
ELEMENTI FONDAMENTALI PER LA REAZIONE DI
PCR
PCR=Polymerase Chain Reaction
(Reazione a Catena della Polimerasi)
Frammento di DNA contenente il segmento da amplificare
La PCR è stata ideata da K.B. Mullis nel 1983 in
California
E’ un procedimento molto semplice che consente di
ottenere un numero elevatissimo di copie di uno specifico
segmento di DNA
5’
3’
3’
5’
Coppia di inneschi (o primers) per la DNA Polimerasi: oligonucleotidi
lunghi 18-25 basi, di sequenza complementare a quelle fiancheggianti il
Primer 1
segmento di DNA da amplificare.
Primer 2
TAQ DNA POLIMERASI
5’
dNTP (N= A, C, G, T)
La PCR è una tecnica molto sensibile che consente di
rivelare la presenza del DNA o dell’RNA di interesse
WWW.SUNHOPE.IT
partendo da quantità minime
di materiale biologico
2.
3.
DENATURAZIONE: il DNA
viene denaturato a 94°C per
separare fra loro i due filamenti
che formano la doppia elica.
ANNEALING: la temperatura
scende a 30-65°C (valore scelto in
funzione della composizione in
basi e della lunghezza degli
inneschi)
per
consentire
l’appaiamento fra gli inneschi e le
due eliche di DNA ad essi
complementari
ESTENSIONE: La temperatura
sale a 72°C e la Taq Polimerasi
sintetizza i nuovi filamenti di DNA
partendo dai due inneschi in
direzione 5’→
→3’utilizzando come
WWW.SUNHOPE.IT
stampo le due eliche di DNA.
WWW.SUNHOPE.IT
3’
3’
3’
5’
5’
Mg2+
Apparecchio per PCR (es. Thermocycler)
WWW.SUNHOPE.IT
CARATTERISTICHE DEI PRIMERS
UN CICLO DI PCR
1.
5’
3’
5’
• Lunghezza compresa tra 14-40 basi
3’
3’
5’
• Contenuto di G+C pari al 40-75 %
5’
3’
5’
3’
3’
• Estremità 3’ end deve contenere un codone
conservato (Trp, Met)
5’
3’
5’
• Evitare la presenza della 3 base ballerina
5’
5’
3’
5’
3’
5’
3’
• Valori di Tm simili.
5’
3’5’
5’3’
3’
5’
WWW.SUNHOPE.IT
• Evitare sequenze
interne complementari
1
Scaricato da www.sunhope.it
FORMAZIONE DI DIMERI E DI LOOP
5’
ACTG
CALCOLO DELLA Tm
CAGT 3’
Tm = 81.5 + 16.6 (log 10 [ J+] + 0.41 (% G+C) - (600/l)
5’
[ J+]= concentrazione di cationi monovalenti
ACTG
3’ TGAC
5’
ACTG
Per esempio:
PCR buffer = 60 mM (J+)
Primers 20 mer
55% G+C
CAGT 3’
3’ TGAC
GTCA
5’
Tm = 81.5 - 20.3 + 22.6 -30 = 53.8
Ta (annealing) = Tm (3 - 12 °C)
WWW.SUNHOPE.IT
POLYMERASE CHAIN REACTION
WWW.SUNHOPE.IT
WWW.SUNHOPE.IT
WWW.SUNHOPE.IT
PCR: AMPLIFICAZIONE ESPONENZIALE DEL DNA
STAMPO
WWW.SUNHOPE.IT
2
Scaricato da www.sunhope.it
QUANTIZZAZIONE DEI PRODOTTI DI PCR
ANALISI SU GEL DI AGAROSIO DI
PRODOTTI DI PCR
1
2
3
4
5
6
MW
β-actina
500 bp
Questa
è la rappresentazione schematica dell’aumento dei prodotti di
amplificazione in funzione del numero di cicli di PCR.
In questo caso, tra il 15 ed il 22mo ciclo ersiste una relazione lineare tra il
numero di cicli ed il prodotto di amplificazione ottenuto. Nei cicli
successivi viene raggiunto il “plateau”.
WWW.SUNHOPE.IT
PRINCIPALI APPLICAZIONI DELLA PCR
Identificazione di agenti patogeni nell’uomo, negli animali
o negli alimenti (batteri e virus)
Identificazione di organismi geneticamente modificati
(OGM)
Analisi del DNA in medicina legale (es. Test di paternità)
Diagnosi pre-natale di malattie genetiche (es. Anemia
falciforme, Distrofia di Duchenne, talassemia)
Diagnosi di malattie tumorali (es. Linfoma)
Clonaggio e sequenziamento di frammenti di DNA
WWW.SUNHOPE.IT
WWW.SUNHOPE.IT
AT1
306 bp
WWW.SUNHOPE.IT
PRINCIPALI TIPI DI PCR a)
RT-PCR: serve a valutare l’espressione di un gene
tramite l’amplificazione dell’mRNA da esso trascritto
PCR ASIMMETRICA: serve ad ottenere DNA a
singolo filamento e si effettua utilizzando uno dei due
inneschi in quantità molto ridotta rispetto all’altro
PCR COMPETITIVA: serve a valutare la
concentrazione iniziale di DNA o RNA attraverso
l’amplificazione in contemporanea con un DNA
standard, aggiunto in quantità nota alla miscela di
reazione. Il campione standard si amplifica con la
stessa coppia di inneschi del DNA di interesse.
WWW.SUNHOPE.IT
3
Scaricato da www.sunhope.it
PRINCIPALI TIPI DI PCR b)
NESTED PCR: serve ad ottenere una maggiore
specificità del prodotto di amplificazione utilizzando
in successione due coppie di inneschi, di cui una più
esterna e poi una più interna rispetto al segmento di
DNA di interesse
MULTIPLEX PCR: consente di amplificare
contemporaneamente diversi segmenti dello stesso
campione di DNA utilizzando coppie di inneschi
diversi (es. Distrofia duchenne)
PCR REAL TIME: consente di seguire in tempo reale
l’andamento della PCR utilizzando inneschi
fluorescenti e misurare le quantità assolute di
molecole di partenzaWWW.SUNHOPE.IT
MISURA DEL LIVELLO DI TRASCRIZIONE DI
UN GENE
Per misurare il livello di mRNA trascritto da un determinato
gene e quindi avere indicazioni sul suo livello di espressione (es.
tessuto, organo, batteri, cellule in coltura, etc.) è necessario:
Estrarre l’RNA totale dal campione biologico che si vuole
esaminare
Trasformare il pool di RNA (che conterrà anche l’mRNA
trascritto dal gene di interesse) in cDNA tramite la reazione di
trascrizione inversa
Evidenziare il cDNA di interesse tramite la reazione di PCR
WWW.SUNHOPE.IT
TRASCRIZIONE INVERSA
RT-PCR=REVERSE TRANSCRIPTASE PCR
La RT-PCR amplifica un RNA
convertendolo prima in cDNA (o
DNA complementare all’RNA)
tramite l’enzima trascrittasi
inversa, e poi amplificandolo
tramite PCR con inneschi
specifici per la sequenza di
interesse.
5’
AAAAA 3’
TTTTT 5’
RT
3’
TTTTT 5’
TAQ Pol
La reazione di TRASCRIZIONE INVERSA consente di produrre una molecola di cDNA
(DNA complementare) a partire da mRNA (RNA messaggero).
L’enzima che catalizza questa reazione è la trascrittasi inversa, una DNA polimerasi RNAdipendente.
Questo enzima è stato inizialmente identificato nei retrovirus (ad esempio il virus HIV).
Le due trascrittasi inverse più utilizzate in laboratorio sono la AMV (isolata dall’Avian
Myeloblastosis Virus) e la MoMLV (isolata dal Moloney Murine Leukemia Virus).
Il pool di cDNA ottenuto dalla reazione
di trascrizione inversa è utilizzato per misurare il
WWW.SUNHOPE.IT
livello di espressione di un gene mediante la tecnicaPCR
WWW.SUNHOPE.IT
La RT-PCR viene utilizzata, ad
esempio, per valutare il livello di
espressione
di
un
gene
misurando la quantità di mRNA
da esso trascritto.
5’
3’
3’
5’
La trascrittasi inversa viene
utilizzata dai retrovirus (o virus
WWW.SUNHOPE.IT
ad RNA, es HIV).
4