Vivere con una malattia rara
Dalla diagnosi alla presa in carico
18 maggio 2016
Nuovi strumenti diagnostici
Paola Grammatico
Il Laboratorio di Genetica medica nella
diagnosi delle malattie rare
Citogenetica classica
Citogenetica molecolare
Array-CGH
Il Laboratorio di Genetica medica nella diagnosi
delle malattie rare
Reverse dot blot
MLPA
Real time PCR
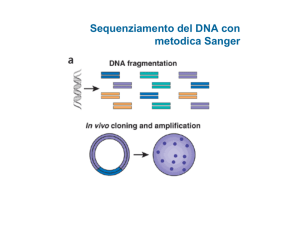
Sequenziamento: Metodo Sanger
Frederick Sanger: due premi Nobel in Chimica
(1958 e 1980)
Sequenziamento del DNA: Metodo Sanger
1987: primo sequenziatore automatico (Applied Biosystem ABI370)
500.000 pb/giorno output: 500 kb/die
Lunghezza massima del frammento: 600 basi
Analisi di un singolo frammento
amplificato mediante PCR
Gene grande > numerosi frammenti di
PCR > numerose sequenze
La sequenza viene allineata al
riferimento e analizzata per identificare
eventuali varianti nucleotidiche
SEQUENCE REVOLUTIONS
NGS: sequenziamento massivo parallelo
THROUGHPUT
whole genome (WGS)
NGS
whole transcriptome
Gb
whole exome (WES)
Throughput
Più geni / pazienti
in un singolo
esperimento
Mb
target seq
Kb
50
300
Lenght of Read (bp)
700
Il Laboratorio di Genetica medica nella diagnosi
delle malattie rare
Ieri…
Oggi..
Next generation sequencing (NGS)
Sequenziamento di centinaia di Mb (milioni di bp)
in un’unica seduta analitica
Ricerca: contributo alla identificazione di
nuovi geni responsabili di tratti fenotipici
Diagnosi: incremento delle potenzialità dei
test genetici nella identificazione della causa
molecolare della patologia
SEQUENZIAMENTO NGS
Piattaforme NGS diverse per concezione e chimica ma accomunate dalla capacità di
generare informazioni di sequenza ad alta produttività
(lettura di decine di migliaia di sequenze in parallelo)
PIATTAFORME HIGH THROUGHPUT
CONCEPITE PER SEQUENZIARE AMPIE
REGIONI GENOMICHE
PIATTAFORME MIDDLE THROUGHPUT
CONCEPITE PER SEQUENZIARE
REGIONI GENOMICHE
DI DIMENSIONI MINORI
La tecnologia NGS consente l’esecuzione di indagini in precedenza
tecnicamente non fattibili o economicamente proibitive
Un genoma umano può
essere sequenziato in poche
settimane ad un costo
paragonabile a quello di
alcuni dei test diagnostici
molecolari convenzionali
NGS: potenzialita’ in ambito diagnostico
Diagnosi
molecolare
di
condizioni
precedentemente
caratterizzate
solo
fenotipicamente
Diagnosi molecolare di malattie associate a
mutazioni in geni di difficile approccio mediante
sequenziamento Sanger
Diagnosi molecolare di malattie a ereditarietà
multigenica
Tipologie dei test NGS
Sequenziamento dell’intero genoma
(Whole genome sequencing, WGS)
Sequenziamento dell’intero esoma
(Whole exome sequencing, WES)
Sequenziamento mirato di pannelli di geni
(Target NGS testing o Panel NGS testing)
Il sequenziamento del DNA di nuova generazione: indicazioni per l’impiego clinico: documento SIGU_NGS, gennaio 2016
NGS, workflow di un esperimento
Disegno
esperimento
Preparazione del campione
Sequenziamento
Analisi bioinformatica
Risultati
Validazione
output dell’esperimento
Allineamento delle letture ottenute
con sequenza di riferimento
Identificazione di TUTTE le varianti
identificazione delle
varianti patogenetiche
Varianti di sequenza
Varianti causative
Varianti responsabili di fenotipi non collegati al
quesito clinico (Incidental findings, IF)
Varianti di sequenza con effetti funzionali e clinici
non definiti (variants of uncertain significance, VUS)
CLASSIFICAZIONE DELLE VARIANTI IDENTIFICATE
American College of Medical Geneticists
Disease causing
già descritte come causa della malattia (es.
delezione F508 in CFTR)
Likely disease causing
mai descritte in precedenza, ma si suppone
siano causative in base alla tipologia della
mutazione (es. Una mutazione nonsense in
un gene per cui alter mutazioni di questa
tipologia sono state già descritte
Possibly disease causing
mai descritte in precedenza; in base al tipo
di mutazione potrebbero o non potrebbero
essere causative di malattia
Likely not disease causing
mai descritte in precedenza; probabilmente
non causative di malattia
Not disease causing
già descritte come varianti neutrali
Variant of unknown clinical significance
varianti nè già descritte nè attese come
causative, ma identificate in associazione a
quadri clinici
Next Generation Sequencing nella diagnosi
delle malattie rare
Whole genome
sequencing
Exome
sequencing
Target
sequencing
Fattori determinanti nella scelta dell’approccio
• Costi: il costo del sequenziamento di un esoma può oggi essere minore
del sequenziamento di un pannello di geni
• Finalità: malattie ad elevata eterogeneità genetica/malattie mendeliane
(o sospette tali) con gene ancora non noto
• Sensibilità: è in funzione del coverage delle sequenze indagate (n.
letture “reads” dei singoli tratti del DNA esaminati e del grado di
sovrapposizione tra queste)
• Probabilità di ottenere IF e VUS: dipende dall’ampiezza della porzione
di genoma analizzata e interrogata.
• Archiviazione dei dati: necessità di disporre di piattaforme adeguate
alla archiviazione di un gran volume di dati.
Sequenziamento dell’intero genoma
(Whole genome sequencing, WGS)
L’analisi mediante WGS non viene ancora
considerata come applicabile a livello
diagnostico per le difficoltà
interpretative.
Il sequenziamento del DNA di nuova generazione: indicazioni per l’impiego clinico: documento SIGU_NGS, gennaio 2016
Whole exome sequencing, WES
Sequenziamento delle regioni
codificanti dei geni di un individuo
Indicazioni principali
Patologie genetiche le cui basi molecolari risultino sconosciute
Vantaggi
Analizzare e identificare nuovi geni malattia in pazienti già
sequenziati
Identificare mutazioni in geni malattia noti in fenotipi “atipici”
Limiti
Necessità di un adeguato sistema di archiviazione dati
Clinical Exome Sequencing
Sequenziamento delle regioni codificanti
dei geni noti per essere associati a
malattia (OMIM)
CASO 1
Diagnosi gestaltica
di:
6 anni, caso isolato, genitori non consanguinei
Alla nascita, fistola tracheoesofagea
ad oggi residuano, reflusso
gastro-esofageo ed anemia
Ritardo di acquisizione della parola
Microcefalia (CC <3°centile, P&A
10°centile)
Ipoplasia zigomatica e mandibolare
Dismorfismo auricolare
Disostosi
mandibolofacciale,
tipo Guion-Almeida
CASO 1
Disostosi mandibolofacciale, tipo Guion-Almeida
Disostosi facciale rara
Base molecolare definita nel 2012 (Lines et al., AJHG)
Unico gene: EFTUD2 (no eterogeneità di locus)
EFTUD2
c.[492+4A>G]
Eterozigote, de novo
Nessun laboratorio in Italia
Tre laboratori in UE
(www.orpha.net)
Approccio NGS
TruSight One (Illumina)
(in coll. Dr.ssa Iascone)
Delezioni e mutazioni di splicing
comuni in EFTUD2
CASO 2
Diagnosi gestaltica
& radiologica di:
Nascita/6 mesi, 1° figlio, genitori consanguinei
SGA, importante microcefalia alla nascita
Fronte sfuggente, overlapping delle suture,
micrograzia
Screening ecografico (addome e cuore) nella
norma
RMN cerebrale: grave microcefalia con
semplificazione delle circonvoluzioni
Microcefalia
primaria/sindrome
di Seckel
CASO 2
Microcefalia primaria/sindrome di Seckel
Spettro clinico raro con eterogeneità clinica e di locus
Almeno 16 geni fin ora identificati
Tre di questi sono stati identificati solo in s. di Seckel
ASPM
p.[Gln509ter]
Omozigote
Nessun laboratorio in Italia
Vari laboratori in UE
(www.orpha.net)
Approccio NGS
TruSight One (Illumina)
(in coll. Dr.ssa Iascone)
Targeted sequencing
Indicazioni principali:
patologie dove mutazioni in geni noti sono responsabili della maggior parte dei
casi (es. Teleangectasia Emorragica Ereditaria – mutazioni geni ENG, ALK1 e
SMAD4 nell’80%-87% dei casi)
Vantaggi
Arricchimento specifiche regioni genomiche
Abbattimento dei costi
Analisi di un numero elevato di pazienti contemporaneamente
Limiti
Necessità di un aggiornamento del pannello con l’identificazione di nuovi geni
associati alla malattia di interesse
Studio di 62 geni sarcomerici e
non sarcomerici in 44 paz con
cardiomiopatia ipertrofica
mediante NGS
Definizione di un protocollo
bioinformatico per
l’interpretazione delle varianti
identificate mediante NGS, sia in
ambito di ricerca che in ambito
diagnostico
RISULTATI
FILTERING
(MAF<0,01) +
18.810
varianti
PRIORITIZATION
(Ex ± 10bp)
SANGER
VALIDATION
155 varianti
36/44 (82%) pazienti
portatori di almeno
una variante nonsinonima rara
(frequenza < 1%)
RISULTATI
identificazione di pazienti affetti da cardiomiopatia ipetrofica secondaria
Paziente 10, ♀, 65 anni
MUTAZIONE: GLA c.A644G,p.N215S (eterozigosi)
GLA: malattia di Fabry, X-linked recessiva (1-5/10.000)
Sebbene la malattia di Fabry sia caratterizzata da segni neurologici,
angiocheratoma, insufficienza renale, cardiomiopatia, la pz.10 mostrava solo
cadiomiopatia
Paziente 24, ♀, 7 anni
sindrome di Cantù (<1/1.000.000)
autosomica dominante, ipertricosi congenita, osteocondrodisplasia, cardiomegalia,
cardiomiopatia e dismorfismi
MUTAZIONE: ABCC9 c.C3460T,p.R1154W (eterozigosi)
Paziente 27, ♀, 23 anni
Malattia di Danon (<1/1.000.000)
X-linked recessiva
cardiomiopatia, debolezza muscolare, ritardo mentale
MUTAZIONE: LAMP2 c.453delT,p.F151fs
MALATTIE DEL SISTEMA GENITO-URINARIO
RENE
POLICISTICO
DELL’ADULTO
ADPKD
Sindrome
Branchio-Oto-Renale
(BOR)
MALATTIA
VON HIPPEL LINDAU
RENE
POLICISTICO
RECESSIVO
ARPKD
SINDROME DI
MECKEL
SINDROME
OROFACIODIGITALE
TIPO 1
MALATTIE DEL SISTEMA GENITO-URINARIO
RENE POLICISTICO
DELL’ADULTO
ADPKD
PKD1, PKD2
SINDROME
OROFACIODIGITALE
TIPO 1
SINDROME DI
MECKEL
OFD1
MKS1, MKS2, MKS3
MALATTIA
VON HIPPEL LINDAU
VHL
RENE POLICISTICO
RECESSIVO
ARPKD
PKHD1
Sindrome
Branchio-Oto-Renale
(BOR)
EYA1, SIX5, SIX1
Analisi di geni selezionati
PANNELLI GENICI SEMPLICI
MALATTIE DEL SISTEMA GENITO-URINARIO
SINDROME DI
ALPORT
SINDROME DI
MECKEL
SINDROME DI
BARDET-BIEDL
SINDROME DI
BARTTER
SINDROME DI
JOUBERT
Congenital Anomalies of the
Kidney and the Urinary Tract
(CAKUT)
MALATTIE MULTIGENICHE AD EREDITARIETA’ COMPLESSA
SINDROME DI
JOUBERT
SINDROME DI
ALPORT
COL4A3, COL4A4, COL4A5, MYH9
AHI1, ARL13B, B9D1, B9D2, C5orf42, CC2D2A, CEP290, CEP41,
KIF7, MKS1, NPHP1, NPHP3, OFD1, RPGRIP1L, TCTN1, TCTN2,
TMEM138, TMEM216, TMEM237, TMEM67, TTC21B
SINDROME DI
BARDET-BIEDL
ALMS1, ARL6, BBS1, BBS10, BBS12, BBS2,
BBS4, BBS5, BBS7, BBS9, CCDC28B, CEP290,
LZTFL1, MKKS, MKS1, NPHP1, SDCCAG8,
TRIM32, TTC8, WDPCP
SINDROME DI
BARTTER
ATP6V1B1, BSND, CA2, CASR,
CLCNKA, CLCNKB, CLDN16, CLDN19,
FXYD2, HSD11B2, KCNJ1, KCNJ10,
KLHL3, NR3C2, SCNN1A, SCNN1B,
SCNN1G, SLC12A1, SLC12A3,
SLC4A1, SLC4A4, WNK1, WNK4
Congenital Anomalies of the
Kidney and the Urinary Tract
(CAKUT)
ALDH1A2, BICC1, BMP4, CHD1L, EYA1, FGF20, FOXC1, FRAS1,
FREM1, FREM2, GATA2, GATA3, GDNF, GRIP1, HNF1B, NPHP3,
OSR1, PAX2, RET, ROBO2, SDCCAG8, SIX1, SIX5, SOX17,
TFAP2A, UPK3A, WT1
Analisi di geni selezionati
PANNELLI GENICI complessi
SINDROME DI BARDET BIEDL
Patologia geneticamente eterogenea (21 geni), trasmessa per lo più come carattere AR; in
alcuni casi è stata osservata una modalità di trasmissione oligogenica
BBS1
23%
Heon et al, 2016
Mutazioni a diversi loci BBS
possono interagire e
determinare o modulare
il fenotipo di alcuni individui
80% dei casi
Mockel et al, 2011
NGS: REGOLAMENTAZIONE
Il documento tratta l’ampio range di aspetti etici, legali,
sociali e pratici che scaturiscono dall’uso della
tecnologia NGS in ambito diagnostico-clinico
25 raccomandazioni per implementare l’uso dei test
dagnostici mediante NGS massimizzandone i benefici
e minimizzando i potenziali rischi dovuti ad un loro
impiego non corretto
Definizione criteri standardizzati per la scelta dei geni da inserire nei pannelli
diagnostici, curati, validati e aggiornati da un team multidisciplinare di esperti
(clinici e laboratoristi)
•
Raccomandazioni operative per i
test NGS
•
Consulenza genetica nei test NGS
(Peculiarità e Contenuti specifici)
•
Comunicazione dei risultati
(consulenza post-test)
•
Analisi dei dati NGS e requisiti di
qualità
•
Conservazione dei dati NGS
Problematiche rilevanti:
Incidental findings (IF)
Identificazione di varianti a carico di geni responsabili
di patologie non correlate al quadro clinico per cui si
sta eseguendo l’accertamento.
Riscontro che abbia potenziale importanza per la
salute o rappresenti un rischio riproduttivo
Consenso informato
Consulenza genetica
Il Laboratorio di Genetica medica nella diagnosi delle
malattie rare
Diagnosi prenatale non invasiva
Diagnosi preimpianto
NIPT - Non Invasive Prenatal Testing
cffDNA: DNA fetale libero circolante
Origina da cellule placentari e viene
rilasciato nel sangue materno
cffDNA è presente nel plasma materno a partire dalla IV-V settimana di gestazione
e la sua concentrazione (mediante compresa tra il 2% e il 40%) aumenta
proporzionalmente con l’età gestazionale.
Entro circa 2 ore dal parto non è più presente nel plasma materno
FRAZIONE FETALE (FF): % di cffDNA nel plasma materno.
Per poter effettuare NIPT la FF deve essere almeno il 4%
NIPT - Non Invasive Prenatal Testing
Il DNA libero circolante viene sequenziato e il numero di sequenze
per ogni cromosoma viene confrontato con un genoma di
riferimento.
Se il numero di sequenze di un cromosoma si discosta dall’atteso,
viene segnalata un’aneuploidia
Anomalie cromosomiche indagate: trisomia 13, 18, 21 + aneuploidie
dei cromosomi sessuali
Riduzione attesa del ricorso a procedure di diagnosi prenatale
invasiva: 95%
NIPT - Non Invasive Prenatal Testing
FATTORI CHE INFLUENZANO AFFIDABILITA’ DEL TEST
-Mosaicismi confinati alla placenta
-Vanishing twin
-Peso della madre (influenza Frazione Fetale)
-Fumo, alcol, droghe da parte della madre (influenzano
Frazione Fetale)
-Infezioni materne
-Neoplasie materne
-Trasfusioni di sangue, trattamento con cellule
staminali, trapianti materni
LA NIPT è un test di screening (non diagnostico)
Il Laboratorio di Genetica medica nella diagnosi delle
malattie rare
Diagnosi prenatale non invasiva
Diagnosi preimpianto
CHI RICHIEDE PGD
Chi richiede la PGD
COPPIE CON RISCHIO AUMENTATO PER UNA PATOLOGIA EREDITARIA GRAVE
• Coppie a rischio infertili
• Coppie fertili con rischio 25-50%
• Coppie a rischio con una storia di ripetuti aborti per
feto affetto e/o che rifiutano aborto
Nuovi strumenti diagnostici nel
Laboratorio di Genetica medica
Conclusioni
Implementazione delle capacità diagnostiche
dei test genetici
Standardizzazione dei percorsi di validazione
dei test genetici
Centralizzazione dei test genetici di II livello
(DCA 549 del 18-11-2015)
Nuovi strumenti diagnostici nel
Laboratorio di Genetica medica
Conclusioni
Adeguamento delle dotazioni organiche
dei Laboratori di Genetica medica
Implementazione degli ambulatori
di consulenza genetica
Forte interazione tra genetisti di laboratorio
e genetisti clinici
Nuovo nomenclatore/tariffario