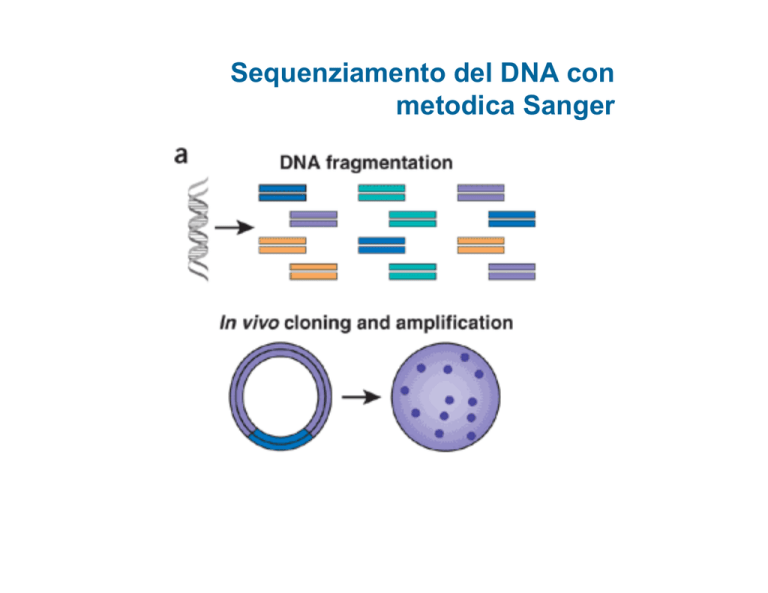
Sequenziamento del DNA con
metodica Sanger
1a generazione
sequenziamento del DNA secondo il metodo
Sanger
Il sequenziamento automatico secondo il metodo Sanger
ha dominato nella scienza e nell’industria per almeno 20
anni e ha consentito il sequenziamento del genoma
umano e la scoperta di oltre 2.500 malattie genetiche
monogeniche
Il metodo Sanger è considerato la tecnologia di “prima
generazione”, mentre i nuovi metodi sono denominati
“next-generation sequencing (NGS)”
Sanger DNA sequencing
2a generazione
“next-generation sequencing (NGS)”
Il principale vantaggio è la possibilità tecnica di produrre un
volume enorme di dati a costi estremamente più bassi
ed in tempi estremamente più rapidi
Il potenziale dell’NGS è simile ai primi tempi della PCR con
il limite principale dovuto all’immaginazione
NGS
Preparazione del DNA da sequenziare
Una comune strategia tecnologie NGS è che il template
è adeso o immobilizzato su una superficie solida o
supporto
L'immobilizzazione di molecole spazialmente separate
permette di eseguite simultaneamente migliaia o milioni
di reazioni di sequenziamento
Come si prepara il DNA?
Tecniche basate sull’amplificazione:
1.
emulsion PCR (emPCR) (454, Life)
2.
solid-phase amplification (Solexa)
Tecniche non basate sull’amplificazione:
1.
immobilizzazione dei primer (Helicos)
2.
immobilizzazione del “template” (Helicos)
3.
immobilizzazione dell’enzima (Pacific, LI-COR)
emulsion PCR (emPCR)
emulsion PCR (emPCR)
Il 5500xl SOLiD legge
200.000.000.000 di basi/corsa
Solid-phase amplification can produce 100–200 million spatially separated
template clusters (Illumina/Solexa)
I gruppi bloccanti
attaccati al 3’
causano un ostacolo
nell’incorporazione
di un altro
nucleotide, ma
l’ostacolo può
essere rimosso
L’Illumina HiSeq2000 legge
oltre 600.000.000.000 di basi
di DNA per corsa
A doubling of sequencing output every 9 months has outpaced and overtaken
performance improvements within the disk storage and high-performance
computation fields
S D Kahn Science 2011;331:728-729
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
Whole Genome Seq
Exome Seq
RNA Seq
MeDIP Seq
miRNA Seq
Epigenetics, DNA methylation, ChIP-Seq
Copy Number Variation
Gut Bacteria / Metagenome
Skin Bacteria / Metagenome
Infectious Diseases
NGS Applications in Cancer
Biomarkers
Stem Cells
Detection of Genome Structural Variation
Personalised Medicine Applications
The term NGS defines very different sizes of
analysis!
• Whole genome @40 x (~ 140 Gb of
sequences/sample) √
• Whole exome @60 x (~ 5 Gb/sample) √
√
• A set of 100 aver. candidate genes
(exons) @100 x (~ 0.1 Gb/sample) √
√√
• A single medium-size gene (exons and
introns) @100x (~ 0.005 Gb/sample) √
The genome of
James Watson
7.4 x coverage
234 runs
24.5 billions bp
11 genetic diseases !!
Whole Genome Seq
very heterogeneous genotype, genetic testing for 15/39 loci
Pedigree of the family and segregation of SH3TC2 mutations
Arricchimento “enrichment”
• Tutti i geni (esoni+introni) di una regione cromosomica
dove è mappata una malattia genetica da causa ignota
• Tutti gli esoni di tutti i geni coinvolti in varie forme di
quella malattia genetica
• Tutti gli esoni di tutti i geni noti del genoma umano
(esoma umano)
Meno dell’1% delle mutazioni che causano
malattie genetiche cadono fuori dagli esoni
The RNA is transcribed from
PCR-amplified
oligodeoxynucleotides
originally synthesized on a
microarray, generating
sufficient bait for multiple
captures at concentrations
high enough to drive the
hybridization
minigenome preparation
Come si fa a comprendere quale variazione nella
sequenza del DNA possa avere un significato
patologico?






![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)





